一般に ゲノムクラスターは同じゲノム位置に存在し、同じ経路に関与したり(Barona-Gómez et al、2004; Yu et al、2000)、同じ機能を持っていたり(Hourcade et al、1992)、共発現したり(Sémonand Duret、2006)、結合または細胞局在化(Jamieson et al、2004)する遺伝子グループを意味している。この定義は、一群の遺伝子に適合するだけでなく、分類的および位置的に記載され得る任意の特徴を持ったグループ群にも当てはまり得る。シークエンシング技術のコストが減少するにつれて、小規模な実験室でも新しいゲノムが生成され、特徴的なクラスターの同定は、貴重な分析データとして標準的なアノテーションプロセスとして採用されうる。ゲノムのクラスターを同定できる種々のツールが存在する(Cimermancic et al、2014; Cruz-Morales et al、2016; Khaldi et al、2010; Li et al、2009; Medema et al、2011; Röttig et al、2011; Starcevic et al、2008; Umemura et al、2013; Vesth et al、2016; Wolf et al、2016; Yi et al、2007)。それらは一般に特定のタイプのクラスタを識別し、検索するクラスタの特定の構成を考慮に入れたアルゴリズムを使用するように特化されている。さらに、それらの多くはバクテリアゲノムのために開発されている(Chavali and Rhee、2017)。これらの制限を克服するために、クラスタを識別する一般的なツールであるClusterScanを開発し、機能やカテゴリの選択に柔軟性を持たせた。このツールは、ゲノム上の近接したゲノムフィーチャのクラスターを同定し、同じカテゴリーに関連づける。位置情報およびカテゴリ情報はユーザーが検索できるようにする表形式で定義され、yーザーがPFAMドメイン、GOカテゴリ、SNP、保存領域、bindiingサイト、転移因子などの任意の種類のクラスターを検索できるようにしている。
ClusterScanはPythonで開発されており、位置解析にはbedtools(Quinlan and Hall、2010)、プロットにはRを使用している。 入力には、解析するフィーチャのゲノム座標を有するタブ区切りのテキストファイルと、そのフィーチャに1つ以上のカテゴリに従って注釈を付けるファイルが必要になる。 クラスタを特定するために、ツールは各カテゴリについて、同じアノテーションとポジションを共有するグループを検索する。 クラスタのポジションと構成、および必要に応じて各クラスタのバイスタンダーのリスト(存在する場合)とシングルトンのリストを格納する複数の出力ファイルを生成する。 クラスタ内のフィーチャの最小数とそれらの間の最大距離はユーザが設定可能であり、探索戦略は2つの異なるアルゴリズム(論文 図1a)の中から選択できる。
ClusterScanのパイプライン。論文より転載。
https://github.com/pyrevo/ClusterScan/wiki/Welcome-to-the-ClusterScan-wiki!
ClusterScanに関するツイート。
インストール
mac os10.12でdockerを使ってテストした。
本体 Github
ここではdokcerイメージを使い導入する。
#pull
docker pull sangeslab/clusterscan:latest
#データフォルダーを共有して立ち上げる。オリジナルはユーザーで立ち上げているが、ここではrootで立ち上げる
docker run -it -v /full/path/to/data/folder:/data sangeslab/clusterscan bash
> clusterscan.py --version
clusterscan.py --version
Options:
-h, --help Show this screen.
-o, --output PATH Specify output path [default: ./].
-a, --analysis NAME Specify optional analysis name for output files.
-n, --nf=<n> Minimum number of features per cluster [default: 2].
-d, --dist=<bp> Maximum distance between features in bp [default: 500000].
-w, --window=<bp> Window size [default: 500000].
-s, --slide=<bp> Sliding size [default: 250000].
-k, --seed=<n> Number of standard deviations to identify a window which serves as the beginning of the cluster [default: 3].
-e, --extension=<n> Number of standard deviations to identify the window(s) which serve to extend the cluster [default: 2].
-c, --category LIST Comma separated list of one or more specific categories to be analyzed [e.g. PF00001,PF00002].
Useful when you need to perform the analysis only for specific categories in the ANNOTATION file.
--info FILE Specify optional file to describe categories.
--singletons Identify singletons after clusters and bystanders annotation.
--version Show program version.
ラン
チュートリアルデータを解析する。
https://github.com/pyrevo/ClusterScan/wiki/ClusterScan-Tutorial
使うのはヒトのFeatureアノテーションファイル。
- Homo_sapiens.GRCh38.85_genes.bed: EnsemblのGRCh38のGFF3からコード領域だけ抽出したファイル。
- Homo_sapiens.GRCh38.85_Pfam.txt: BiomartからGene typeでフィルタリングして得たファイル。
- Pfam-descriptors.txt: Pfamのアノテーションファイル。
Pfam、Biomartについては統合TVに簡単な説明があります(Pfam、Biomart)。
cd /appdata/ClusterScan/tutorial/
clusterscan.py clusterdist Homo_sapiens.GRCh38.85_genes.bed Homo_sapiens.GRCh38.85_Pfam.txt --info Pfam-descriptors.txt -o human_test -a human_01
- -o Specify output path [default: ./].
- -a Specify optional analysis name for output
- --info Specify optional file to describe
total number of unique features scanned: 19919
Total number of unique categories scanned: 6056
Total number of clusters found: 2287
--- 266 seconds ---
ジョブが終了するとhuman_test/に結果が出力される。結果を/dataにコピーする。
cp human_test/* /data
macに戻る(ログイン状態も抜けたいなら"control"を押した状態で"P、Q")。
共有ディレクトリを確認。


- human_01_clusters.bed : human_01_clusters.tsvのbedバージョン。
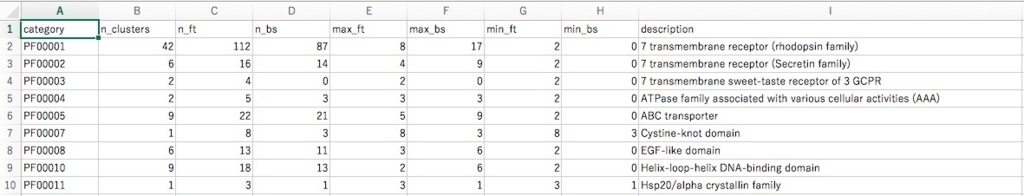
- human_01_features.tsv: クラスタと重複していると判明したフィーチャのリスト。

詳細はチュートリアルを読んでください。
ClusterScan Tutorial · pyrevo/ClusterScan Wiki · GitHub
引用
ClusterScan: simple and generalistic identification of genomic clusters
Massimiliano Volpe, Marco Miralto Stefano Gustincich Remo Sanges
Bioinformatics, bty486, 15 June 2018
