植物に有益な微生物は、持続可能な植物生産や植物衛生上の実践において重要性を増している。しかし、植物成長促進(PGP)に関連する細菌形質を対象とした計算論的アプローチが不足しており、植物刺激性細菌株のin-silico同定、比較、選択を妨げている。この問題に対処するため、本著者らは新しいウェブリソースPLaBAse(v1.01, http://plabase.informatik.uni-tuebingen.de/pb/plabase.php)を開発した。このリソースでは、(i)5,565の植物関連細菌をスクリーニングするためのデータベース(PLaBA-db)、(ii)単一細菌ゲノムの植物成長促進形質(PGPT)を予測するためのツール(PGPT-Pred)、(iii)マーカー遺伝子同定による植物関連細菌予測ツール(PIFAR-Pred)など、多くのサービスを提供している。後者はMartínez-Garcíaらによって開発され、現在Tuebingen大学でホストされている。PGPT-Predツールは本著者らの新しいPGPTオントロジーに基づいており、これは文献とOMICsによってキュレートされた、6,965,955のタンパク質配列に関連する約6,900のPGPTの包括的かつ階層的なコレクションである。異なる環境におけるPGPTの分布を調べるため、(i)7つの異なる環境(植物を含む)、(iii)5つの異なる植物球(器官)、および(iii)細菌が誘導する2つの植物表現型に関連する70,540の細菌株に適用した。この解析から、植物共生細菌は一般的にゲノムサイズが大きく、PGPTにアノテーションされたタンパク質コード遺伝子の数が多いことが明らかになった。もちろん、報告されているすべてのPGPTが植物関連細菌や植物共生細菌に限定されているわけではなく、また植物共生細菌だけに濃縮されているわけでもない。PGPTの中には、ヒトや動物に共生する細菌にも存在するものがある。おそらく、PGPBが環境間で伝播するためか、あるいはいくつかの機能が様々な環境への適応過程に関与しているためであろう。ここでは、PLaBA-dbを使用して、様々な系統や分離部位にわたる細菌ゲノム中のPGPTをスクリーニングし、PGPT-Predを使用して標準化されたアノテーションを行うための使いやすいアプローチを提供する。このリソースは、PGPプロセス全体に関する理解を深め、バイオ接種剤としてのPGPBの予測やバイオセーフティ戦略を容易にし、将来的に持続可能で標的を絞った細菌を組み込んだ植物生産システムの確立に役立つと確信している。
manual
https://plabase.cs.uni-tuebingen.de/pb/manual.php
https://plabase.cs.uni-tuebingen.de/pb/plabase.php にアクセスする。
GET STARTEDをクリック。
5つ利用できる。

PlaBA-db
約5,565の植物性アシスト菌、その植物分離部位、植物相互作用表現型、PGPT(plant growth-promoting traits)アノテーションなどをスクリーニング。
PIFAR-Pred
植物細菌の相互作用因子(タンパク質)"PIFAR "のアノテーション。PIFARタンパク質のコレクションと分類に対してblastp+hmermerを使用し、植物関連細菌の決定につながる。8,000タンパク質に限定してアップロード。
PGPT-Pred
PGPTオントロジーに対してblast(relaxed mode)、blastp+hmmer(strict)またはIMG-KEGG-annoation mappingを用いた、細菌の植物成長促進形質(タンパク質)「PGPT」のアノテーション。アップロードは8,000タンパク質に制限。
CPTP-db
タンパク質の相同性に基づき、公開データセットからテールシンをコードする可能性のある細菌とその標的細菌をスクリーニングする。アクセス制限あり。
CPTP-Pred
タンパク質相同性、シンテニー、HMM-Modelsに基づく、テールシンシンテニック遺伝子クラスター候補とその推定ターゲット細菌のアノテーション。アクセス制限あり。
PlaBA-dbを見てみる。
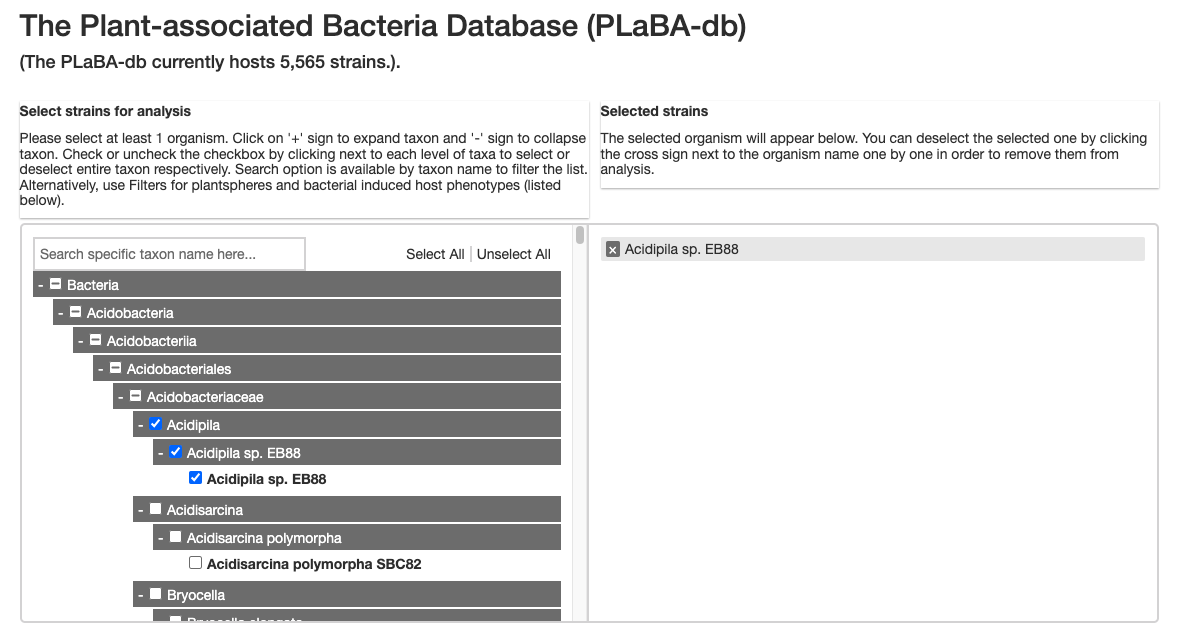
ウィンドウ内では1つの階層的分類しか見えていないが、画面はずっと下までスクロールできる。チェックボックスを1つ以上選択する。
キーワードで選択することもできる。

選択された菌の情報を入手するにはsubmitをクリックする。
出力

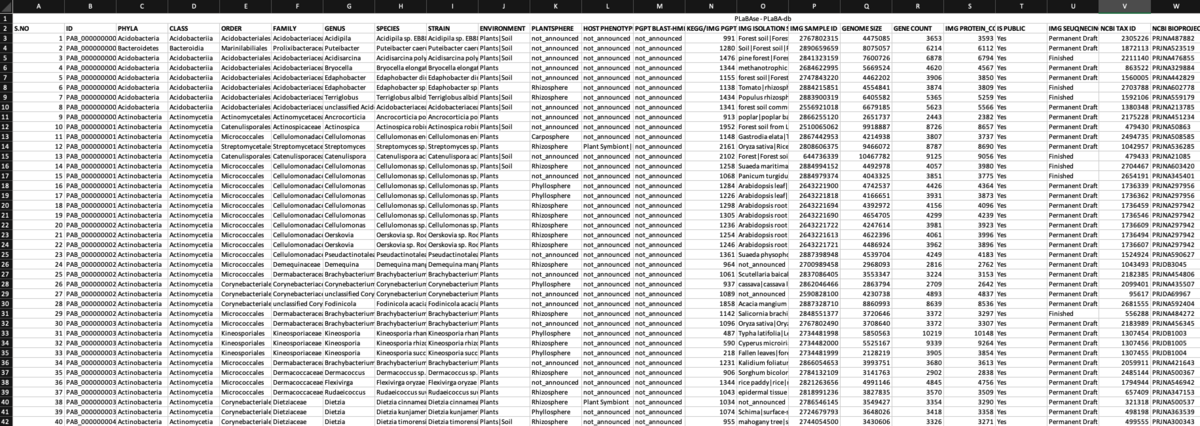
結果はexcel形式などでダウンロードできる。現在29列からなり、full taxonomy、株名、サンプリング場所、ゲノムアクセッション、ゲノムサイズ、遺伝子数、タンパク質数などの基本的な情報の他、PGPT BLAST-HMM COUNTなどの情報を含んでいる。
続いてPIFAR-Pred

植物と相互作用している可能性がある細菌の相互作用因子(タンパク質)"PIFAR "のアノテーションを探索するサービスとなっている。
PIFARタンパク質のコレクションと分類DBに対して、クエリとなるタンパク質を指定して探索する。タンパク質を指定する。上限8000タンパク質。

blastp+hmermerを使って検索される。8,000タンパク質に限定してアップロード。緑色のフロッピーマークをクリックするとexample fastaファイルをダウンロードできる。
ジョブ待ちの時はキューに追加され、ジョブ終了時にはメールで通知される。
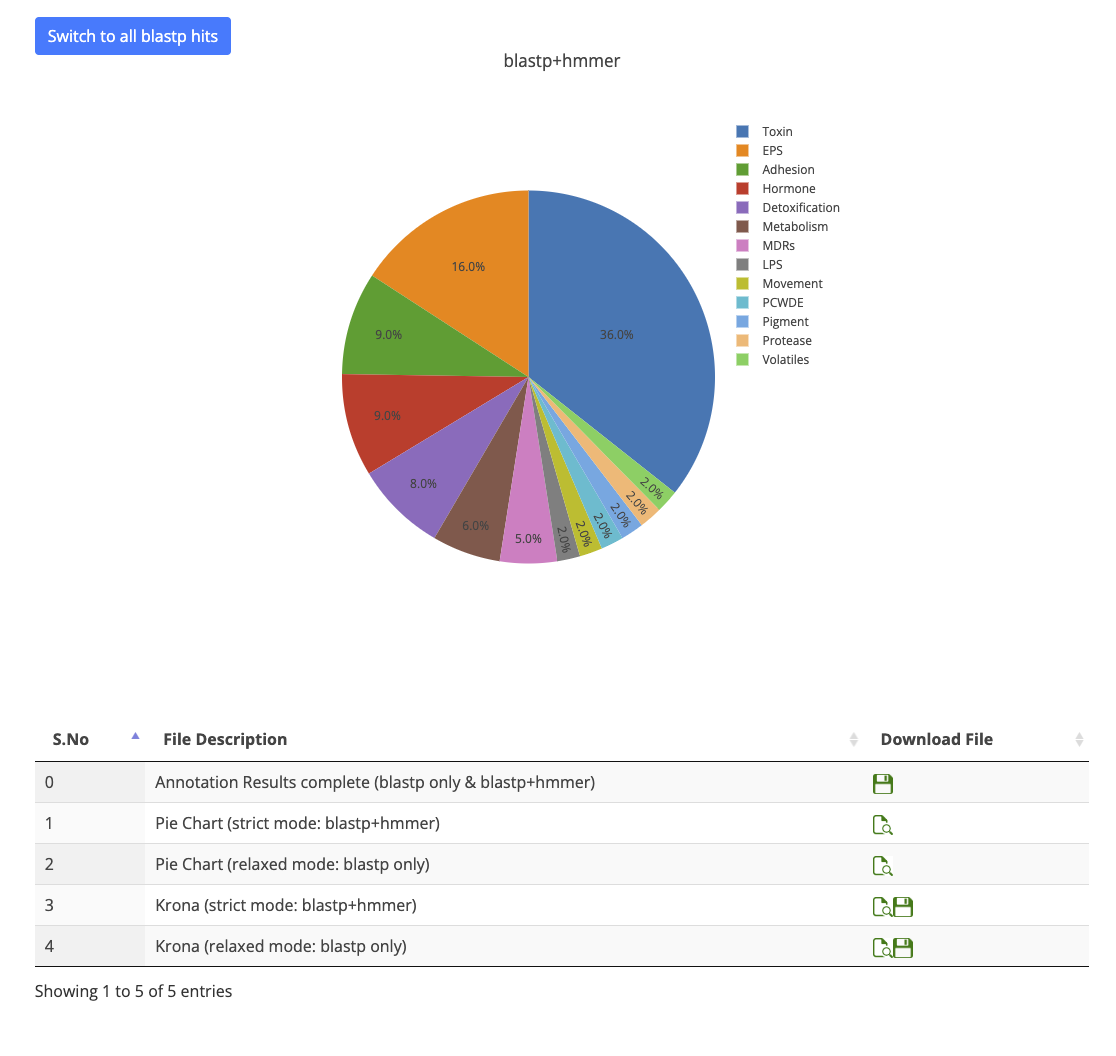
全ての結果はダウンロードできる。kronaで可視化するためのテキストも含まれている。デモデータを使ってジョブを投げたときは終了の通知が来るまでに20時間ほどかかった。
PGPT-Pred
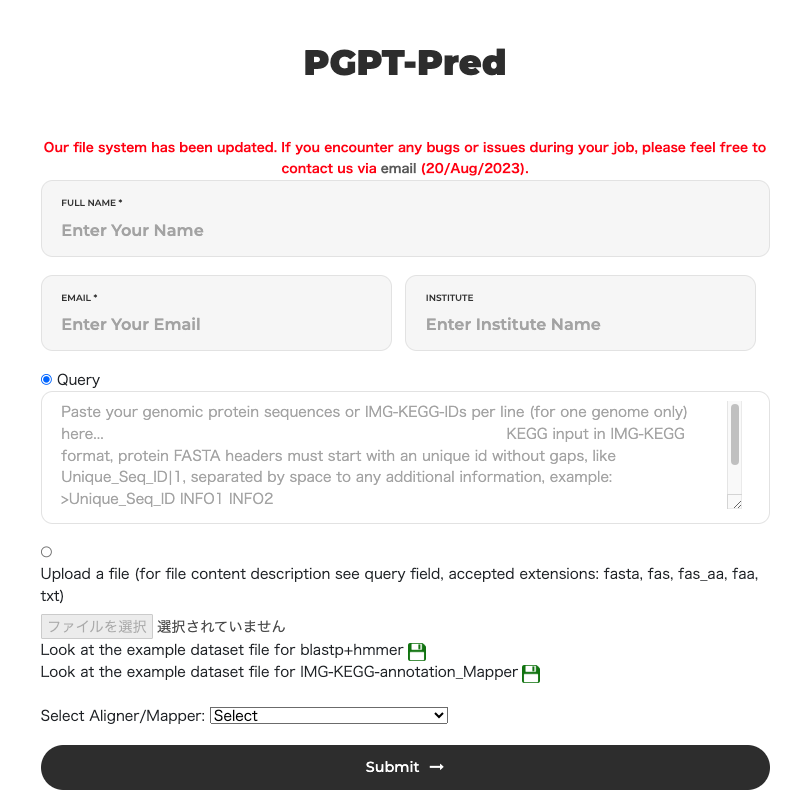
PGPTオントロジーに対してblast(relaxed mode)、blastp+hmmer(strict)またはIMG-KEGG-annoation mappingを用いて、細菌の植物成長促進形質(タンパク質)「PGPT」のアノテーションを探索する。上限8,000タンパク質。
引用
PLaBAse: A comprehensive web resource for analyzing the plant growth-promoting potential of plant-associated bacteria
S. Patz, A. Gautam, M. Becker, S. Ruppel, P. Rodríguez-Palenzuela, DH. Huson
bioRxiv, Posted December 15, 2021
