2020 2/12 タイトル修正、2/20 コメント追加、2/25 インストール手順修正、10/5 コマンド微修正、10/9 インストール微修正
2024/05/06 python3.7 => 3.8に修正
このモジュールはいくつかの代替方法のうちの1つに従って平均ヌクレオチド同一性ANIを計算する。ANIは、DNA-DNAハイブリダイゼーション(DDH )の適切なin silico代替物であると提案されており、したがって種の境界を描写するのに有用となる。 文献中の種の境界に対する典型的な閾値は95%ANIになっている(例えばRichter et al、2009)。
インストール
ubuntu18.04 LTSでcondaの仮想環境を作ってテストした。
依存
Python3
For ANI analysis
- Biopython http://www.biopython.org
- NumPy http://www.numpy.org/
- pandas http://pandas.pydata.org/
- SciPy http://www.scipy.org/
Alignment tools
- BLAST+ executable in the $PATH, or available on the command line (required for ANIb analysis) ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
- legacy BLAST executable in the $PATH or available on the command line (required for ANIblastall analysis) ftp://ftp.ncbi.nlm.nih.gov/blast/executables/release/LATEST/
- MUMmer executables in the $PATH, or available on the command line (required for ANIm analysis) http://mummer.sourceforge.net/
For graphical output
- matplotlib http://matplotlib.org/
- seaborn https://github.com/mwaskom/seaborn
本体 Github
#anacondaを使っているならcondaで導入可能(mac,linux)
mamba create -n pyani python=3.8 -y
conda activate pyani
mamba install -c bioconda -y pyani
#pipでも導入可能(本体とらいぶりのみ。サードのツールはインストールされない)
pip3 install pyani
> average_nucleotide_identity.py -h
$ average_nucleotide_identity.py -h
usage: average_nucleotide_identity.py [-h] [--version] -o OUTDIRNAME -i
INDIRNAME [-v] [-f] [-s FRAGSIZE]
[-l LOGFILE] [--skip_nucmer]
[--skip_blastn] [--noclobber]
[--nocompress] [-g] [--gformat GFORMAT]
[--gmethod {mpl,seaborn}]
[--labels LABELS] [--classes CLASSES]
[-m {ANIm,ANIb,ANIblastall,TETRA}]
[--scheduler {multiprocessing,SGE}]
[--workers WORKERS]
[--SGEgroupsize SGEGROUPSIZE]
[--SGEargs SGEARGS] [--maxmatch]
[--nucmer_exe NUCMER_EXE]
[--filter_exe FILTER_EXE]
[--blastn_exe BLASTN_EXE]
[--makeblastdb_exe MAKEBLASTDB_EXE]
[--blastall_exe BLASTALL_EXE]
[--formatdb_exe FORMATDB_EXE]
[--write_excel] [--rerender]
[--subsample SUBSAMPLE] [--seed SEED]
[--jobprefix JOBPREFIX]
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
-o OUTDIRNAME, --outdir OUTDIRNAME
Output directory (required)
-i INDIRNAME, --indir INDIRNAME
Input directory name (required)
-v, --verbose Give verbose output
-f, --force Force file overwriting
-s FRAGSIZE, --fragsize FRAGSIZE
Sequence fragment size for ANIb (default 1020)
-l LOGFILE, --logfile LOGFILE
Logfile location
--skip_nucmer Skip NUCmer runs, for testing (e.g. if output already
present)
--skip_blastn Skip BLASTN runs, for testing (e.g. if output already
present)
--noclobber Don't nuke existing files
--nocompress Don't compress/delete the comparison output
-g, --graphics Generate heatmap of ANI
--gformat GFORMAT Graphics output format(s) [pdf|png|jpg|svg] (default
pdf,png,eps meaning three file formats)
--gmethod {mpl,seaborn}
Graphics output method (default mpl)
--labels LABELS Path to file containing sequence labels
--classes CLASSES Path to file containing sequence classes
-m {ANIm,ANIb,ANIblastall,TETRA}, --method {ANIm,ANIb,ANIblastall,TETRA}
ANI method (default ANIm)
--scheduler {multiprocessing,SGE}
Job scheduler (default multiprocessing, i.e. locally)
--workers WORKERS Number of worker processes for multiprocessing
(default zero, meaning use all available cores)
--SGEgroupsize SGEGROUPSIZE
Number of jobs to place in an SGE array group (default
10000)
--SGEargs SGEARGS Additional arguments for qsub
--maxmatch Override MUMmer to allow all NUCmer matches
--nucmer_exe NUCMER_EXE
Path to NUCmer executable
--filter_exe FILTER_EXE
Path to delta-filter executable
--blastn_exe BLASTN_EXE
Path to BLASTN+ executable
--makeblastdb_exe MAKEBLASTDB_EXE
Path to BLAST+ makeblastdb executable
--blastall_exe BLASTALL_EXE
Path to BLASTALL executable
--formatdb_exe FORMATDB_EXE
Path to BLAST formatdb executable
--write_excel Write Excel format output tables
--rerender Rerender graphics output without recalculation
--subsample SUBSAMPLE
Subsample a percentage [0-1] or specific number (1-n)
of input sequences
--seed SEED Set random seed for reproducible subsampling.
--jobprefix JOBPREFIX
Prefix for SGE jobs (default ANI).
> genbank_get_genomes_by_taxon.py -h
$ genbank_get_genomes_by_taxon.py -h
usage: genbank_get_genomes_by_taxon.py [-h] -o OUTDIRNAME [-t TAXON] [-v] [-f]
[--noclobber] [-l LOGFILE]
[--format FORMAT] --email EMAIL
[--retries RETRIES]
[--batchsize BATCHSIZE]
[--timeout TIMEOUT]
optional arguments:
-h, --help show this help message and exit
-o OUTDIRNAME, --outdir OUTDIRNAME
Output directory (required)
-t TAXON, --taxon TAXON
NCBI taxonomy ID
-v, --verbose Give verbose output
-f, --force Force file overwriting
--noclobber Don't nuke existing files
-l LOGFILE, --logfile LOGFILE
Logfile location
--format FORMAT Output file format [gbk|fasta]
--email EMAIL Email associated with NCBI queries (required)
--retries RETRIES Number of Entrez retry attempts per request.
--batchsize BATCHSIZE
Entrez record return batch size
--timeout TIMEOUT Timeout for URL connection (s)
テストラン
git clone https://github.com/widdowquinn/pyani.git
cd pyani/
使うのはtest/の4ゲノム(fastaファイル)。
genome.fastaのディレクトリを指定して実行する。gzip等で圧縮しているとエラーが出るので注意。tests/test_ani_data/を使う。
cd tests/
#mummer
average_nucleotide_identity.py -m ANIm -g -i test_ani_data/ -o ANIm_out
#blastn (mummerはANIが80%より低いと頭打ちになる)
average_nucleotide_identity.py -m ANIb -g -i test_ani_data/ -o ANIb_out
- -m {ANIm, ANIb, ANIblastall, TETRA} ANI method (default ANIm)
- -g Generate heatmap of ANI
- -i Input directory name (required)
- -o Output directory (required)
ANImはmummer、ANIbはblast+、ANIblastallはlegacy blastが使われる。
- ANIm: uses MUMmer (NUCmer) to align the input sequences.
- ANIb: uses BLASTN+ to align 1020nt fragments of the input sequences
- ANIblastall: uses legacy BLASTN to align 1020nt fragments of the input sequences
- TETRA: calculates tetranucleotide frequencies of each input sequence
画像出力
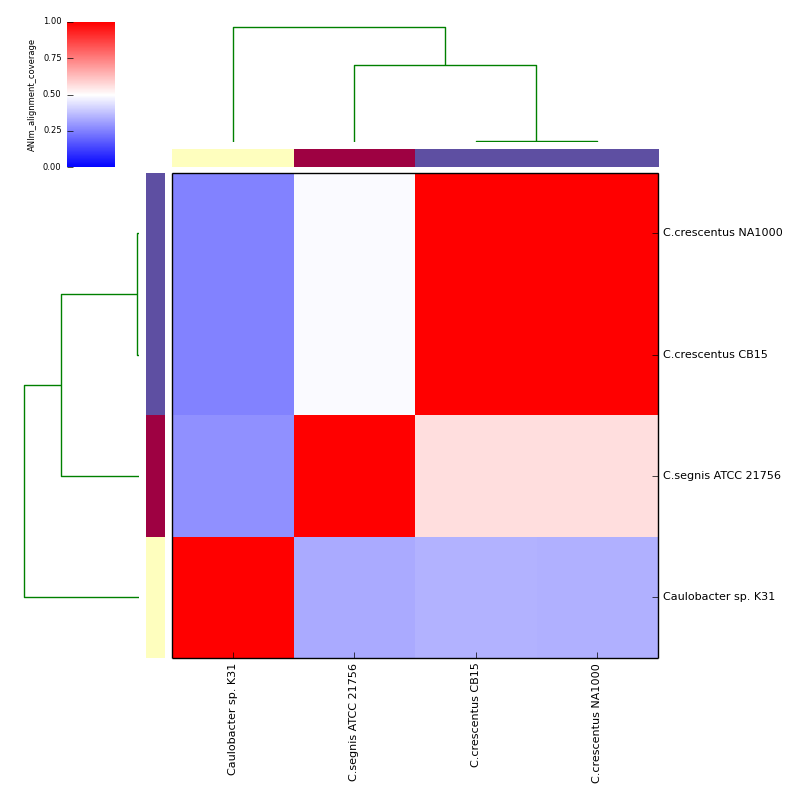


付属スクリプト"genbank_get_genomes_by_taxon.py"を使ってRefseqからゲノムをダウンロードし、それらゲノムを使ってANI比較を実行する。taxIDはNCBI taxonomy browser(リンク)で調べる。
#例えば1703751をダウンロード
genbank_get_genomes_by_taxon.py -t 1703751 -o OUTDIRNAME --email my-mail-adress
#OUTDIRNAMEにそれぞれのゲノムの~ .fna.gzがダウンロード、解凍される。
#pyaniを実行
average_nucleotide_identity.py -i OUTDIRNAME/ -o ANIblast -m ANIb -g
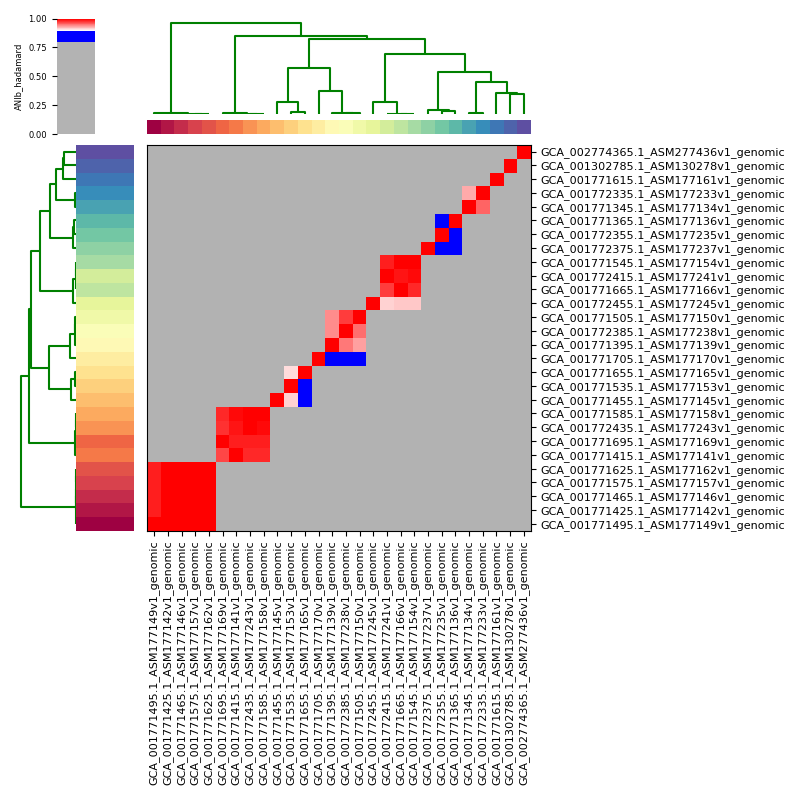
出力(ANI下限値以下は灰色をアサイン)
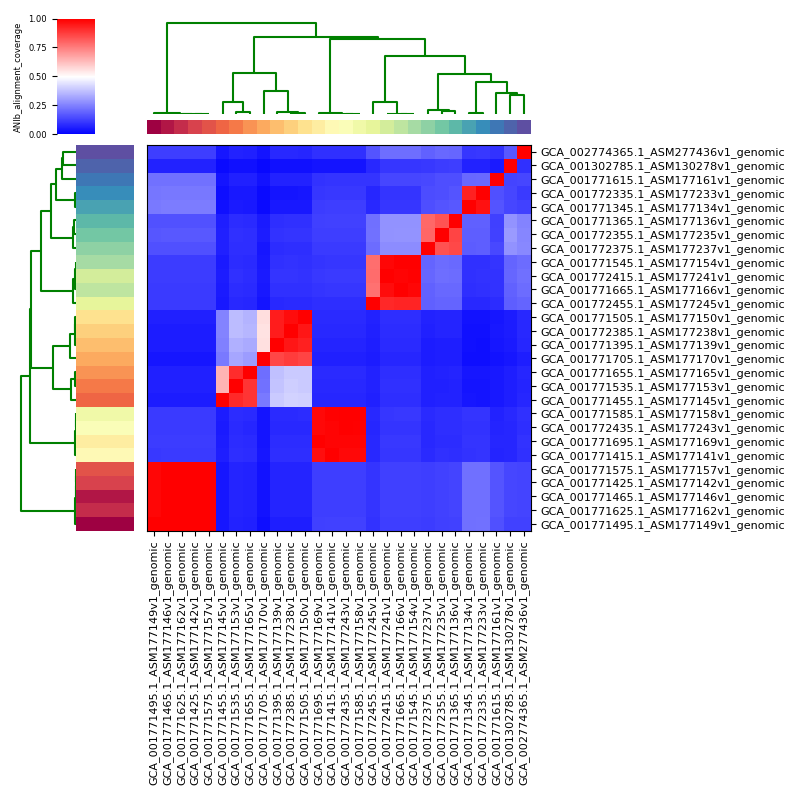
出力(下限なし)
参考
アライナーによって異なる(LASTのように固定または可変の)ワード長を使用していること、また、ギャップ作成とギャップ拡張のペナルティとアラインメントのドロップオフスコアが異なることで、感度が異なる。例えば、blastnは11の固定ワード長を有し、Mummerは20のワード長を有し、一方、megablast(ヌクレオチドBLASTオプションの1つ)は28のワード長を有し、Mummerはblastnよりも感度が低く、megablastはMummerよりもさらに感度が低い。これは一般的に、より長いワード長を使用するアライナーはゲノム間のほぼ同一性の一致のみを見つけ、一方、BLASTnはより低い%の同一性を有するより発散性の高い配列を見つけることを意味する。その結果、種内ANIの特定のカットオフは、ANIの計算に使用する特定のアライナー(特定のBLASTオプション(BLASTn vs megablast vs discontiguous megablast)を含む)によって若干変動する。
https://mgm.jgi.doe.gov/img-webinar-ani-average-nucleotide-identity/
引用
Genomics and taxonomy in diagnostics for food security: soft-rotting enterobacterial plant pathogens
Leighton Pritchard, Rachel H. Glover, Sonia Humphris, John G. Elphinstone, Ian K. Toth
Analytical Methods, 8(1), 12-24, 2016
Make Heatmap/dendogram figure just from ANI matrix table?
https://github.com/widdowquinn/pyani/issues/85
関連
ANI近似
2024/04/28追記
v0.3でfastANIも使えるようだが、まだβらしい(fastANI.py)。最新のリリースはv0.2.12