近年、技術が絶えず改良され科学者や開業医を含む広範囲の顧客にとって利用しやすいためにDNAシーケンシングブームが起きている。メタゲノミクスから植物生理学、医学まで、多くの分野の研究者が、彼らの研究にシーケンシング実験を実施してきた。 Oxford Nanopore Technologies(ONT)は、技術的スキルとバイオインフォマティクスに関する知識を最低限しか必要としない携帯型マシンであるMinIONシーケンサーを導入することで、このプロセスを大幅にスピードアップした。したがって、DNAシーケンシング実験は、野外研究でも、小さな実験室でも、そして診療所での医学的応用のためにも実行可能になってきている[ref.1]。
NCBI PubMedデータベースには、2009年から2018年の間(2018年9月1日まで)にpublishされた「Oxford Nanopore」という語句を含む261の科学論文が含まれている(その大部分は過去3年間にpublishされた)。これは、ONTシーケンサーの人気の高まりと、過去3年間におけるテクノロジとシーケンス品質の大幅な向上の両方を指している。例えば、MiniIONフローセルのR10バージョンが最近発表され、ホモポリマーストレッチについてもシーケンス品質の向上を約束している。ハイスループットおよびロングリードは、MinIONの多様な応用を可能にする。ウイルス学[ref.2 link、3 pubmed]、植物病理学および農業[ref.4、5]、結核研究[ref.6]、メタゲノミクスおよび食べ物[ref.7 link]、ならびに獣医学研究[ref.8 pubmed]。ポータブルプラットフォームとしては、野外生物多様性研究[ref.9]、現場での患者におけるエボラウイルスの検出[ref.10、11]、および宇宙でのシーケンシング[ref.12, pubmed]。 de novoゲノムアセンブリ、既存のゲノムアセンブリの改良、構造変異および長いリピート配列の発見など、ロングリードシーケンス研究の基本的な用途も忘れない[ref.13–16]。小型のバクテリアゲノムは1回のMinIONリードでカバーできる[ref.17, 18]、高解像度なゲノムアーキテクチャを提供できるため、ONTシーケンスは微生物学研究に適している。
ONTはユーザーにMinIONの電気信号を一連のヌクレオチドに変換するbase callingを行うために必要なソフトウェアを提供している。それはMinNNOWとオフラインのAlbacoreである。どちらのアプリケーションも複雑で再帰的なニューラルネットワークアルゴリズムを利用している。これは最近コンピュータ科学で非常に人気がある。それはソフトウェアが既存のデータから学び、そのパフォーマンスを改善することを可能にする。base callingプロセスがONTシーケンステクノロジの精度を向上させるための中心であり、そのアルゴリズムが絶えず改善および更新されていることは注目に値する。出力は、FAST5および/またはFASTQファイルのコレクションになる。これらは、バイオインフォマティクスのシーケンス解析に使用されるファイルである。したがって、base calling元は、MinIONからデータ解釈への「ゲート」と呼ぶことができる。それにもかかわらず、ONTが提供する分析ツールの範囲は限られており、ユーザーに任されている一般的な処理を除いて、特定のアプリケーションだけに関係している。例えば、MetrichorプラットフォームをベースとしたEPI2MEソフトウェアスイートには、バーコード分析、メタゲノミクス、抗菌剤耐性分析、およびいくつかの技術的試験のためのアプリケーションが含まれている[ref.19]。最近、いくつかの研究グループがMinION特有のバイオインフォマティクスツールの開発に焦点を当てている[ref.20-22]。これらのほとんどはかなりのバイオインフォマティクス知識を必要とする。これらの条件は、MinIONベースのDNAシーケンスが、情報技術(IT)の経験が少ない医師や研究者にもたらすことができる利益を妨げる。このギャップを埋めるために、MinIONによって生成されたデータを迅速かつ容易に処理し、必要に応じてさらなるバイオインフォマティクス分析に必要なファイルを提供することができるWeb駆動の自動パイプラインでNanoPipeを開発した。
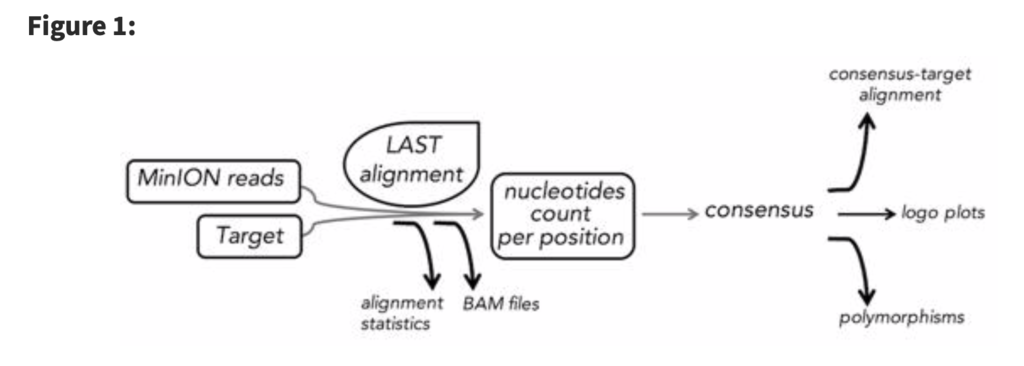
A schematic representation of the NanoPipe workflow. 論文より転載。
usage
Institute of Bioinformatics WWU Münster
使い方
http://bioinformatics.uni-muenster.de/tools/nanopipe2/generate/index.pl?lang=en
にアクセスする。
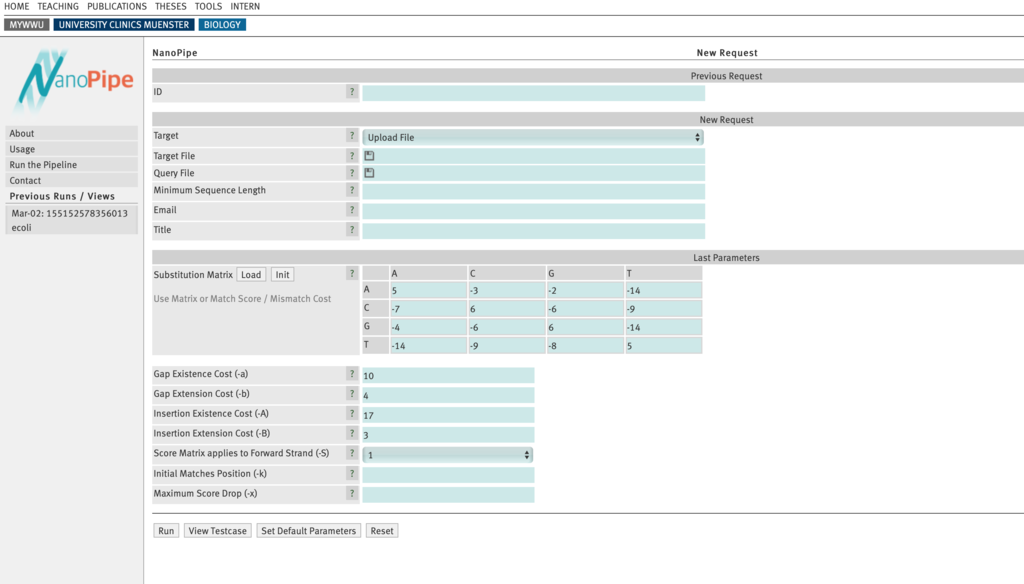
Target Fileにリファレンスゲノム、Quey Fileにシーケンスデータを選択する。複数シーケンスデータがある場合は圧縮して送る(zip archive.zip file1 file2 ...)。ファイルサイズは最大3GB。

いくつかのモデル生物のリファレンス配列はすでにサーバーに準備されている。Upload Fileをクリックして一覧から選択する。
あとはマッピングに使われる LASTのパラメータを選択してランする。テスト時は数時間で結果が得られた。
デモラン
View Testcaseをクリックすると、デモランの結果を表示できる。

Overview
マッピング率、LASTのパラメータなどが表示される。

Mapping distribution
どのchrmosomeにどれだけマッピングされたか確認できる。

Alignment distribution
リードのアライメントされた長さの分布がまとめられる。

BAM FIles
ダウンロードして使う。bamとbam.bai、リファレンスfastaをダウンロードできる。

Target ID
Target IDのタブで表示するリファレンスを選ぶ(これより下は1つのリファレンスのみ分析対象になるため)

Nucleotide Plots
各ポジションの塩基が表示される。下のボタンで上流/下流に移動する。

Consensus
コンセンサス配列が表示される。コンセンサス配列はDownloadボタンからダウンロードできる。

コンセンサス配列は、そのポジションでもっと割合が多い塩基がアサインされる(割合が80%以上になること)。ナノポアはエラー率が高いので、カバレッジが10以下は一律ギャップ(-)になる。
Polymorphisms
推定SNPが表示される。

以下の3つの条件で残ったものにウエイトがつけてまとめられる。
- The coverage of the target nucleotide is lower than 80 % of the total coverage at that position.
- For non-target nucleotides at that position: The coverage must be greater than 20% of the total coverage.
- A position must have a coverage of more than 30% of the general target's maximum coverage. E.g.: The maximum read coverage of a target position is 5000, thus, SNPs with a coverage below 1500 are discarded.
ヒトゲノムとPlasmodium falciparum(熱帯熱マラリア原虫)は、dbSNP / PlasmoDBのデータベースとマッチするかも表示される。
Alignments
リファレンスとコンセンサス配列のpairwiseアライメント結果が表示される。

引用
NanoPipe—a web server for nanopore MinION sequencing data analysis
Victoria Shabardina, Tabea Kischka, Felix Manske, Norbert Grundmann, Martin C Frith, Yutaka Suzuki, Wojciech Makałowski
GigaScience, Volume 8, Issue 2, 1 February 2019
関連





 image2.
image2.








 Pan genomeを 選択。Pan genomeモードでは親ゲノムというものはないので、そのままContinueをクリック。
Pan genomeを 選択。Pan genomeモードでは親ゲノムというものはないので、そのままContinueをクリック。





