2020/07/23 モニターコマンド追記
2021/01/8 helpのバージョン更新
2021/08/22 更新
2022/1/7 v6に更新(helpはv4)
2022/02/16 helpをv6に更新
タイトルの通り、GuppyのGPU版を使うまでの流れをまとめておきます。
ubuntuへのインストール
#レポジトリの追加
sudo add-apt-repository ppa:graphics-drivers/ppa
sudo apt update
#NVIDIA driverのインストール。最新GPUだとより最新のNvidiaドライバーを入れる必要があるかもしれない(ONTのGuppy documentより)。
sudo apt install nvidia-384
#OS reboot
sudo reboot
libcuda.so.1がないというエラーが出たら、/libcuda.soから/libcuda.so.1にシンボリックリンクを張って、$LD_LIBRARY_PATHに追加することでとりあえず解決。
#私の環境では
ln -s /usr/local/cuda/lib64/stubs/libcuda.so /usr/local/cuda/lib64/stubs/libcuda.so.1
export LD_LIBRARY_PATH=/usr/local/cuda/lib64/stubs:${LD_LIBRARY_PATH}
バージョン確認
> modinfo nvidia | grep version
2、GuppyのGPU版ダウンロード
2020 1/19現在、Guppyのv3.4.4が提供されている。log inしてsoftware downloadからlinux GPUビルドをダウンロードする(=> 2023 5/28現在v6.5.7が最新)。
https://community.nanoporetech.com/downloads

注;少し前からGuppyはをダウンロードしなくてもパッケージマネージャでインストールできるようになっています。画像中央列の各プラットフォーム向けマニュアルを確認して下さい。
cd ont-guppy/bin/
> ./guppy_basecaller #v.6.01
$ guppy_basecaller
: Guppy Basecalling Software, (C) Oxford Nanopore Technologies, Limited. Version 6.0.1+652ffd179
Usage:
With config file:"
guppy_basecaller -i <input path> -s <save path> -c <config file> [options]
With flowcell and kit name:
guppy_basecaller -i <input path> -s <save path> --flowcell <flowcell name>
--kit <kit name>
List supported flowcells and kits:
guppy_basecaller --print_workflows
Use GPU for basecalling:
guppy_basecaller -i <input path> -s <save path> -c <config file>
--device <cuda device name> [options]
Command line parameters:
--trim_threshold arg Threshold above which data will be
trimmed (in standard deviations of
current level distribution).
--trim_min_events arg Adapter trimmer minimum stride
intervals after stall that must be
seen.
--max_search_len arg Maximum number of samples to search
through for the stall
--override_scaling Manually provide scaling parameters
rather than estimating them from each
read.
--scaling_med arg Median current value to use for manual
scaling.
--scaling_mad arg Median absolute deviation to use for
manual scaling.
--trim_strategy arg Trimming strategy to apply: 'dna' or
'rna' (or 'none' to disable trimming)
--dmean_win_size arg Window size for coarse stall event
detection
--dmean_threshold arg Threshold for coarse stall event
detection
--jump_threshold arg Threshold level for rna stall detection
--pt_scaling Enable polyT/adapter max detection for
read scaling.
--pt_median_offset arg Set polyT median offset for setting
read scaling median (default 2.5)
--adapter_pt_range_scale arg Set polyT/adapter range scale for
setting read scaling median absolute
deviation (default 5.2)
--pt_required_adapter_drop arg Set minimum required current drop from
adapter max to polyT detection.
(default 30.0)
--pt_minimum_read_start_index arg Set minimum index for read start sample
required to attempt polyT scaling.
(default 30)
--as_model_file arg Path to JSON model file for adapter
scaling.
--as_gpu_runners_per_device arg Number of runners per GPU device for
adapter scaling.
--as_cpu_threads_per_scaler arg Number of CPU worker threads per
adapter scaler
--as_reads_per_runner arg Maximum reads per runner for adapter
scaling.
--as_num_scalers arg Number of parallel scalers for adapter
scaling.
--noisiest_section_scaling_max_size arg
Threshold read size in samples under
which nosiest-section scaling will be
performed.
-m [ --model_file ] arg Path to JSON model file.
-k [ --kernel_path ] arg Path to GPU kernel files location (only
needed if builtin_scripts is false).
-x [ --device ] arg Specify basecalling device: 'auto', or
'cuda:<device_id>'.
--builtin_scripts arg Whether to use GPU kernels that were
included at compile-time.
--chunk_size arg Stride intervals per chunk.
--chunks_per_runner arg Maximum chunks per runner.
--chunks_per_caller arg Soft limit on number of chunks in each
caller's queue. New reads will not be
queued while this is exceeded.
--high_priority_threshold arg Number of high priority chunks to
process for each medium priority chunk.
--medium_priority_threshold arg Number of medium priority chunks to
process for each low priority chunk.
--overlap arg Overlap between chunks (in stride
intervals).
--gpu_runners_per_device arg Number of runners per GPU device.
--cpu_threads_per_caller arg Number of CPU worker threads per
basecaller.
--num_callers arg Number of parallel basecallers to
create.
--post_out Return full posterior matrix in output
fast5 file and/or called read message
from server.
--stay_penalty arg Scaling factor to apply to stay
probability calculation during
transducer decode.
--qscore_offset arg Qscore calibration offset.
--qscore_scale arg Qscore calibration scale factor.
--temp_weight arg Temperature adjustment for weight
matrix in softmax layer of RNN.
--temp_bias arg Temperature adjustment for bias vector
in softmax layer of RNN.
--beam_cut arg Beam score cutoff for beam search
decoding.
--beam_width arg Beam score cutoff for beam search
decoding.
--duplex_window_size arg Window size to use for prefix search in
duplex decoding.
--disable_qscore_filtering Disable filtering of reads into
PASS/FAIL folders based on min qscore.
--min_qscore arg Minimum acceptable qscore for a read to
be filtered into the PASS folder
--reverse_sequence arg Reverse the called sequence (for RNA
sequencing).
--u_substitution arg Substitute 'U' for 'T' in the called
sequence (for RNA sequencing).
--log_speed_frequency arg How often to print out basecalling
speed.
--barcode_kits arg Space separated list of barcoding
kit(s) or expansion kit(s) to detect
against. Must be in double quotes.
--trim_barcodes Trim the barcodes from the sequences in
the output files.
--trim_adapters Trim the adapters from the sequences in
the output files.
--trim_primers Trim the primers from the sequences in
the output files.
--num_extra_bases_trim arg How vigorous to be in trimming the
barcode. Default is 0 i.e. the length
of the detected barcode. A positive
integer means extra bases will be
trimmed, a negative number is how many
fewer bases (less vigorous) will be
trimmed.
--score_matrix_filename arg File containing mismatch score matrix.
--start_gap1 arg Gap penalty for aligning before the
reference.
--end_gap1 arg Gap penalty for aligning after the
reference.
--open_gap1 arg Penalty for opening a new gap in the
reference.
--extend_gap1 arg Penalty for extending a gap in the
reference.
--start_gap2 arg Gap penalty for aligning before the
query.
--end_gap2 arg Gap penalty for aligning after the
query.
--open_gap2 arg Penalty for opening a new gap in the
query.
--extend_gap2 arg Penalty for extending a gap in the
query.
--min_score_barcode_front arg Minimum score to consider a front
barcode to be a valid barcode
alignment.
--min_score_barcode_rear arg Minimum score to consider a rear
barcode to be a valid alignment (and
min_score_front will then be used for
the front only when this is set).
--min_score_barcode_mask arg Minimum score for a barcode context to
be considered a valid alignment.
--min_score_adapter_mid arg Minimum score for a mid-strand adapter
to be considered a valid alignment.
--min_score_adapter arg Minimum score for an adapter to be
considered a valid alignment.
--min_score_primer arg Minimum score for a primer to be
considered to be a valid alignment.
--front_window_size arg Window size for the beginning barcode.
--rear_window_size arg Window size for the ending barcode.
--require_barcodes_both_ends Reads will only be classified if there
is a barcode above the min_score at
both ends of the read.
--allow_inferior_barcodes Reads will still be classified even if
both the barcodes at the front and rear
(if applicable) were not the best
scoring barcodes above the min_score.
--detect_barcodes Detect barcode sequences at the front
and rear of the read.
--detect_adapter Detect adapter sequences at the front
and rear of the read.
--detect_primer Detect primer sequences at the front
and rear of the read.
--detect_mid_strand_adapter Detect adapter sequences within reads.
--detect_mid_strand_barcodes Search for barcodes through the entire
length of the read.
--min_score_barcode_mid arg Minimum score for a barcode to be
detected in the middle of a read.
--lamp_kit arg LAMP barcoding kit to perform LAMP
detection against.
--min_score_lamp arg Minimum score for a LAMP barcode to be
classified.
--min_score_lamp_mask arg Minimum score for a LAMP barcode mask
context to be classified.
--min_score_lamp_target arg Minimum score for a LAMP target to be
classified.
--min_length_lamp_target arg Minimum align length for a LAMP target
to be classified.
--min_length_lamp_context arg Minimum align length for a LAMP barcode
mask context to be classified.
--additional_lamp_context_bases arg Number of bases from a lamp FIP barcode
context to append to the front and rear
of the FIP barcode before performing
matching. Default is 2.
--num_barcoding_buffers arg Number of GPU memory buffers to
allocate to perform barcoding into.
Controls level of parallelism on GPU
for barcoding.
--num_mid_barcoding_buffers arg Number of GPU memory buffers to
allocate to perform barcoding into.
Controls level of parallelism on GPU
for mid barcoding.
--num_barcode_threads arg Number of worker threads to use for
barcoding.
--read_splitting_arrangement_files arg
Files containing arrangements for read
splitting.
--read_splitting_score_matrix_filename arg
File containing mismatch score matrix
for read splitting.
--num_read_splitting_buffers arg Number of GPU memory buffers to
allocate to perform read splitting.
Controls level of parallelism on GPU
for read splitting using mid adapter
detection.
--num_read_splitting_threads arg Number of worker threads to use for
read splitting.
--min_score_read_splitting arg Minimum alignment score for the mid
adapter on which to split the read.
--do_read_splitting Perform read splitting based on
mid-strand adapter detection.
--max_read_split_depth arg The maximum number of iterations of
read splitting that should be
performed.
--num_reads_per_barcoding_buffer arg The maximum number of reads to process
at once in each barcoding buffer.
--calib_detect Enable calibration strand detection and
filtering.
--calib_reference arg Reference FASTA file containing
calibration strand.
--calib_min_sequence_length arg Minimum sequence length for reads to be
considered candidate calibration
strands.
--calib_max_sequence_length arg Maximum sequence length for reads to be
considered candidate calibration
strands.
--calib_min_coverage arg Minimum reference coverage to pass
calibration strand detection.
--print_workflows Output available workflows.
--flowcell arg Flowcell to find a configuration for
--kit arg Kit to find a configuration for
-a [ --align_ref ] arg Path to alignment reference.
--bed_file arg Path to .bed file containing areas of
interest in reference genome.
--align_type arg Specify whether you wand full or coarse
alignment. Valid values are
(auto/full/coarse).
--num_alignment_threads arg Number of worker threads to use for
alignment.
-z [ --quiet ] Quiet mode. Nothing will be output to
STDOUT if this option is set.
--trace_categories_logs arg Enable trace logs - list of strings
with the desired names.
--verbose_logs Enable verbose logs.
--trace_domains_config arg Configuration file containing list of
trace domains to include in verbose
logging (if enabled)
--disable_pings Disable the transmission of telemetry
pings.
--ping_url arg URL to send pings to
--ping_segment_duration arg Duration in minutes of each ping
segment.
--progress_stats_frequency arg Frequency in seconds in which to report
progress statistics, if supplied will
replace the default progress display.
-q [ --records_per_fastq ] arg Maximum number of records per fastq
file, 0 means use a single file (per
worker, per run id).
--read_batch_size arg Maximum batch size, in reads, for
grouping input files.
--compress_fastq Compress fastq output files with gzip.
-i [ --input_path ] arg Path to input fast5 files.
--input_file_list arg Optional file containing list of input
fast5 files to process from the
input_path.
-s [ --save_path ] arg Path to save fastq files.
-l [ --read_id_list ] arg File containing list of read ids to
filter to
-r [ --recursive ] Search for input files recursively.
--fast5_out Choice of whether to do fast5 output.
--bam_out Choice of whether to do BAM file
output.
--index Choice of whether to output BAM index
file.
--bam_methylation_threshold arg The value below which a predicted
methylation probability will not be
emitted into a BAM file, expressed as a
percentage. Default is 5.0(%).
--resume Resume a previous basecall run using
the same output folder.
--client_id arg Optional unique identifier
(non-negative integer) for this
instance of the Guppy Client
Basecaller, if supplied will form part
of the output filenames.
--nested_output_folder If flagged output fastq files will be
written to a nested folder structure,
based on: protocol_group/sample/protoco
l/qscore_pass_fail/barcode_arrangement/
--max_queued_reads arg Maximum number of reads to be submitted
for processing at any one time.
-h [ --help ] produce help message
-v [ --version ] print version number
-c [ --config ] arg Config file to use
-d [ --data_path ] arg Path to use for loading any data files
the application requires.
GPUを確認しておく。
> nvidia-smi
$ nvidia-smi
Sun Jan 19 12:06:32 2020
+-----------------------------------------------------------------------------+
| NVIDIA-SMI 390.129 Driver Version: 390.129 |
|-------------------------------+----------------------+----------------------+
| GPU Name Persistence-M| Bus-Id Disp.A | Volatile Uncorr. ECC |
| Fan Temp Perf Pwr:Usage/Cap| Memory-Usage | GPU-Util Compute M. |
|===============================+======================+======================|
| 0 GeForce GTX 1080 Off | 00000000:07:00.0 On | N/A |
| 19% 52C P8 9W / 200W | 334MiB / 8118MiB | 0% Default |
+-------------------------------+----------------------+----------------------+
+-----------------------------------------------------------------------------+
| Processes: GPU Memory |
| GPU PID Type Process name Usage |
|=============================================================================|
| 0 1256 G /usr/lib/xorg/Xorg 162MiB |
| 0 1501 G /usr/bin/gnome-shell 100MiB |
| 0 3217 G ...uest-channel-token=12657443395681243138 68MiB |
+-----------------------------------------------------------------------------+
GTX 1080の8 GB VRAM(GDDR5X)になっている。
実行方法
CPU版と同様の流れでランできる。異なるのはCPUスレッドの代わりにデバイス番号を指定するところ。1台しか利用できないなら--device autoか--device cuda:0を指定すればO.K。
guppy_basecaller \
--flowcell FLO-MIN106 \
--kit SQK-LSK109 \
-x cuda:0 \
-i fast5_dir \
-s output_dir2 -r
- -x [ --device ] Specify basecalling device: 'auto', or 'cuda:<device_id>'.
- --flowcell Flowcell to find a configuration for
- -kit Kit to find a configuration for
- -i [ --input_path ] Path to input fast5 files.
- -s [ --save_path ] Path to save fastq files.
100MB程度の小さなfast5データを使ってランタイムを調べた。
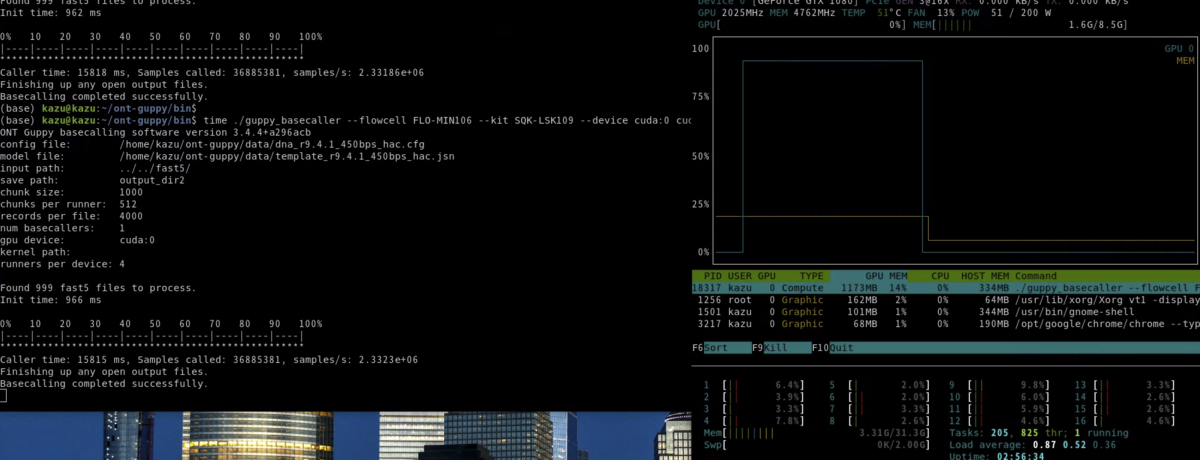
ラン中はGPU使用率がほぼ100%になる(右上)(nvtopを使用 *1)。
結果は
GTX 1080 =>18.5s
CPU(AMD 3700x) => 7m56.6s
25倍の差がついた。大きなデータでは、GPU版を使わないと終わらないのがよく分かりました。
参考
*1
nvtopのインストール
git clone https://github.com/Syllo/nvtop.git
mkdir -p nvtop/build && cd nvtop/build
cmake .. -DNVML_RETRIEVE_HEADER_ONLINE=True
make
sudo make install
#help
nvtop -h
nvtopは複数GPUもモニターできます。上では、Terminator(参考にしたHP)を入れて端末を分割しています。
nvtop
#追記 nvtopが導入できない環境ならnvidia-smiを使う。GPU1をモニター。1秒おきに更新(-l 1)。
nvidia-smi -i 1 -l 1



















