2019 7/28 help追記、タイトル修正、コマンド例追記
2019 10/20 リンク追加
2020 1/11 インストール修正
現代のDNAシーケンシング技術は染色体の全配列を読み取ることができない。代わりに、それらはゲノムの異なる部分からサンプリングされた多数のリードを生成する。低コストで高品質の第2世代シーケンシング(次世代シークエンシングまたはNGSとも呼ばれる)の出現により、科学者は以前にシーケンシングれていない数多くの生物を解読することができるようになった。しかしながら、ゲノムシーケンシングの市場は急速に発展しており、NGS技術の支配力も、第3世代(long-read sequencing(LRS)の絶え間ない改善のために、現在疑問視されている。 Pacific BiosciencesとOxford Nanopore TechnologiesのLRS技術によって作成されたリードの長さは、NGSによって生成された長さより数桁高く10万塩基を超えることができる。同時に、これらの技術は、NGS競合他社と比較してエラー率が大幅に高いリードを生成し、かなり高価なままである。従って、両方の種類のDNAシーケンシング技術は重大な欠点があるが、しかし、計算方法で克服することができる。
ゲノムアセンブリソフトウェアは、リードをコンティグと呼ばれるより大きな配列に組み合わせる。ゲノムの長いリピートは、完全な染色体の再構成を妨げる。完全な染色体の再構成は、NGS技術によって生成されたショートリードのみを使用するとほとんどの場合不可能である。しかし、ショートリードアセンブリは長距離のメイトペアライブラリー(Chaisson et al、2009; Vasilinetc et al、2015)を用いて有意に改善することができる。典型的なインサートサイズが1キロベース(kb)未満のペアエンドライブラリとは対照的に、現在のメイトペアプロトコルは、より長いインサート(最大20kb)を生成する。アセンブリソフトウェアは、その情報を利用して、長いリピートとカバレッジギャップをまたぎ、コンティグをscaffoldsと呼ばれるより長いシーケンスに拡張する。ロングリードのアセンブリアルゴリズムは、リード長が複雑なリピートを解決するのに十分なので、通常、ペアは必要ない。 LRS技術の最も重要な欠点は、高いベース・エラー率であり、より高いシーケンスデプス(Koren et al。、2017)に向け投資するか、高品質のショートリードを使用してエラーを訂正する(Walker et al、2014)。
ゲノムアセンブリの課題に取り組むため、様々なアセンブリツールが異なるヒューリスティックアプローチを使用しており、その結果、生じるコンティグおよびScaffoldsのに大きな違いが生じる。それは、アセンブリを互いに比較するという問題を引き起こす。 Assemblathon1(Earl et al、2011)は、主要ゲノムアセンブリソフトウェアを評価する最初の試みの1つだった。主催者は、未知のシミュレートされたゲノムを使いシミュレーションのショートリードデータセットを提供した。このデータセットは、世界中のアセンブラ開発者とユーザーの17グループによってアセンブリされ、結果として得られた41のアセンブリが慎重に評価された。その後の研究であるAssemblathon 2(Bradnam et al、2013)は同様の競合モデルを使用したが、3つの脊椎動物種からの実際のシーケンシングデータを提供した。残念なことに、両方のアセンブリプロジェクトで使用された評価スクリプトは、ほとんどがそれらの特定のゲノムに集中しており、ルーティンの品質評価に簡単には適用できなかった。 GAGEの研究(Salzberg et al、2012)は、ゲノムアセンブリ評価のゴールデンスタンダードを確立し、それを実装するプログラムを作成した。 GAGEツールは、その後のバクテリアゲノムアセンブラ(Magoc et al、2013)のGAGE-B競争の主な評価ツールとなったQUAST(Gurevich et al。、2013)ソフトウェアでさらに拡張され改良された。メタゲノムの解釈の研究(Sczyrba et al、2017)の最近の批判的評価は、メタゲノム評価のためQUASTの拡張であるMetaQUAST(Mikheenko et al、2016b)を用いた。
QUASTパッケージは幅広く使用されているが、普遍的なゲノムアセンブリ評価ツールとして扱うことはほとんどない。その理由は、QUAST設計の元々の目的がバクテリアや小型の真核生物アセンブリの評価であり、100メガよりも小さいゲノムに制限されているからである。この研究では、ラージゲノムの配列を解析できる大幅にアップグレードされたQUASTのバージョンであるQUAST-LGを提示する。著者らのツールは、通常のサーバー上で数時間以内に哺乳動物サイズのアセンブリを評価することができる。 QUAST-LGには、トランスポゾンの豊富さなど、真核生物ゲノムの特定の特徴を考慮に入れたスピードアップの改善と新しい品質メトリックの両方が含まれている。 REAPR(Hunt et al、2013)(紹介)、ALE(Clark et al、2013)およびLAP(Ghodsi et al、2013)のような純粋なリファレンスフリーアプローチとは対照的に、元のQUASTは既知のリファレンスゲノムに依存していた。 QUAST-LGは、パイプラインの一部にリファレンスフリーのツールを組み込むことで、新規種の評価を可能にする。しかし、リファレンスフリー解析はQUAST-LGの主要なユースケースではない。
QUAST-LGの別の改良点は、ゲノムの長いリピートおよび低いカバレッジの領域のために、リファレンスゲノムが未処理のリードから完全に再構築され得ないという事実に基づくアセンブリ上限の概念である。 near-optimalな アセンブリ問題(Bresler et al、2013; Lam et al、2014)に関する以前の研究で、いくつかの理論的仮定の下で、ゲノム再構成を成功させるのに必要なショットガンシーケンシングデータのリード長とカバレッジ深さが推定されている。著者らは反対の問題にアプローチする:データセットが与えられた場合、QUAST-LGは、この特定のリードセットからアセンブリソフトウェアによって理論的に到達可能な完全性および連続性の上限を推定する。さらに、本方法は、データセットがNGSとLRSの両方の技術によって生成された複数のシークエンシングライブラリで構成されていることを考慮している。
QUAST-LGの機能性を実証するために、著者らはミディアムサイズおよびラージサイズのゲノムの最先端のゲノムアセンブリツールの能力を評価する。2つのライブラリ、すなわちイルミナペアエンドリードとハイクオリティなイルミナメイトペア、またはイルミナペアエンドリードとロングリードライブラリーのいずれかを含むWGSデータセットを使用する。パシフィックバイオサイエンスの1分子リアルタイム(PacBio SMRT)シークエンシングとOxford Nanoporeシークエンシングの両方の主要LRS技術は、これらのテストデータセットの中に存在する。結果の簡便な再現を可能にするために、この研究で使用されたすべてのデータセットおよびソフトウェアツールは自由に利用可能となっている。
QUAST-LG HP(論文内のアセンブリツール比較に使われたデータと、QUAST-LG resultsのリンクあり(S. cerevisiae、human、Fruit fly、C. elegans))
QUAST-LG – Center for Algorithmic Biotechnology
マニュアル
http://quast.bioinf.spbau.ru/manual.html
プレゼンテーションPDF
http://cab.spbu.ru/files/quast/quast-lg/talk_and_poster/Gurevich_QUAST-LG_ISMB2018.pdf

Poster
http://cab.spbu.ru/files/quast/quast-lg/talk_and_poster/QUAST-LG_poster_ISMB2018.png
QUAST-LGに関するツイート
インストール
Linux and Mac OS X are supported.
依存
For the main pipeline:
- Python2 (2.5 or higher) or Python3 (3.3 or higher)
- Perl 5.6.0 or higher
- GCC 4.7 or higher
- basic UNIX tools (make, sh, sed, awk, ar)
For the optional submodules:
- Time::HiRes perl module for GeneMark-ES (needed when using --gene-finding --eukaryote)
- Java 1.8 or later for GRIDSS (needed for SV detection)
- R for GRIDSS (needed for SV detection)
- QUAST draws plots in two formats: HTML and PDF. If you need the PDF versions, make sure that you have installed Matplotlib.
本体 GIthub
バージョン5からQUASTのパッケージに QUAST-LGも含まれるようになった(conda link)。
#依存が多いので仮想環境に入れる
conda create -n quast -y
conda activate quast
conda install -c bioconda -y quast
#インストール後
#To be able to use those, please run
quast-download-gridss
quast-download-silva
quast-download-busco
> quast -h # v5.0.2
# quast -h
WARNING: Python locale settings can't be changed
QUAST: Quality Assessment Tool for Genome Assemblies
Version: 5.0.2
Usage: python /root/.pyenv/versions/miniconda3-4.0.5/bin/quast [options] <files_with_contigs>
Options:
-o --output-dir <dirname> Directory to store all result files [default: quast_results/results_<datetime>]
-r <filename> Reference genome file
-g --features [type:]<filename> File with genomic feature coordinates in the reference (GFF, BED, NCBI or TXT)
Optional 'type' can be specified for extracting only a specific feature type from GFF
-m --min-contig <int> Lower threshold for contig length [default: 500]
-t --threads <int> Maximum number of threads [default: 25% of CPUs]
Advanced options:
-s --split-scaffolds Split assemblies by continuous fragments of N's and add such "contigs" to the comparison
-l --labels "label, label, ..." Names of assemblies to use in reports, comma-separated. If contain spaces, use quotes
-L Take assembly names from their parent directory names
-e --eukaryote Genome is eukaryotic (primarily affects gene prediction)
--fungus Genome is fungal (primarily affects gene prediction)
--large Use optimal parameters for evaluation of large genomes
In particular, imposes '-e -m 3000 -i 500 -x 7000' (can be overridden manually)
-k --k-mer-stats Compute k-mer-based quality metrics (recommended for large genomes)
This may significantly increase memory and time consumption on large genomes
--k-mer-size Size of k used in --k-mer-stats [default: 101]
--circos Draw Circos plot
-f --gene-finding Predict genes using GeneMarkS (prokaryotes, default) or GeneMark-ES (eukaryotes, use --eukaryote)
--mgm Use MetaGeneMark for gene prediction (instead of the default finder, see above)
--glimmer Use GlimmerHMM for gene prediction (instead of the default finder, see above)
--gene-thresholds <int,int,...> Comma-separated list of threshold lengths of genes to search with Gene Finding module
[default: 0,300,1500,3000]
--rna-finding Predict ribosomal RNA genes using Barrnap
-b --conserved-genes-finding Count conserved orthologs using BUSCO (only on Linux)
--operons <filename> File with operon coordinates in the reference (GFF, BED, NCBI or TXT)
--est-ref-size <int> Estimated reference size (for computing NGx metrics without a reference)
--contig-thresholds <int,int,...> Comma-separated list of contig length thresholds [default: 0,1000,5000,10000,25000,50000]
-u --use-all-alignments Compute genome fraction, # genes, # operons in QUAST v1.* style.
By default, QUAST filters Minimap's alignments to keep only best ones
-i --min-alignment <int> The minimum alignment length [default: 65]
--min-identity <float> The minimum alignment identity (80.0, 100.0) [default: 95.0]
-a --ambiguity-usage <none|one|all> Use none, one, or all alignments of a contig when all of them
are almost equally good (see --ambiguity-score) [default: one]
--ambiguity-score <float> Score S for defining equally good alignments of a single contig. All alignments are sorted
by decreasing LEN * IDY% value. All alignments with LEN * IDY% < S * best(LEN * IDY%) are
discarded. S should be between 0.8 and 1.0 [default: 0.99]
--strict-NA Break contigs in any misassembly event when compute NAx and NGAx.
By default, QUAST breaks contigs only by extensive misassemblies (not local ones)
-x --extensive-mis-size <int> Lower threshold for extensive misassembly size. All relocations with inconsistency
less than extensive-mis-size are counted as local misassemblies [default: 1000]
--scaffold-gap-max-size <int> Max allowed scaffold gap length difference. All relocations with inconsistency
less than scaffold-gap-size are counted as scaffold gap misassemblies [default: 10000]
--unaligned-part-size <int> Lower threshold for detecting partially unaligned contigs. Such contig should have
at least one unaligned fragment >= the threshold [default: 500]
--skip-unaligned-mis-contigs Do not distinguish contigs with >= 50% unaligned bases as a separate group
By default, QUAST does not count misassemblies in them
--fragmented Reference genome may be fragmented into small pieces (e.g. scaffolded reference)
--fragmented-max-indent <int> Mark translocation as fake if both alignments are located no further than N bases
from the ends of the reference fragments [default: 85]
Requires --fragmented option
--upper-bound-assembly Simulate upper bound assembly based on the reference genome and reads
--upper-bound-min-con <int> Minimal number of 'connecting reads' needed for joining upper bound contigs into a scaffold
[default: 2 for mate-pairs and 1 for long reads]
--est-insert-size <int> Use provided insert size in upper bound assembly simulation [default: auto detect from reads or 255]
--plots-format <str> Save plots in specified format [default: pdf].
Supported formats: emf, eps, pdf, png, ps, raw, rgba, svg, svgz
--memory-efficient Run everything using one thread, separately per each assembly.
This may significantly reduce memory consumption on large genomes
--space-efficient Create only reports and plots files. Aux files including .stdout, .stderr, .coords will not be created.
This may significantly reduce space consumption on large genomes. Icarus viewers also will not be built
-1 --pe1 <filename> File with forward paired-end reads (in FASTQ format, may be gzipped)
-2 --pe2 <filename> File with reverse paired-end reads (in FASTQ format, may be gzipped)
--pe12 <filename> File with interlaced forward and reverse paired-end reads. (in FASTQ format, may be gzipped)
--mp1 <filename> File with forward mate-pair reads (in FASTQ format, may be gzipped)
--mp2 <filename> File with reverse mate-pair reads (in FASTQ format, may be gzipped)
--mp12 <filename> File with interlaced forward and reverse mate-pair reads (in FASTQ format, may be gzipped)
--single <filename> File with unpaired short reads (in FASTQ format, may be gzipped)
--pacbio <filename> File with PacBio reads (in FASTQ format, may be gzipped)
--nanopore <filename> File with Oxford Nanopore reads (in FASTQ format, may be gzipped)
--ref-sam <filename> SAM alignment file obtained by aligning reads to reference genome file
--ref-bam <filename> BAM alignment file obtained by aligning reads to reference genome file
--sam <filename,filename,...> Comma-separated list of SAM alignment files obtained by aligning reads to assemblies
(use the same order as for files with contigs)
--bam <filename,filename,...> Comma-separated list of BAM alignment files obtained by aligning reads to assemblies
(use the same order as for files with contigs)
Reads (or SAM/BAM file) are used for structural variation detection and
coverage histogram building in Icarus
--sv-bedpe <filename> File with structural variations (in BEDPE format)
Speedup options:
--no-check Do not check and correct input fasta files. Use at your own risk (see manual)
--no-plots Do not draw plots
--no-html Do not build html reports and Icarus viewers
--no-icarus Do not build Icarus viewers
--no-snps Do not report SNPs (may significantly reduce memory consumption on large genomes)
--no-gc Do not compute GC% and GC-distribution
--no-sv Do not run structural variation detection (make sense only if reads are specified)
--no-gzip Do not compress large output files
--no-read-stats Do not align reads to assemblies
Reads will be aligned to reference and used for coverage analysis,
upper bound assembly simulation, and structural variation detection.
Use this option if you do not need read statistics for assemblies.
--fast A combination of all speedup options except --no-check
Other:
--silent Do not print detailed information about each step to stdout (log file is not affected)
--test Run QUAST on the data from the test_data folder, output to quast_test_output
--test-sv Run QUAST with structural variants detection on the data from the test_data folder,
output to quast_test_output
-h --help Print full usage message
-v --version Print version
Online QUAST manual is available at http://quast.sf.net/manual
root@47647a654823:/data#
ラン
アセンブリした配列3つを評価する。
quast contig1.fa contig2.fa contig3.fa \
-R reference.fa.gz \
-G genes.txt \
-O operons.txt \
-1 reads1.fastq.gz -2 reads2.fastq.gz \
-o quast_test_output \
-t 8 --large
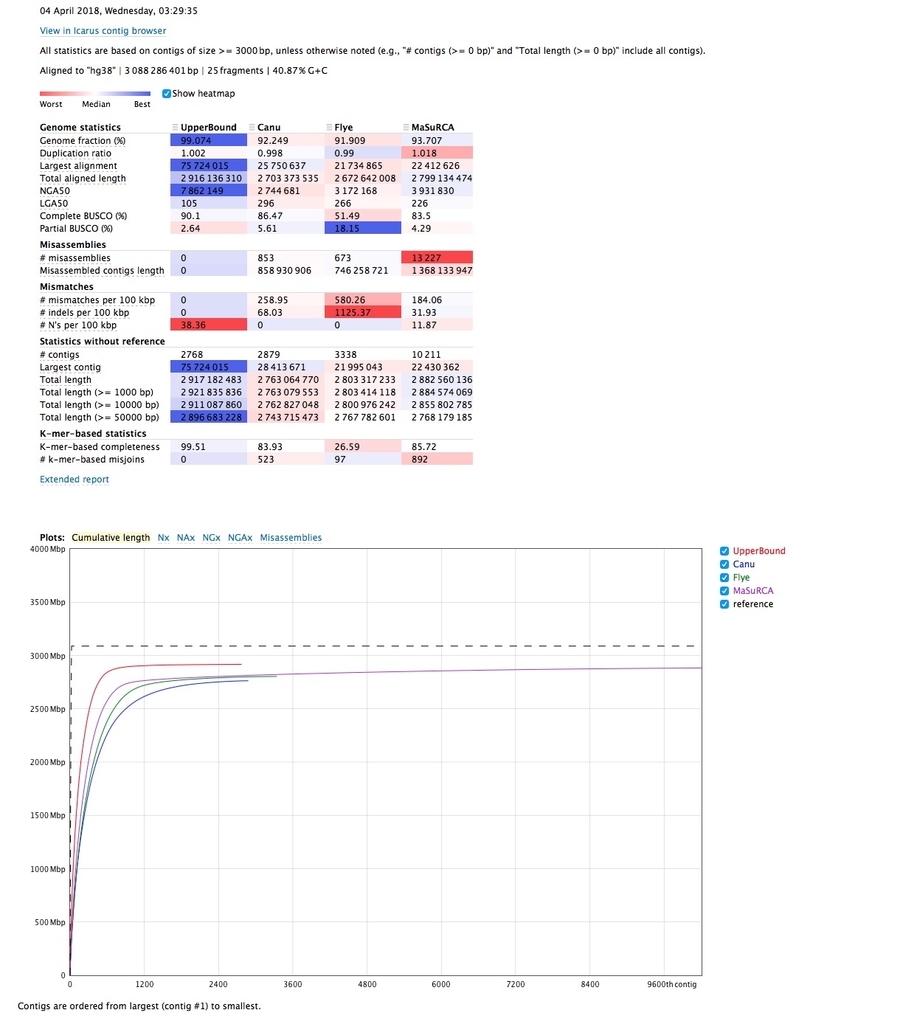
論文中のデータとしてQUAST-LG HPに登録されている、cell line HG001 (NA12878)の解析結果を見てみる( reportへのダイレクトリンク )。
下の方のExtended reportをクリックするとより詳細なmetricsが表示される。

Icarusを使い、contigのリファレンスへのアライメント結果を確認する。上のView in Icarus contig browserをクリックする。
表示する染色体を選択する。
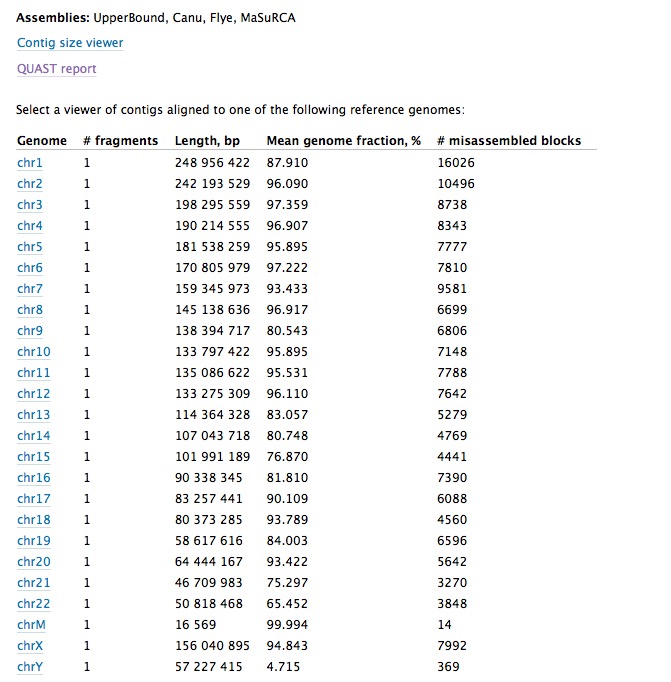
1番染色体を表示する。

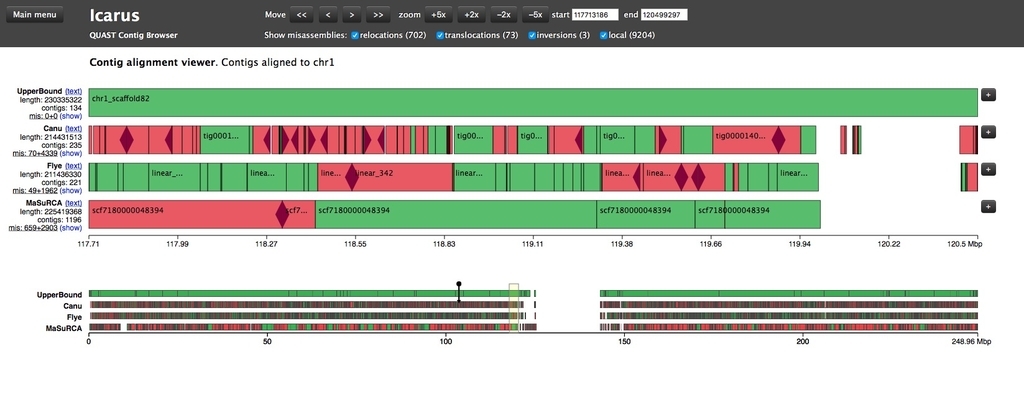
拡大縮小したり、左右にスクロールできる。

ブロックをクリックすれば、アライメントされた領域、ミスアセンブリのポジションなどを確認できる。

上のメニューからは、表示するミスアセンブリの種類を限定できる。ローカルミスアセンブリを表示。
公式ページにも記載されている評価項目を簡単にまとめておく。
リファレンスの有無に関係なく出力されるmetrics
- コンティグ数
- 最大コンティグ長(bp)
- N50、N75(アセンブリの連続性に関するmetrics。N50ならアセンブリの少なくとも50%をカバーする最短コンティグの長さで定義される)
- L50、L75(こちらはコンティグの数。L50なら、N50を満たす最小contig数として定義される)
- 予測された遺伝子数(GeneMark.hmm (for prokaryotes、or GlimmerHMM (for eukaryotes), or MetaGeneMark (for metagenomes)
- Nの数
リファレンスがある時のみ出力されるmetrics
- ミスアセンブリの数 (inversions, relocations, translocations, interspecies translocations (metaQUAST only) ).
- アライメントされたcontigの数とそのトータルサイズ
- アライメントされなかったcontigの数とそのトータルサイズ
- ミスマッチとindelの数
- Genome fraction %(リファレンスのカバー率)
- Duplication率(the total number of aligned bases in the assembly divided by the total number of those in the reference.)
- 全長 / 一部カバーされている遺伝子の割合(ユーザーが提供したリファレンスの遺伝子リストを使う)
- NG50(N50に似ているが、NG50はリファレンスの少なくとも50%をカバーする最短コンティグの長さ)
- NGA50, a reference-aware version of N50 metric. It is calculated using aligned blocks instead of contigs. Such blocks are obtained after removing unaligned regions, and then splitting contigs at misassembly breakpoints. Thus, NGA50 is the length of a block, such that all the blocks of at least the same length together cover at least 50% of the reference.
- Complete BUSCO (%)
- K-mer-based completeness
論文内では、最新のmummer4(紹介)やminimap2(紹介)によるcontigのアライメント、KMC3(紹介)によるk-mer評価を元にしたアセンブリ完全性の評価、BUSCO(紹介)によるハウスキーピング遺伝子のhit率に基づいたアセンブリ評価、などの詳細が書かれています。またSupplementary documentsには、アセンブラのパフォーマンス、ラージゲノム評価時のメモリ使用量などもまとめられています。
追記
eukaryoteのラージゲノム(fungiは"--fungus")、遺伝子予測、 rRNA予測、buscoによる評価、も行う。
quast contig1.fa contig2.fa contig3.fa contig4.fa contig5.fa contig6.fa \
-R reference.fa.gz \
--labels miniasm,redbeans,canu,flye,ra,shasta \
--eukaryote --large\
--gene-finding \
--rna-finding \
--conserved-genes-finding \
-1 reads1.fastq.gz -2 reads2.fastq.gz \
--nanopore ONT.fq.gz --pacbio pacbio.fq.gz\
-t 50 \
-o quast_output
引用
Versatile genome assembly evaluation with QUAST-LG
Mikheenko A, Prjibelski A, Saveliev V, Antipov D, Gurevich A
Bioinformatics. 2018 Jul 1;34(13):i142-i150
Icarus: visualizer for de novo assembly evaluation.
Mikheenko A, Valin G, Prjibelski A, Saveliev V, Gurevich A
Bioinformatics. 2016 Nov 1;32(21):3321-3323.
関連