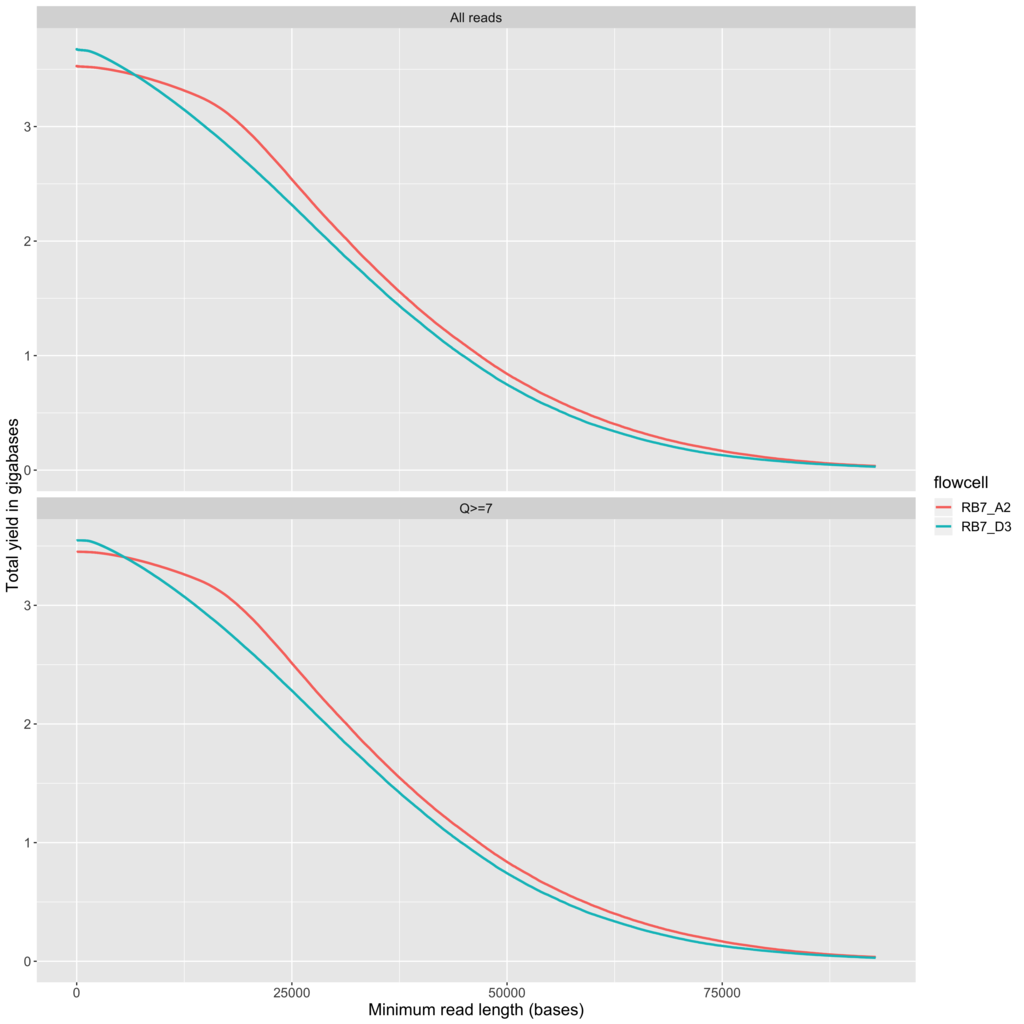
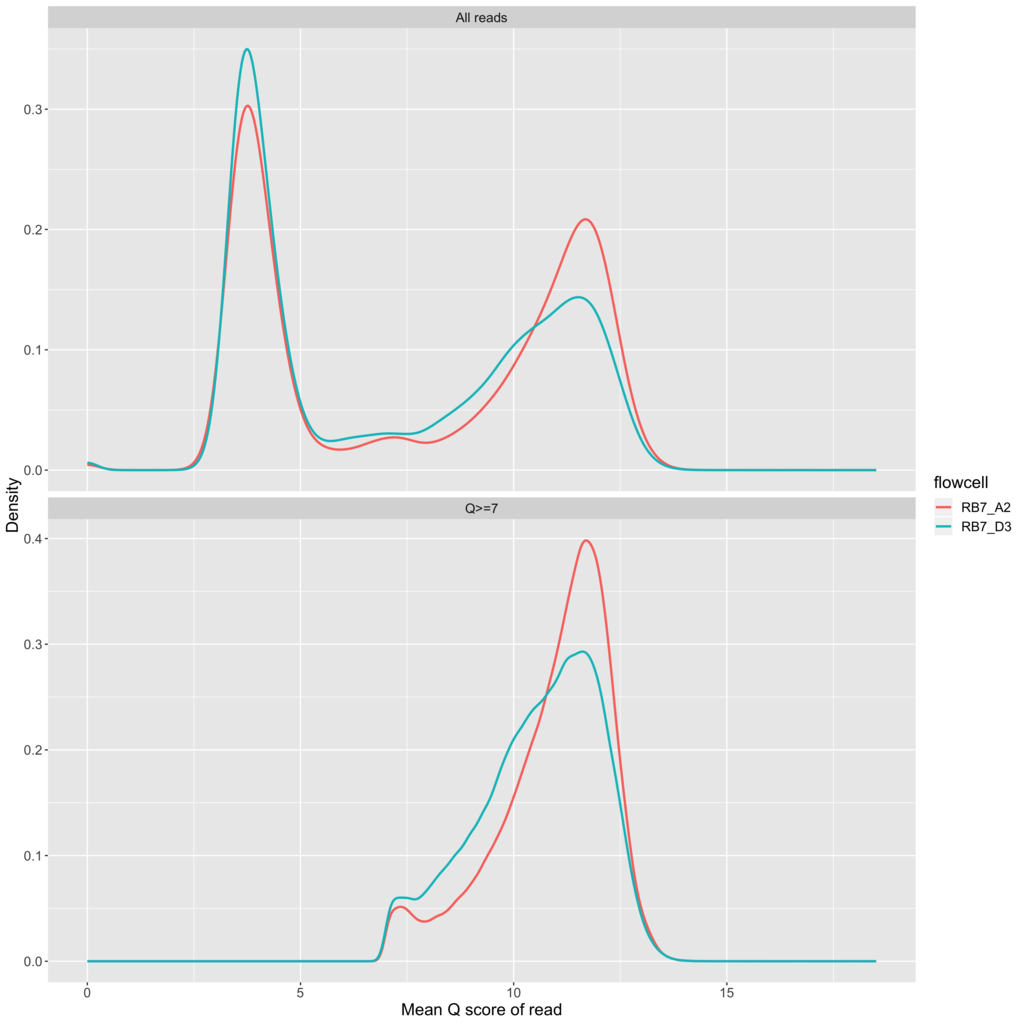
2019 5/23 tweet追記、9/26 動画追加、11/30 ツイート追記、12/22 統合TVリンク追加
2020 2/2 8章補足資料リンク追加、12/15 ツイート追加
2024/04/04 論文追加引用
RNAシークエンシング(RNA-Seq)[1]は、ゲノムワイドな発現解析のための日常的な技術となった。ますます低コストで、ライブラリー構築およびシーケンシングはしばしば標準的なプロトコルに従って実施することができる。多くの研究者、特にバイオインフォマティクスの経験がない研究者にとって、この技術を十分に活用するためのボトルネックは、発現プロファイルを実用的な洞察に変換する方法である。典型的な分析ワークフローは多くのステップを含み、各ステップは異なるツールを必要とする。これらのツールを正しく学習し、調整し、繋ぐのは時間がかかる。もう1つの障害は、多様な種類の遺伝子IDを持つ散在したアノテーションデータベースである。これらの問題を軽減するため、研究者は、RNA-Seqデータを分析するのに必要な時間と労力を大幅に削減できるアプリケーションの開発を目指している。
RNA-Seqデータ解析は、クオリティ管理、前処理、マッピング、そしてrawシークエンスリードのsummarizingから始まる。これらのステップは、従来のTuxedo Suite [ref.2、3]またはより高速でアライメントフリーの定量方法[ref.4、5]のいずれかを使用して完了したと想定する。これらのツールは、スタンドアロンでも、GenePattern [ref.6]、Galaxy [ref.7]、CyVerse [ref.8](紹介)のようなプラットフォームでも使用できる。
リードマッピングの後、よくあるのは、遺伝子レベルのリードカウントまたは正規化された発現レベルの行列(Fragments Per Kilobase Million、またはFPKM)を取得することである。Rは、そのDNAマイクロアレイデータのような表形式データの強力な可視化と統計分析ツールである。さらに、differentially expressed genes(DEG)および変更されたパスウェイを同定するために、多くの専用のRおよびBioconductor [ref.9]パッケージが開発されてきた。 DESeq2 [ref.10]のようないくつかのパッケージは、特にリードカウントの統計的モデリングのために開発され、広く使われている。しかし、これらのパッケージは時間がかかり、コーディング経験がないと研究者にとって手が届かないことさえある。
発現データを分析するいくつかのウェブアプリケーションが開発されている(論文 表1)。 STARTアプリ(shinnyトランスクリプトーム解析リソースツール)は、階層的クラスタリング、主成分分析(PCA)、遺伝子レベルのボックスプロット、およびDEGを実行するshinnyアプリである[ref.11]。別の同様のツールであるDegust [ref.12]はEdgeR [ref.13]またはlimma-voom [ref.14]を使って発現解析を実行し、インタラクティブに結果をプロットできる。他のツールには、Sleuth [ref.15]とShinyNGS [ref.16]がある。shinnyでないアプリケーションも開発された。これにはDEIVA [ref.17]とVisRseq [ref.18]が含まれる。いくつかのツールはパスウェイ分析のいくつかの能力を組み込んでいる。定量された発現データについては、ASAP(自動化シングルセル分析パイプライン)[ref.19]により、Gene Ontology(GO)[ref.20]およびKEGG [ref.21]データベースに基づいて正規化、フィルタリング、クラスタリング、およびエンリッチメント分析を行うことができる。 EXPath Tool [ref.22]を使用すると、ユーザーはパスウェイ探索、GOエンリッチメント解析および共発現解析を実行できる。 IRISなどの他のいくつかのShinyベースのツール[ref.23]も開発されている。過去数年間におけるこれらのツールの開発は、RNA-Seqデータの解釈を容易にした。
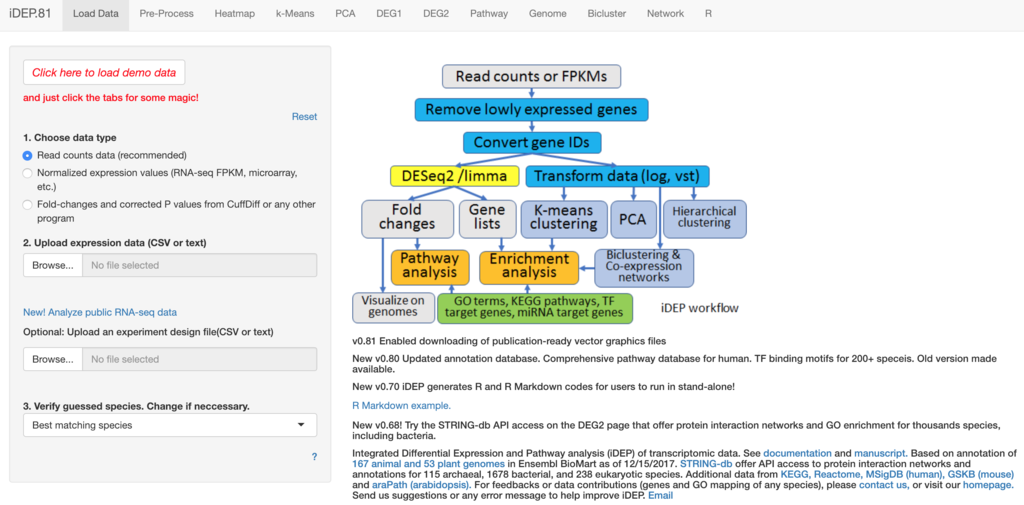
本研究では、(1)広範囲にわたる自動遺伝子ID変換、(2)植物と動物の両方に対する包括的な遺伝子アノテーションとパスウェイデータベース、(3)いくつかの方法、詳細なEDAおよびパスウェイ解析、(4)アプリケーションプログラミングインタフェース(API)を介したKEGG [ref.21]、STRING-db [ref.24]などのWebサービスへのアクセス、および(5)スタンドアロン解析用のRスクリプトの生成による再現性の向上、を含むwebアプリケーションを開発する。iDEPを使用して、2つのサンプルデータセットを分析し、論文表1と図1以外のすべての図と表を生成した。(以下略)
iDEP overview
https://idepsite.wordpress.com/
FAQ
https://idepsite.wordpress.com/faq/
データフォーマット
Data format – iDEP: Gain Insights from RNA-seq
2022/04/26
Cut the crap and let biologists interact with their data!
— Steven Ge (@StevenXGe) April 24, 2022
With iDEP 0.951 update https://t.co/JaBKVawVzZ, you can now easily visualize the expression of all genes on all KEGG pathways.
Just upload your data, define comparisons, and select any pathway at the KEGG tab. pic.twitter.com/MUIVkUxm5T
So happy that I improved the speed of data loading and converting in iDEP(https://t.co/kaZKnaRsF2). Much faster! Initially, I thought it is the database. But it turns out that I only need to examine a small portion of your genes to determine which species your data is from.
— Steven Ge (@StevenXGe) April 26, 2022
2021 10/20
New features for RNA-seq analysis tool iDEP (https://t.co/LlIaeQ71gL)
— Steven Ge (@StevenXGe) October 19, 2021
1. By default enrichment analysis is done using background defined by genes passed the filter.
2. Interactive network enabling users to easily visualize the relatedness of significant pathways EnrichmentMap pic.twitter.com/iFLI6FPPrP
2021 1/19
A new video demo for iDEP focusing on the biological questions.
— Steven Ge (@StevenXGe) 2021年1月19日
iDEP web application for RNA-Seq data analysis https://t.co/HBgFkmznDR via @YouTube
Our iDEP paper is finally out. In addition to the source code in Github, all the supporting databases are available on Zenodo.
— Steven Ge (@StevenXGe) December 20, 2018
iDEP: an integrated web application for differential expression and pathway analysis of RNA-Seq data https://t.co/9D1J1eX04A #bmcbioinformatics
2019/03/21
Big update to iDEP!
— Steven Ge (@StevenXGe) May 21, 2019
Besides the 315 genomes in Ensembl, we incorporated the annotation for 115 archaeal, 1678 bacterial, and 238 eukaryotic species from STRING-db. iDEP is designed to help smaller labs making sense of transcriptomic data.
Please help spread the word! pic.twitter.com/6rw0X7tFNw
Re-analyze public RNA-seq data can be done easily with interactive tools like iDEP and uniformly processed read counts data from ARCHS4 and DEE2. See how I did it in under 12 minutes.
— Steven Ge (@StevenXGe) September 23, 2019
No more excuses!
We just need good questions and critical thinkers!https://t.co/CZB28e0U0Y
使い方
local環境でも簡単に実行できるが、ここではidepサイトにアクセスして動作を確認する。
http://bioinformatics.sdstate.edu/idep/
v1.1 test mode
http://bioinformatics.sdstate.edu/idep11/
デモデータで読み込むフォーマットを確認する。左上のClick Hereをクリック。

読み込まれた。

1列目がgene IDになる。Ensembl IDを使うが、一般的なgene IDなら自動認識してEnsembl IDに内部変換してくれる。1行目にはサンプル名を記載する。Control、 TreatmentA、TreatmentBの3条件でそれぞれbiological/technical replicatesが2つずつあるなら、Ctrl_1, Ctrl_2, TrtA_1, TrtA_2, TrtB_1, TrtB_2のように記載する。これでreplicatesとして認識される。
実際に読み込めるデータは、RNA seqのリードカウントデータ(正規化前)、またはFPKMで正規化したデータ、マイクロアレイのデータなどになる。
他の入力例(データフォーマットの解説より)
Gene expression matrix ex.1

Gene expression matrix ex.2

fold-change and P values

Public RNA-Seq and ChIP-Seq dataでは、ARCHS4でマイニングされた7000以上のSRAサンプルの定量データ(CSVファイル)(paper)を解析できる。

DownloadからARCHS4でリードカウントされたデータのCSVをダウンロードして使ってみる。1行目はこのようにした。

左上のリードカウントからCSVをアップロード。

アップロード完了。フォーマットにエラーがあると赤字のメッセージが出る。gene IDは、Ensembl IDに変換され、以後の解析が行われる。IDはEnsembl release 92に基づいており、220の生物種に対応している(link)(2018/12/29現在)。
1、Pre-process(説明): データの正規化


replicatesとして認識されたサンプルはグラフで同じ色がアサインされている。動作は非常に俊敏で、データのアップロード完了から10秒くらいで結果は可視化される。
左上のウィンドウからパラメータを変更できる。デフォルトでは1サンプルで0.5 counts per million (CPM) 以上の遺伝子が分析対象となる。 pseudo countとして追加される値はデフォルト4になっている。

2、Heatmap(説明)階層的クラスタリングおよびデンドログラムとヒートマップによる可視化

defaultでは全サンプル間のSD top1000が対象となる。左上のgene SD distributionで可視化。


左上のinteractive heatmapボタンからスケールをリアルタイムに変更できる。50遺伝子に絞った。

グラフはマウスで操作できる。
図と対象遺伝子のCSVは左下からダウンロードできる。

3、k-means(説明)非階層クラスタリングおよびデンドログラムとヒートマップによる可視化

defaultでは全サンプル間のSD top 2000がクラスター分析対象となる。クラスター数のdefaultは4。
t-SNEによる次元圧縮結果

エンリッチされた転写因子(TF)結合モチーフ

エンリッチされたpathway

デンドログラムで可視化(左のVisualize enrichmentボタンより)

4、PCA(説明)主成分分析
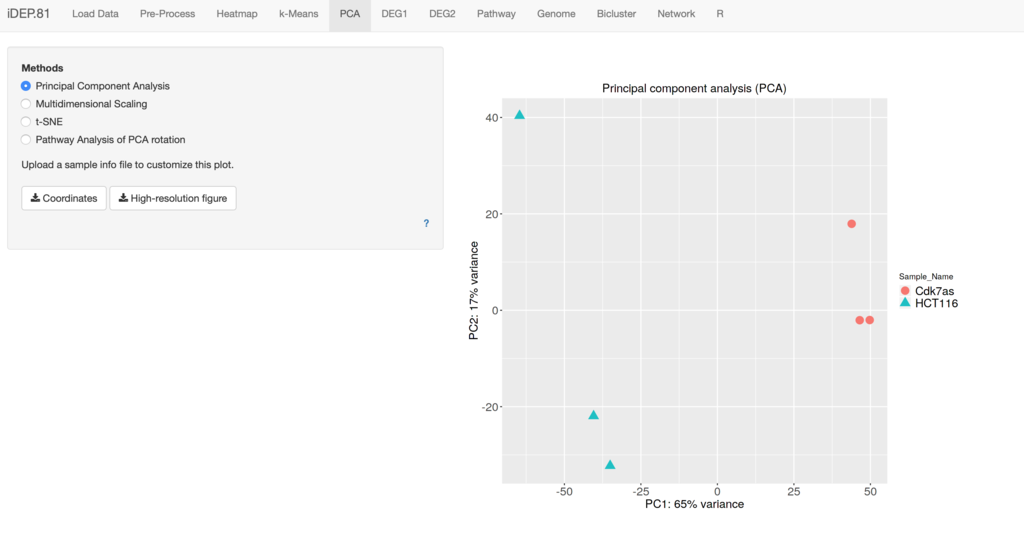
最初の次元は、サンプルを最もよく分離し、データの変動の最も大きな割合を説明する倍率変化を表す。それ以降の次元は効果が小さく、その前の次元と直交している。実験計画で複数の因子が含まれる場合、各因子を複数の次元で調査する。ある次元において、ある因子によってサンプルがクラスタリングされる場合、その因子は発現の違いに寄与していることが示唆される(線形モデリングに含める価値がある)。一方、ほとんどあるいは全く効果を示さない因子は、ダウンストリーム解析から除外できる。
5、DEG(説明)発現変動遺伝子の検出

デフォルトではDESeq2が使われる。FDR cutoffは0.1、Min fold changeは2。3つ以上のグループがある場合、全てのペアワイズ比較が実施される。これまでと同様に、結果は左のメニューからダウンロードできる。
ベン図(Venn Diagramボタンより実行できる)

3サンプルの場合
DEGのタブは2つある。DEG2のタブでは、3群以上のデータでも、組み合わせを選び、それぞれ2群比較を実行できる。結果はヒートマップ、MA-plot、Scatter plotなどで可視化される。また、検定されてエンリッチされていると判定されたpathwayが下にp-value付きで表示される。
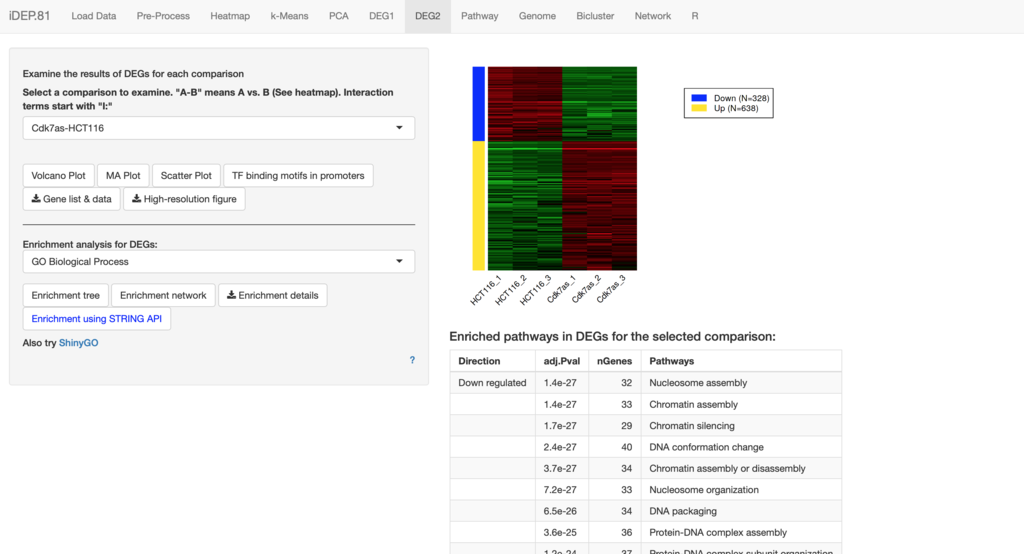
操作パネル左下にはShinyGO(prepirnt)へのリンクもある。
ShinyGOはユーザーが指定した遺伝子リストを元に、エンリッチされた系を可視化する。機能はiDEPとかなり重複しているが、iDEPにないツールもある。
2群間比較結果の可視化
Scatter plot

MA plot

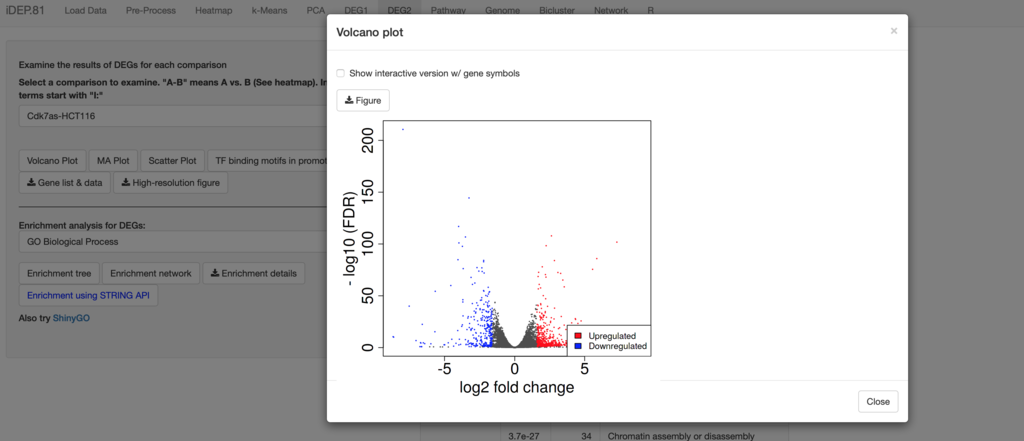
Volcano plot
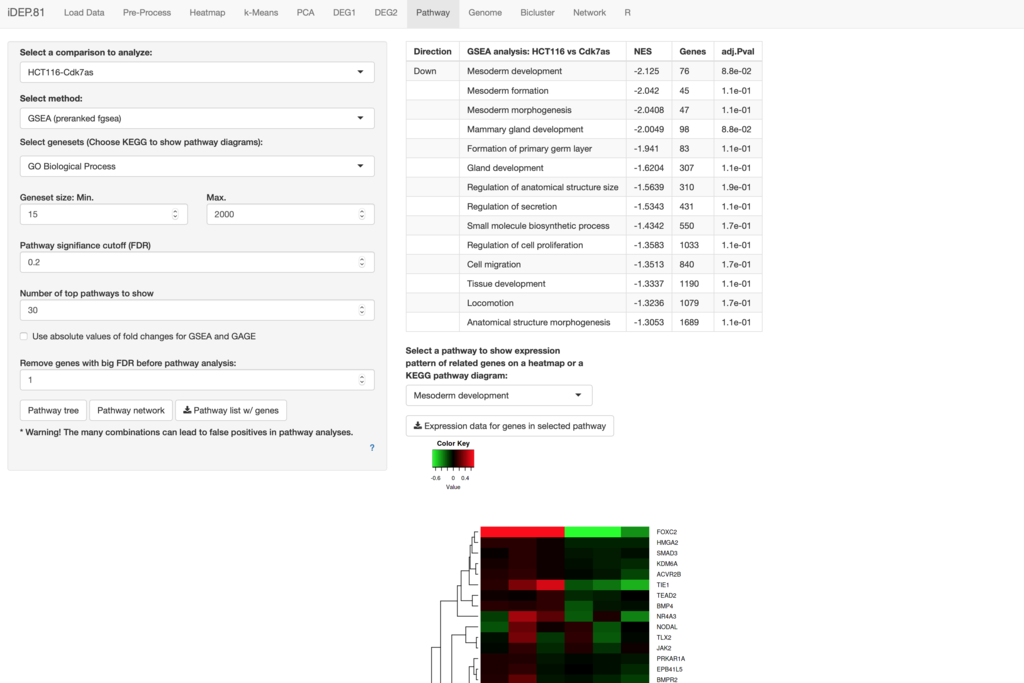
6、Pathway(説明)GOエンリッチメント解析


pathway解析はDEGで指定してフィルタリングした条件ではなく、全データからDESeq2 / limmaで出力したfold change値を使って行われることに注意する。有意水準を決めるFDR cutoffはpathway解析パネルにもあるので、こちらで厳密さは調節する。
有名な、いくつかのGOエンリッチメント解析ツールを利用できる。
GAGE(Generally Applicable Gene-set Enrichment )(pubmed)(遅い)
GSEA (Gene Set Enrichment Analysis) (preranked fgsea)(link)(解説HP)
PGSEA: PAGE (Parametric Analysis of Gene Set Enrichment) の実装 (PDF link)2群間比較

PGSEA w/all sample: 全サンプル間比較

上記の分析は全てbuint-inのデータを使っているが、最後のReactomePA (Reactome Pathway Analysis) は、 Reactomeの遺伝子セットデータベースを使う(Reactome 統合TV解説)。
 Reactome (HP) は歴史あるデータベースで、ヒト以外のモデル生物種にも対応している。ペーパーは多数出ている (Google scholar検索結果)。
Reactome (HP) は歴史あるデータベースで、ヒト以外のモデル生物種にも対応している。ペーパーは多数出ている (Google scholar検索結果)。
たくさんのgenesetを利用できる。一覧はマニュアル参照(link)。KEGGに切り替えてエンリッチと判定されたpathwayを可視化する。

Down regulationが35で、NES(Normalised enrichment score for the given gene set)がもっとも低かったHedgehog signaling pathway

可視化するKEGG pathwayは図の上のメニューから選ぶ。
7、Genomes DGEのGenome上の位置を可視化する

8、Biclustering(説明)サンプル数の多いラージデータセットの解析で(>10)、相関関係にある遺伝子のグループを検出する

biclust R、BCCC、BCXmotifs、BCPlaid、BCSpectral、BCBimax、BCQuest、QUBICなどのRパッケージを利用できる。解説は上の説明リンク参照。
9、Network(説明)共発現解析およびネットワークの可視化。かなり巨大なデータセットで(>15)相関関係がありそうな遺伝子セットを探すために使う(小さなデータセットではクラスタリング解析を行う)。
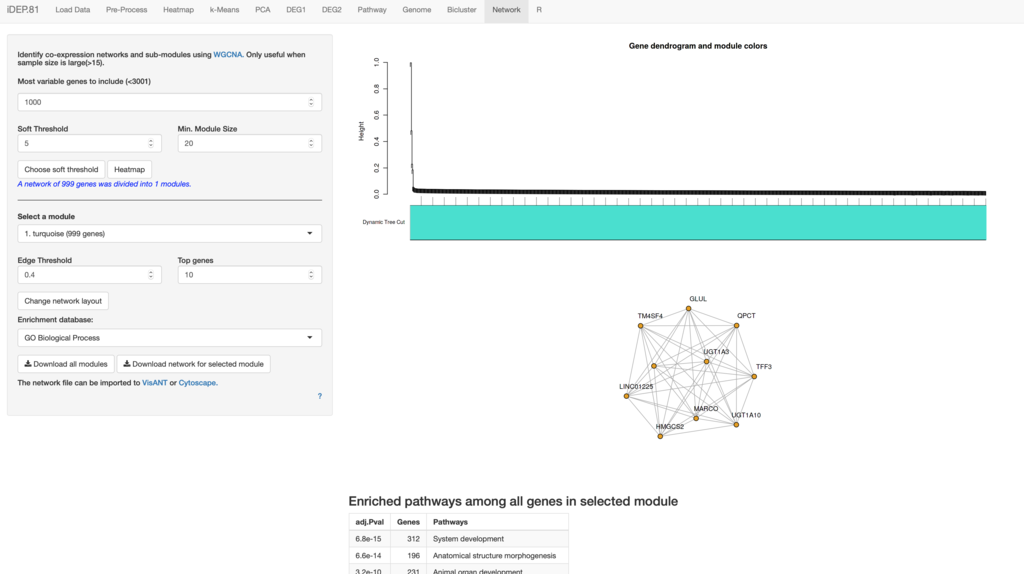
ネットワークは巨大になるので、全ネットワークの可視化には、localでcytoscape等を使うことが推奨されている。
感想
インタラクティブにパラメータを変更して、結果を見比べながら進められる素晴らしいツールですね。salmonやkallistoなどと組み合わせれば、rawデータが手に入ってから数時間以内にラフな結果は出せるのではないでしょうか(*1)。もちろん、方法について理解していないと使いこなせない訳で、誰でもRNA seq解析できるとは言いません。しかし、RNA seq解析の敷居が下がっているのは確かで、これだけ完成度の高いアプリケーションが出てくると、リードカウントはCyverse(紹介)、DEG検出などはiDEP、と使い分けることで、コンソールが一切使えない環境でも結果を出せてしまえますね。
2019 11/30追記
羊土社さんからRNA-Seqデータ解析の専門書が発売されました。その中の1章で、iDEPの使い方を紹介しています。よろしければ手にとってご確認下さい。どの章もたいへん見応えのある内容になっています。
【新刊】『RNA-Seqデータ解析 WETラボのための鉄板レシピ』発売! 食材調達(データ入手)から盛付(論文投稿)までシェフ直伝!「内容の更新情報が届く」という驚きの付録つき! #RNAseqRecipe https://t.co/bPPUTmA2tt
— 羊土社 実験医学 (@Yodosha_EM) 2019年11月29日
実験医学別冊:RNA-Seqデータ解析 WETラボのための鉄板レシピ - 羊土社
8章補足資料
2020 12/14
#RNAseqRecipe の Annual Update が出たようですゾ “実験医学別冊RNA-Seqデータ解析 WETラボのための鉄板レシピ 坊農秀雅/編 2019年11月29日発行 AB判 255ページ 付録:Annual Updateサービスの登録コード ISBN 978-4-7581-2243-6” @Yodosha_sheep / “実験医学別冊:RNA-S…” https://t.co/YJe6vJKIIL
— bonohu / ぼうのう ひでまさ (@bonohu) 2020年12月14日
2019 12/22
ShinyGO
2020 4/21
統合TVでBioJupiesの使い方が紹介されています。
BioJupiesを使ってウェブブラウザ上でRNA-seqデータ解析を行う
2020 5/26
2022/10
少し前にメールで連絡をいただきましたが、完全に新しくなったiDEP 1.0がテストモードで公開されています。
20/27
iDEPにファイルをアップするとすぐに切断されるという現象を経験しましたが、原因は、自分の不注意で、アップしたテキストファイルの先頭に不要な空白のタブが挿入されていたためでした。タブの後ろに隠れスペースがあってもこの現象が発生します。注意してください。
引用
iDEP: an integrated web application for differential expression and pathway analysis of RNA-Seq data
Steven Xijin Ge, Eun Wo Son, Runan Yao
BMC Bioinformatics 2018 19:534
追記
iDEP Web Application for RNA-Seq Data Analysis
Xijin Ge
Methods Mol Biol. 2021:2284:417-443.
参考
slideshare バイオインフォマティクスによる遺伝子発現解析
https://www.slideshare.net/sesejun/ss-24923282
*1
75-bpのシングルエンドシーケンシングと組み合わせれば、ラン開始から24時間で解析結果まで得られることになる。
pre-processing
mapping
read count
関連ツール