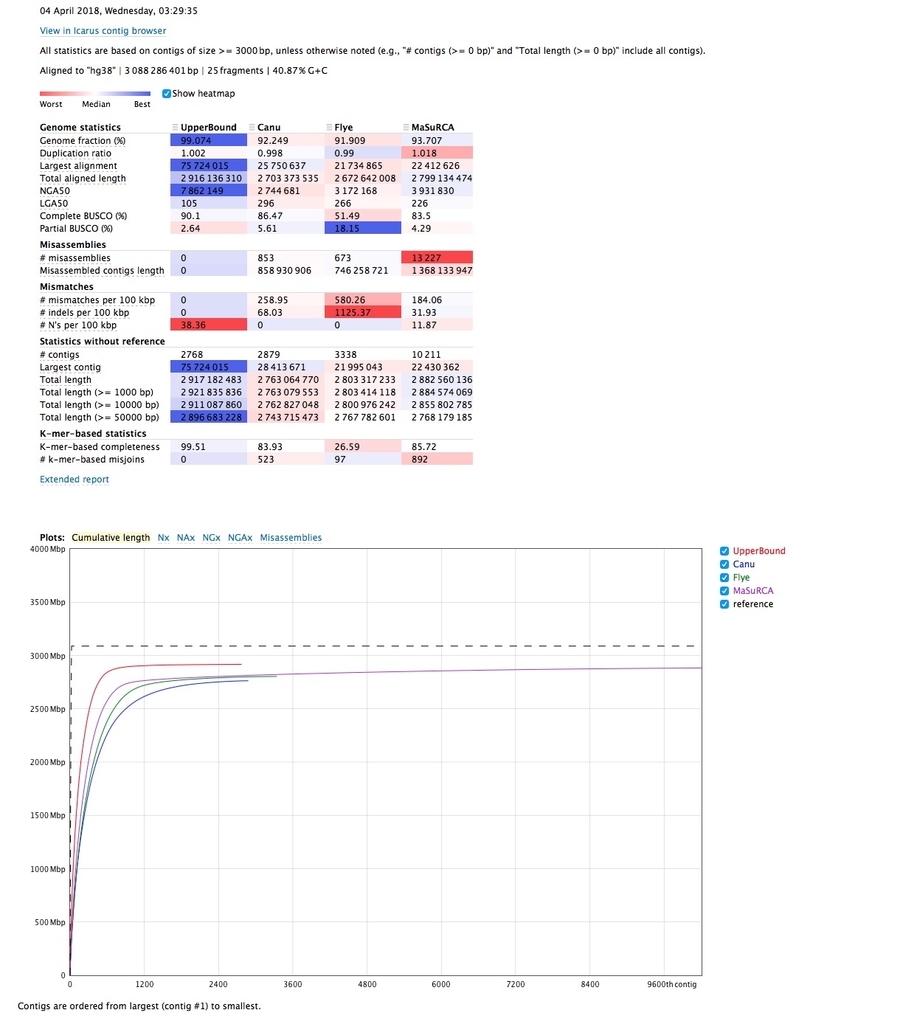
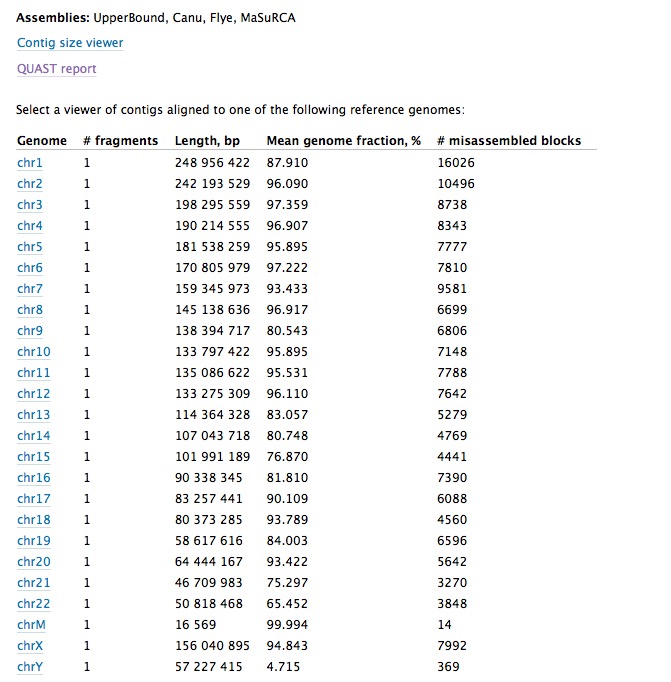
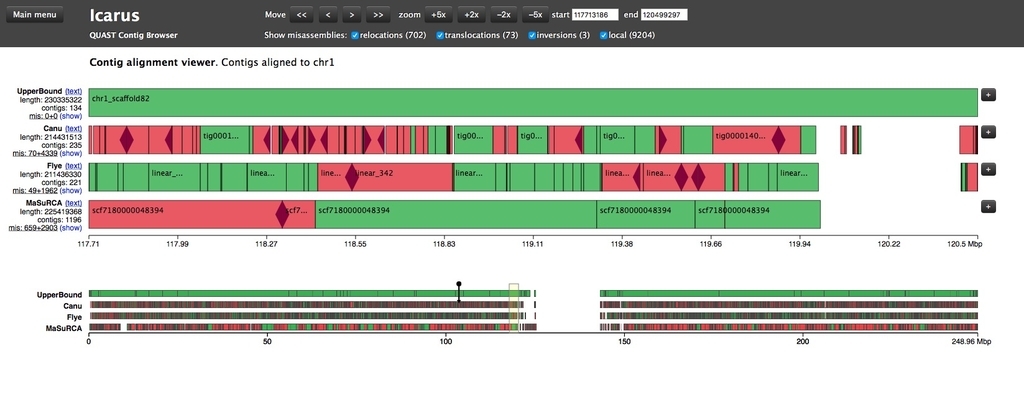
2020 4/1 関連ツールリンク追加
DNAシーケンシングデータによる生物の進化的および分類学的関係の再現は、バクテリアにおいて長い歴史を持つ(Cavalier-Smith、1993; Woese and 33Fox、1977; Woese、1987)。バクテリアは形態学的に区別し分類するのが難しく、DNAバーコードと分子系統学が細菌の関係を決定しようとする研究者のための選択肢となっている。しかしながら、DNAシークエンスの使用による系統発生的関係を解決することは困難な作業であり得る。適切な遺伝マーカーの選択、分類を区別するのに十分な情報、比較を有効かつ決定的にするために十分な相同性を有するものを選ぶのが正しい再構成のために不可欠になる(Capella-Gutierrez et al、402014; Wu et al。しばしば、情報と保存の間のこのようなトレードオフを補うために、低レベル(strain/species/genus)および高レベル(family/class/order/phylum)の異なるマーカーが系統解析に使用される(Capella-Gutierrez et al 、2014; Wu et al。、2013)。
バクテリアでは、現在、16S rDNAがほとんどの系統学的および生態学的研究のための選択肢として、比類のない普遍的に適用されるマーカーである。このマーカーはいくつかの可変領域および保存領域が存在し、情報の豊富な領域の増幅が可能でclosely relatedな分類群の区別を可能にする。 16S rDNAはすべての原核生物種にわたってよく保存されており、phyla間の比較が可能である。しかし、このアプローチには、ハイレベル分析とローレベル分析(Capella-Gutierrez et al、2014; Wu et al、2013)の両方に関して、その限界がある。 バクテリアの系統発生は依然としてその基礎の枝の部分で未解決であり、これらは単一のマーカーを用いて解決される可能性は低い。さらに、16S配列は、生態学的機能を有する特定の遺伝的に異なる株を区別するためのこのマーカーの可能性を排除する(Jaspers and Overmann、2004)、すなわち、大規模なゲノム再構成にもかかわらず株間で同一であり得る。さらに、リボソームrDNAは各ゲノムに複数コピー存在し、コピーが同一ゲノム内で可変なこともある(Větrovskýand Baldrian、2013)。両方の効果は、特に、機能的または生態学的な解析において、また相互主義的または病原性の宿主 - バクテリア関係と同様に、分類学的帰属の解釈を混乱させる可能性がある。
16S rDNAベースの系統発生解析は、バクテリアの同一性と進化的関係を理解する上で非常に重要であり、1つのマーカー配列で系統樹を計算することには欠陥がある。ハイスループットシークエンシング技術における現在の進歩により、この単一のマーカーコンベンションよりも広範な分析が可能になる。バクテリアゲノムは、大多数の真核生物と比べると、通常130kbpから14Mbpの範囲にサイズが制限されている。bp当たりのコストダウンと小さなゲノムサイズにより、限られた財源しか有さないワーキンググループであっても、完全なバクテリアゲノムのシーケンシングが可能になっているKeller et al、2014; Metzker、2009)。これはまた、シーケンシングされ、depositされるコンプリートなバクテリアゲノムの増加を導く(Pruitt et al、2007; Uchiyama et al。、2013)。
しかしながら、ゲノム全体から系統発生を導き出すことは、それ自体チャレンジングである。 例えば、大抵の場合、明確な類似性がない大きなゲノム領域が存在する。 また、相同性を有する領域については、下流の系統分析のために抽出する必要があり、これは広範なバイオインフォマティクスの専門知識を必要とするプロセスである。この課題に取り組む一つの方法は、あらかじめアライメントが計算されたタイトなゲノムクラスター(ATGC、Novichkov et al、2009)のデータベースを構築し、高分解能なmicro-evolutionary analysesを可能にすることである。しかし、利用可能なゲノムのうちのほんの一部しか含まれておらず、ユーザが提供もできないため、このアプローチは限定されている。この問題の1つの解決策は、関心のある生物の大多数に存在する保存された領域に集中することである(Ciccarelli et al、822006)。
ここでは、全ゲノムデータのアミノ酸配列から必須の107個のシングルコピー遺伝子のセットを同定および抽出するツールであるbcgTreeを報告する。ここで使用される「essential core genes」の定義は、Dupont et al(2012)(pubmed)の研究に基づいており、生物学的ではなく統計的な議論に従う。本発明者らのソフトウェアは、コア遺伝子配列データを自動的にコンパイルし、これを用いて分割最尤解析を用いて系統樹を再構築する。 妥当性確認の目的のために、本発明者らは、Lactobacillalesで現在利用可能なほとんどのゲノムを含む、90のLactobacillus属の系統発生の解明にbcgTreeを適用した。さらに、評価目的のために、高レベルおよび低レベルの系統発生分析テストを対応する16S rDNAツリーと直接比較した。

bcgTreeパイプライン。論文より転載。
依存
- perl5、Java Runtime Environment (version8)
以下のperlモジュールとjavaフレームワークはレポジトリに含まれている。
- Getopt::Long
- Pod::Usage
- FindBin
- File::Path
- File::Spec
- SwingX 1.6.5 (LGPL)
必要な外部ツール
- hmmsearch (HMMER version 3.1b1) - Eddy et al, 2010. HMMER3: a new generation of sequence homology search software.
- muscle (v3.8.31) - Edgar et al, 2004. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32, 1792–1797.
- Gblocks (version 0.91b) - Castresana et al, 2000. Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis. Mol. Biol. Evol. 17, 540–552.
- RAxML (version 8.2.4) - Stamatakis et al, 2014. RAxML version 8: A tool for phylogenetic analysis and post-analysis of large phylogenies. Bioinformatics 30, 1312–1313.
#Anaconda環境にて
conda install -c bioconda raxml==8.2.4
conda install -c bioconda gblocks==0.91b
conda install -c bioconda muscle==3.8.31
conda install -c bioconda hmmer==3.1b2
本体 github
git clone https://github.com/molbiodiv/bcgTree.git
# Now you can run bcgTree.pl
bcgTree/bin/bcgTree.pl --help
# Or start the java GUI
cd bcgTree/bcgTreeGUI
java -jar bcgTree.jar
CUI版のヘルプ
> perl bcgTree/bin/bcgTree.pl --help
$ perl bcgTree.pl -help
Usage:
$ bcgTree.pl [@configfile] --proteome bac1=bacterium1.pep.fa --proteome bac2=bacterium2.faa [options]
Options:
[@configfile] Optional path to a configfile with @ as prefix.
Config files consist of command line parameters
and arguments just as passed on the command
line. Space and comment lines are allowed (and
ignored). Spreading over multiple lines is
supported.
--proteome <ORGANISM>=<FASTA> [--proteome <ORGANISM>=<FASTA> ..]
Multiple pairs of organism and proteomes as
fasta file paths
[--outdir <STRING>] output directory for the generated output files
(default: bcgTree)
[--help] show help
[--version] show version number of bcgTree and exit
[--check-external-programs]
Check if all of the required external programs
can be found and are executable, then exit.
Report table with program, status (ok or
!fail!) and path. If all external programs are
found exit code is 0 otherwise 1. Note that
this parameter does not check that the paths
belong to the actual programs, it only checks
that the given locations are executable files.
[--hmmsearch-bin=<FILE>] Path to hmmsearch binary file. Default tries if
hmmsearch is in PATH;
[--muscle-bin=<FILE>] Path to muscle binary file. Default tries if
muscle is in PATH;
[--gblocks-bin=<FILE>] Path to the Gblocks binary file. Default tries
if Gblocks is in PATH;
[--raxml-bin=<FILE>] Path to the raxml binary file. Default tries if
raxmlHPC is in PATH;
[--threads=<INT>]
Number of threads to be used (currently only relevant for raxml).
Default: 2 From the raxml man page: PTHREADS VERSION ONLY! Specify
the number of threads you want to run. Make sure to set "-T" to at
most the number of CPUs you have on your machine, otherwise, there
will be a huge performance decrease!
[--bootstraps=<INT>]
Number of bootstraps to be used (passed to raxml). Default: 100
[--min-proteomes=<INT>]
Minimum number of proteomes in which a gene must occur in order to
be kept. Default: 2 All genes with less hits are discarded prior to
the alignment step. This option is ignored if --all-proteomes is
set.
[--all-proteomes]
Sets --min-proteomes to the total number of proteomes supplied.
Default: not set All genes that do not hit all of the proteomes are
discarded prior to the alignment step. If set --min-proteomes is
ignored.
[--hmmfile=<PATH>]
Path to HMM file to be used for hmmsearch. Default:
<bcgTreeDir>/data/essential.hmm
[--raxml-x-rapidBootstrapRandomNumberSeed=<INT>]
Random number seed for raxml (passed through as -x option to raxml).
Default: Random number in range 1..1000000 (see raxml command in log
file to find out the actual value). Note: you can abbreviate options
(as long as they stay unique) so --raxml-x=12345 is equivalent to
--raxml-x-rapidBootstrapRandomNumberSeed=12345
[--raxml-p-parsimonyRandomSeed=<INT>]
Random number seed for raxml (passed through as -p option to raxml).
Default: Random number in range 1..1000000 (see raxml command in log
file to find out the actual value). Note: you can abbreviate options
(as long as they stay unique) so --raxml-p=12345 is equivalent to
--raxml-p-parsimonyRandomSeed=12345
[--raxml-aa-substitiution-model "<MODEL>"]
The aminoacid substitution model used for the partitions by RAxML.
Valid options for RAxML 8.x are: DAYHOFF, DCMUT, JTT, MTREV, WAG,
RTREV, CPREV, VT, BLOSUM62, MTMAM, LG, MTART, MTZOA, PMB, HIVB,
HIVW, JTTDCMUT, FLU, STMTREV, DUMMY, DUMMY2, AUTO, LG4M, LG4X,
PROT_FILE, GTR_UNLINKED, GTR bcgTree will not check whether the
provided option is valid but rather pass it to RAxML literally.
Default: AUTO
[--raxml-args "<ARGS>"]
Arbitrary options to pass through to RAxML. The ARGS part should be
in quotes and is appended to the RAxML command as given.
ラン
GUI版を起動する(違いはない。CUI版のラッパー、いわゆるガワ)。
cd bcgTree/bcgTreeGUI/
java -jar bcgTree.jar
Add proteomesから比較する生物種のproteomeのfastaファイル (amino-acids.fasta) を入力する。

バクテリアのprotein.fasta5つを登録したところ。
GIthubより。

SupplementaryにはbcgTreeのランニングタイムの図もあります。
引用
bcgTree: automatized phylogenetic tree building from bacterial core genomes
Ankenbrand MJ, Keller A
Genome. 2016 Oct;59(10):783-791. Epub 2016 May 11.
PhyloPhlAnは以前簡単に紹介しています。
関連