現在、ほとんどのゲノムアセンブリプロジェクトは、アセンブリのより包括的な表現を提供するアセンブリグラフよりもむしろコンティグおよびscaffoldsに焦点を合わせている。 大きなアセンブリグラフのインタラクティブな視覚化は未解決の問題であり続けているため、本著者らは大きなアセンブリグラフを視覚化するAssembly Graph Browser (AGB) ツールを開発し、以前に開発された視覚化アプローチの機能を拡張した。 AGBは、リピート分析、縮小されたアセンブリグラフ(すなわち、選択された一組のエッジを折りたたむことによって得られたグラフ)の構築、および大きなアセンブリグラフを視覚化するための新しいアプローチを含む多数の新しい機能を含んでいる。
manual
https://almiheenko.github.io/AGB/manual.html
GFA1、GFA2に対応しています(2019年3月現在)。
インストール
macos10.14のminiconda3-4.3.14環境でテストした。
#condaの仮想環境を作って導入することが推奨されている
mamba create -c almiheenko -c bioconda -n AGB agb -y
conda activate AGB
#または直接導入
mamba install -c almiheenko -c bioconda -y agb
#またはソースからビルド
git clone https://github.com/almiheenko/AGB.git
cd AGB/
./setup.py install
> agb.py -h
$ agb.py -h
Usage:
1) /Users/kazuma/.pyenv/versions/miniconda3-4.3.14/bin/agb.py [options] --graph assembly_graph_file -a <assembler_name> [--fasta file_with_graph_edge_sequences]
2) /Users/kazuma/.pyenv/versions/miniconda3-4.3.14/bin/agb.py [options] -a <assembler_name> -i <assembler_output_dir> (supported for Canu, Flye, SPAdes)
The program will create interactive assembly graph viewer
Options:
-h, --help show this help message and exit
Common Options:
Options that can be used in any mode
-a ASSEMBLER, --assembler=ASSEMBLER
Required. Assembler name.
-o OUTPUT_DIR Output directory [default: agb_output]
-r REFERENCE Path to the reference genome
-t THREADS Maximum number of threads [default: 4]
-m MIN_EDGE_LEN Lower threshold for edge length [default: 500]
--meta Use QUAST options for metagenome
Special Options:
-i INPUT_DIR Assembler output folder
--graph=INPUT_FILE Assembly graph in GFA1/GFA2/Graphviz/FASTG format.
Cannot be used with -i option
FASTA file with graph edge sequences. Cannot be used
with -i option
テストラン
GFAファイルを指定する。オーサーらが用意したassembly graphを可視化してみる。
git clone https://github.com/almiheenko/AGB.git
cd AGB-master/test_data/
#canuのデータを使用。-aでアセンブラを指定する。Canu, Flye, SPAdesの3つがサポートされている。
agb.py --graph canu.contigs.gfa -a canu
- -a <ASSEMBLER> Required. Assembler name.
- --graph Cannot be used with -i option
ランが終わるとagb_output/ができる。

htmlを開く。
Bandageでは全graphを一画面に視覚化するが、AGBではGFAに記載されたedgeの頂点(vertex、またはnode)を介した繋がりを一定の単位、コンポーネント単位で視覚化する。コンポーネント切り替えはviewer画面上の方にある矢印<- ->から行う。

アセンブリのgraphは方向性のある有向graphなので、向き(矢印で表す)がある。
Edge(contig配列)をクリックすればそのedgeの長さとカバレッジが表示される。また、trackpadの上下やマウスで拡大縮小でき、ドラッグすればスクロールできる。

あくまでedgeが配列であることに注意する。頂点 (node) はアセンブル結果をgraphとして表現するため生じているジャンクションで、頂点にはシーケンス配列は含まれない(よく使われるBandageでは、例外的にcontig配列を頂点、contig間のリンケージをedgeにして可視化している)。
Show helpから、各記号の意味を確認できる。

In Degreeは頂点(node)に対して向かっているedgeの数、Out degreeは頂点(nodes)から外に向かっているedgeの数を意味する。
頂点(node)をクリックすれば関連するedgeに色が付く。edgesの向きによって色は変わる。

緑edgeはincoming edge、紫edgeはoutcoming edgeを表す。
もう1つテストする。
agb.py --graph=flye_yeast/assembly_graph.gfa -a flye -o oudir -t 12
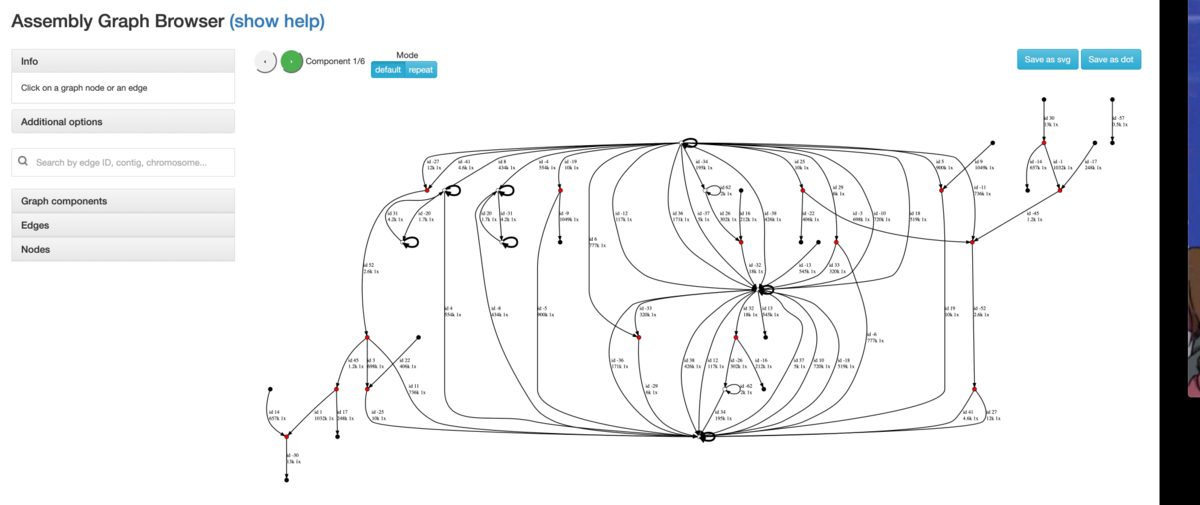
FlyeやCanuではedgeがrepetitive regionかどうかの情報も出力している。repeat-focused modeでは、これを利用してrepetitive regionのedgeを強調表示する。repetitive regionかどうかの情報が提供されない場合、AGBは各edgeのカバレッジをカバレッジ中央値で割った比でrepetitive regionかどうか判定する。
上のModeボタンから。default <=> repeat-focused mode 切り替えを行う。

リファレンスも指定すると、AGBは内部でQUAST-LGを走らせ、リファレンスへのアライメントを実行する。
agb.py --graph=flye_yeast/assembly_graph.gfa -a flye -o oudir -t 12 -r reference_genome.fa
リファレンスに沿ってgraphが並ぶ。

trackpadの上下やマウスで拡大縮小し、ドラッグで移動できる(リファレンスもgraphもそれぞれ独立に動くので移動するとずれる)。

引用
Assembly Graph Browser: interactive visualization of assembly graphs
Alla Mikheenko, Mikhail Kolmogorov
Bioinformatics, btz072, Published: 04 February 2019
関連