2019 5/17 condaインストール追記、イントロ文章修正、
2019 7/2タイトル修正
2019 7/4 インストール追記
2019 7/6 インストール追記タイトル修正、誤解を招く文章を削除
2019 10/8 インストール追記
2020 8/24 condaインストール追記
2021 7/16 mambaに変更
MetaPhlAnは、メタゲノムショットガンシーケンスデータから微生物群集の構成をプロファイリングするための計算ツールである。 MetaPhlAnは、〜17,000のリファレンスゲノム(〜13,500の細菌および古細菌、〜3,500のウイルス、〜110の真核生物)から同定された独自のクレード特異的マーカー遺伝子に依存している。
- 1秒間に最大25,000回のリード(1つのCPU上)の分析速度(既存の方法と比較して桁違いに速い)。
- MetaPhlAnマーカーとしての明確な分類アサインはクレード固有のものである。
- 生物学的相対abundanceの正確な推定(リードの割合よりもむしろ細胞数の観点から)。
- 細菌、古細菌、真核生物、ウイルスの種レベルの解像度。
- いくつかの合成データセットおよび何千もの実際のメタゲノムに関するプロファイリング精度の広範な検証。
2016年には、同じ研究チームからPanPhlAnも発表されている。PanPhlAnは種内の多様性を遺伝子の有無で評価するツールである。MetaPhlAn2でメタゲノムにどんな種のゲノムがあるか調べ、PanPhlAnで種内の多様性を調べることが可能になった。
マニュアル
https://bitbucket.org/biobakery/biobakery/wiki/metaphlan2
オーサーらのツール一覧
http://segatalab.cibio.unitn.it/tools/
インストール
condaやbrewで本体含め全ての依存をワンライナーで導入できる。
#全て仮想環境に入れてしまう
mamba create -n metaphlan2 python=2.7 -y
conda activate metaphlan2
#metaphylan2
mamba install -c bioconda -y metaphlan2
#or (faster)
mamba install -c bioconda/label/cf201901 metaphlan2
#hclust2
mamba install -c bioconda -y hclust2
#graphlan
mamba install -c bioconda -y graphlan
mamba install -c bioconda -y export2graphlan
#homebrew
brew install biobakery/biobakery/graphlan
brew install metaphlan2
#docker
docker pull biobakery/metaphlan2:2.7.7
#help
docker run --rm biobakery/metaphlan2 metaphlan2.py -h
#run
docker run --rm -itv $PWD:/data -w /data biobakery/metaphlan2 metaphlan2.py -h
util/のスクリプトが入ってないなら、hg cloneして直接本体を叩く(mergeのステップで使うスクリプト)。
#hgコマンドがないなら sudo apt update && apt install mercurial
hg clone https://bitbucket.org/biobakery/biobakery
> metaphlan2.py -h
# metaphlan2.py -h
usage: metaphlan2.py --input_type
{fastq,fasta,multifasta,multifastq,bowtie2out,sam}
[--mpa_pkl MPA_PKL] [--force]
[--bowtie2db METAPHLAN_BOWTIE2_DB] [-x INDEX]
[--bt2_ps BowTie2 presets] [--bowtie2_exe BOWTIE2_EXE]
[--bowtie2_build BOWTIE2_BUILD] [--bowtie2out FILE_NAME]
[--no_map] [--tmp_dir] [--tax_lev TAXONOMIC_LEVEL]
[--min_cu_len] [--min_alignment_len]
[--ignore_eukaryotes] [--ignore_bacteria]
[--ignore_archaea] [--stat_q] [--perc_nonzero]
[--ignore_markers IGNORE_MARKERS] [--avoid_disqm]
[--stat] [-t ANALYSIS TYPE] [--nreads NUMBER_OF_READS]
[--pres_th PRESENCE_THRESHOLD] [--clade] [--min_ab]
[-o output file] [--sample_id_key name]
[--sample_id value] [-s sam_output_file]
[--legacy-output] [--no-unknown-estimation]
[--biom biom_output] [--mdelim mdelim] [--nproc N]
[--install] [--force_download]
[--read_min_len READ_MIN_LEN] [-v] [-h]
[INPUT_FILE] [OUTPUT_FILE]
DESCRIPTION
MetaPhlAn version 2.9.12 (12 Jun 2019):
METAgenomic PHyLogenetic ANalysis for metagenomic taxonomic profiling.
AUTHORS: Nicola Segata (nicola.segata@unitn.it), Duy Tin Truong, Francesco Asnicar (f.asnicar@unitn.it), Francesco Beghini (francesco.beghini@unitn.it)
COMMON COMMANDS
We assume here that metaphlan2.py is in the system path and that mpa_dir bash variable contains the
main MetaPhlAn folder. Also BowTie2 should be in the system path with execution and read
permissions, and Perl should be installed)
========== MetaPhlAn 2 clade-abundance estimation =================
The basic usage of MetaPhlAn 2 consists in the identification of the clades (from phyla to species and
strains in particular cases) present in the metagenome obtained from a microbiome sample and their
relative abundance. This correspond to the default analysis type (-t rel_ab).
* Profiling a metagenome from raw reads:
$ metaphlan2.py metagenome.fastq --input_type fastq -o profiled_metagenome.txt
* You can take advantage of multiple CPUs and save the intermediate BowTie2 output for re-running
MetaPhlAn extremely quickly:
$ metaphlan2.py metagenome.fastq --bowtie2out metagenome.bowtie2.bz2 --nproc 5 --input_type fastq -o profiled_metagenome.txt
* If you already mapped your metagenome against the marker DB (using a previous MetaPhlAn run), you
can obtain the results in few seconds by using the previously saved --bowtie2out file and
specifying the input (--input_type bowtie2out):
$ metaphlan2.py metagenome.bowtie2.bz2 --nproc 5 --input_type bowtie2out -o profiled_metagenome.txt
* bowtie2out files generated with MetaPhlAn2 versions below 2.9 are not compatibile.
Starting from MetaPhlAn2 2.9, the BowTie2 ouput now includes the size of the profiled metagenome.
If you want to re-run MetaPhlAn2 using these file you should provide the metagenome size via --nreads:
$ metaphlan2.py metagenome.bowtie2.bz2 --nproc 5 --input_type bowtie2out --nreads 520000 -o profiled_metagenome.txt
* You can also provide an externally BowTie2-mapped SAM if you specify this format with
--input_type. Two steps: first apply BowTie2 and then feed MetaPhlAn2 with the obtained sam:
$ bowtie2 --sam-no-hd --sam-no-sq --no-unal --very-sensitive -S metagenome.sam -x ${mpa_dir}/metaphlan_databases/mpa_v25_CHOCOPhlAn_201901 -U metagenome.fastq
$ metaphlan2.py metagenome.sam --input_type sam -o profiled_metagenome.txt
* We can also natively handle paired-end metagenomes, and, more generally, metagenomes stored in
multiple files (but you need to specify the --bowtie2out parameter):
$ metaphlan2.py metagenome_1.fastq,metagenome_2.fastq --bowtie2out metagenome.bowtie2.bz2 --nproc 5 --input_type fastq
-------------------------------------------------------------------
========== Marker level analysis ============================
MetaPhlAn 2 introduces the capability of charachterizing organisms at the strain level using non
aggregated marker information. Such capability comes with several slightly different flavours and
are a way to perform strain tracking and comparison across multiple samples.
Usually, MetaPhlAn 2 is first ran with the default -t to profile the species present in
the community, and then a strain-level profiling can be performed to zoom-in into specific species
of interest. This operation can be performed quickly as it exploits the --bowtie2out intermediate
file saved during the execution of the default analysis type.
* The following command will output the abundance of each marker with a RPK (reads per kilo-base)
higher 0.0. (we are assuming that metagenome_outfmt.bz2 has been generated before as
shown above).
$ metaphlan2.py -t marker_ab_table metagenome_outfmt.bz2 --input_type bowtie2out -o marker_abundance_table.txt
The obtained RPK can be optionally normalized by the total number of reads in the metagenome
to guarantee fair comparisons of abundances across samples. The number of reads in the metagenome
needs to be passed with the '--nreads' argument
* The list of markers present in the sample can be obtained with '-t marker_pres_table'
$ metaphlan2.py -t marker_pres_table metagenome_outfmt.bz2 --input_type bowtie2out -o marker_abundance_table.txt
The --pres_th argument (default 1.0) set the minimum RPK value to consider a marker present
* The list '-t clade_profiles' analysis type reports the same information of '-t marker_ab_table'
but the markers are reported on a clade-by-clade basis.
$ metaphlan2.py -t clade_profiles metagenome_outfmt.bz2 --input_type bowtie2out -o marker_abundance_table.txt
* Finally, to obtain all markers present for a specific clade and all its subclades, the
'-t clade_specific_strain_tracker' should be used. For example, the following command
is reporting the presence/absence of the markers for the B. fragulis species and its strains
the optional argument --min_ab specifies the minimum clade abundance for reporting the markers
$ metaphlan2.py -t clade_specific_strain_tracker --clade s__Bacteroides_fragilis metagenome_outfmt.bz2 --input_type bowtie2out -o marker_abundance_table.txt
-------------------------------------------------------------------
positional arguments:
INPUT_FILE the input file can be:
* a fastq file containing metagenomic reads
OR
* a BowTie2 produced SAM file.
OR
* an intermediary mapping file of the metagenome generated by a previous MetaPhlAn run
If the input file is missing, the script assumes that the input is provided using the standard
input, or named pipes.
IMPORTANT: the type of input needs to be specified with --input_type
OUTPUT_FILE the tab-separated output file of the predicted taxon relative abundances
[stdout if not present]
Required arguments:
--input_type {fastq,fasta,multifasta,multifastq,bowtie2out,sam}
set whether the input is the multifasta file of metagenomic reads or
the SAM file of the mapping of the reads against the MetaPhlAn db.
[default 'automatic', i.e. the script will try to guess the input format]
Mapping arguments:
--mpa_pkl MPA_PKL The metadata pickled MetaPhlAn file [deprecated]
--force
--bowtie2db METAPHLAN_BOWTIE2_DB
The BowTie2 database file of the MetaPhlAn database. Used if --input_type is fastq, fasta, multifasta, or multifastq [default /root/.pyenv/versions/miniconda2-4.0.5/bin/metaphlan_databases]
-x INDEX, --index INDEX
Specify the id of the database version to use. If the database
files are not found on the local MetaPhlAn2 installation they
will be automatically downloaded [default latest]
--bt2_ps BowTie2 presets
Presets options for BowTie2 (applied only when a multifasta file is provided)
The choices enabled in MetaPhlAn are:
* sensitive
* very-sensitive
* sensitive-local
* very-sensitive-local
[default very-sensitive]
--bowtie2_exe BOWTIE2_EXE
Full path and name of the BowTie2 executable. This option allowsMetaPhlAn to reach the executable even when it is not in the system PATH or the system PATH is unreachable
--bowtie2_build BOWTIE2_BUILD
Full path to the bowtie2-build command to use, deafult assumes that 'bowtie2-build is present in the system path
--bowtie2out FILE_NAME
The file for saving the output of BowTie2
--no_map Avoid storing the --bowtie2out map file
--tmp_dir The folder used to store temporary files [default is the OS dependent tmp dir]
Post-mapping arguments:
--tax_lev TAXONOMIC_LEVEL
The taxonomic level for the relative abundance output:
'a' : all taxonomic levels
'k' : kingdoms
'p' : phyla only
'c' : classes only
'o' : orders only
'f' : families only
'g' : genera only
's' : species only
[default 'a']
--min_cu_len minimum total nucleotide length for the markers in a clade for
estimating the abundance without considering sub-clade abundances
[default 2000]
--min_alignment_len The sam records for aligned reads with the longest subalignment
length smaller than this threshold will be discarded.
[default None]
--ignore_eukaryotes Do not profile eukaryotic organisms
--ignore_bacteria Do not profile bacterial organisms
--ignore_archaea Do not profile archeal organisms
--stat_q Quantile value for the robust average
[default 0.1]
--perc_nonzero Percentage of markers with a non zero relative abundance for misidentify a species
[default 0.33]
--ignore_markers IGNORE_MARKERS
File containing a list of markers to ignore.
--avoid_disqm Deactivate the procedure of disambiguating the quasi-markers based on the
marker abundance pattern found in the sample. It is generally recommended
to keep the disambiguation procedure in order to minimize false positives
--stat EXPERIMENTAL! Statistical approach for converting marker abundances into clade abundances
'avg_g' : clade global (i.e. normalizing all markers together) average
'avg_l' : average of length-normalized marker counts
'tavg_g' : truncated clade global average at --stat_q quantile
'tavg_l' : trunated average of length-normalized marker counts (at --stat_q)
'wavg_g' : winsorized clade global average (at --stat_q)
'wavg_l' : winsorized average of length-normalized marker counts (at --stat_q)
'med' : median of length-normalized marker counts
[default tavg_g]
Additional analysis types and arguments:
-t ANALYSIS TYPE Type of analysis to perform:
* rel_ab: profiling a metagenomes in terms of relative abundances
* rel_ab_w_read_stats: profiling a metagenomes in terms of relative abundances and estimate the number of reads comming from each clade.
* reads_map: mapping from reads to clades (only reads hitting a marker)
* clade_profiles: normalized marker counts for clades with at least a non-null marker
* marker_ab_table: normalized marker counts (only when > 0.0 and normalized by metagenome size if --nreads is specified)
* marker_counts: non-normalized marker counts [use with extreme caution]
* marker_pres_table: list of markers present in the sample (threshold at 1.0 if not differently specified with --pres_th
[default 'rel_ab']
--nreads NUMBER_OF_READS
The total number of reads in the original metagenome. It is used only when
-t marker_table is specified for normalizing the length-normalized counts
with the metagenome size as well. No normalization applied if --nreads is not
specified
--pres_th PRESENCE_THRESHOLD
Threshold for calling a marker present by the -t marker_pres_table option
--clade The clade for clade_specific_strain_tracker analysis
--min_ab The minimum percentage abundace for the clade in the clade_specific_strain_tracker analysis
Output arguments:
-o output file, --output_file output file
The output file (if not specified as positional argument)
--sample_id_key name Specify the sample ID key for this analysis. Defaults to '#SampleID'.
--sample_id value Specify the sample ID for this analysis. Defaults to 'Metaphlan2_Analysis'.
-s sam_output_file, --samout sam_output_file
The sam output file
--legacy-output Old two columns output
--no-unknown-estimation
Ignore estimation of reads mapping to unkwnown clades
--biom biom_output, --biom_output_file biom_output
If requesting biom file output: The name of the output file in biom format
--mdelim mdelim, --metadata_delimiter_char mdelim
Delimiter for bug metadata: - defaults to pipe. e.g. the pipe in k__Bacteria|p__Proteobacteria
Other arguments:
--nproc N The number of CPUs to use for parallelizing the mapping [default 4]
--install Only checks if the MetaPhlAn2 DB is installed and installs it if not. All other parameters are ignored.
--force_download Force the re-download of the latest MetaPhlAn2 database.
--read_min_len READ_MIN_LEN
Specify the minimum length of the reads to be considered when parsing the input file with 'read_fastx.py' script, default value is 70
-v, --version Prints the current MetaPhlAn version and exit
-h, --help show this help message and exit
>python2.7 hclust2/hclust2.py -h
$ python2.7 hclust2/hclust2.py -h
usage: hclust2.py [-h] [-i [INPUT_FILE]] [-o [OUTPUT_FILE]]
[--legend_file [LEGEND_FILE]] [-t INPUT_TYPE] [--sep SEP]
[--out_table OUT_TABLE] [--fname_row FNAME_ROW]
[--sname_row SNAME_ROW] [--metadata_rows METADATA_ROWS]
[--skip_rows SKIP_ROWS] [--sperc SPERC] [--fperc FPERC]
[--stop STOP] [--ftop FTOP] [--def_na DEF_NA]
[--f_dist_f F_DIST_F] [--s_dist_f S_DIST_F]
[--load_dist_matrix_f LOAD_DIST_MATRIX_F]
[--load_dist_matrix_s LOAD_DIST_MATRIX_S]
[--load_pickled_dist_matrix_f LOAD_PICKLED_DIST_MATRIX_F]
[--load_pickled_dist_matrix_s LOAD_PICKLED_DIST_MATRIX_S]
[--save_pickled_dist_matrix_f SAVE_PICKLED_DIST_MATRIX_F]
[--save_pickled_dist_matrix_s SAVE_PICKLED_DIST_MATRIX_S]
[--no_fclustering] [--no_plot_fclustering]
[--no_sclustering] [--no_plot_sclustering]
[--flinkage FLINKAGE] [--slinkage SLINKAGE] [--dpi DPI] [-l]
[--title TITLE] [--title_fontsize TITLE_FONTSIZE] [-s]
[--no_slabels] [--minv MINV] [--maxv MAXV] [--no_flabels]
[--max_slabel_len MAX_SLABEL_LEN]
[--max_flabel_len MAX_FLABEL_LEN]
[--flabel_size FLABEL_SIZE] [--slabel_size SLABEL_SIZE]
[--fdend_width FDEND_WIDTH] [--sdend_height SDEND_HEIGHT]
[--metadata_height METADATA_HEIGHT]
[--metadata_separation METADATA_SEPARATION]
[--colorbar_font_size COLORBAR_FONT_SIZE]
[--image_size IMAGE_SIZE]
[--cell_aspect_ratio CELL_ASPECT_RATIO]
[-c {Accent,Blues,BrBG,BuGn,BuPu,Dark2,GnBu,Greens,Greys,OrRd,Oranges,PRGn,Paired,Pastel1,Pastel2,PiYG,PuBu,PuBuGn,PuOr,PuRd,Purples,RdBu,RdGy,RdPu,RdYlBu,RdYlGn,Reds,Set1,Set2,Set3,Spectral,YlGn,YlGnBu,YlOrBr,YlOrRd,afmhot,autumn,binary,bone,brg,bwr,cool,copper,flag,gist_earth,gist_gray,gist_heat,gist_ncar,gist_rainbow,gist_stern,gist_yarg,gnuplot,gnuplot2,gray,hot,hsv,jet,ocean,pink,prism,rainbow,seismic,spectral,spring,summer,terrain,winter,bbcyr,bbcry,bcry}]
[--bottom_c BOTTOM_C] [--top_c TOP_C] [--nan_c NAN_C]
TBA
optional arguments:
-h, --help show this help message and exit
-i [INPUT_FILE], --inp [INPUT_FILE], --in [INPUT_FILE]
The input matrix
-o [OUTPUT_FILE], --out [OUTPUT_FILE]
The output image file [image on screen of not
specified]
--legend_file [LEGEND_FILE]
The output file for the legend of the provided
metadata
-t INPUT_TYPE, --input_type INPUT_TYPE
The input type can be a data matrix or distance matrix
[default data_matrix]
Input data matrix parameters:
--sep SEP
--out_table OUT_TABLE
Write processed data matrix to file
--fname_row FNAME_ROW
row number containing the names of the features
[default 0, specify -1 if no names are present in the
matrix
--sname_row SNAME_ROW
column number containing the names of the samples
[default 0, specify -1 if no names are present in the
matrix
--metadata_rows METADATA_ROWS
Row numbers to use as metadata[default None, meaning
no metadata
--skip_rows SKIP_ROWS
Row numbers to skip (0-indexed, comma separated) from
the input file[default None, meaning no rows skipped
--sperc SPERC Percentile of sample value distribution for sample
selection
--fperc FPERC Percentile of feature value distribution for sample
selection
--stop STOP Number of top samples to select (ordering based on
percentile specified by --sperc)
--ftop FTOP Number of top features to select (ordering based on
percentile specified by --fperc)
--def_na DEF_NA Set the default value for missing values [default None
which means no replacement]
Distance parameters:
--f_dist_f F_DIST_F Distance function for features [default correlation]
--s_dist_f S_DIST_F Distance function for sample [default euclidean]
--load_dist_matrix_f LOAD_DIST_MATRIX_F
Load the distance matrix to be used for features
[default None].
--load_dist_matrix_s LOAD_DIST_MATRIX_S
Load the distance matrix to be used for samples
[default None].
--load_pickled_dist_matrix_f LOAD_PICKLED_DIST_MATRIX_F
Load the distance matrix to be used for features as
previously saved as pickle file using hclust2 itself
[default None].
--load_pickled_dist_matrix_s LOAD_PICKLED_DIST_MATRIX_S
Load the distance matrix to be used for samples as
previously saved as pickle file using hclust2 itself
[default None].
--save_pickled_dist_matrix_f SAVE_PICKLED_DIST_MATRIX_F
Save the distance matrix for features to file [default
None].
--save_pickled_dist_matrix_s SAVE_PICKLED_DIST_MATRIX_S
Save the distance matrix for samples to file [default
None].
Clustering parameters:
--no_fclustering avoid clustering features
--no_plot_fclustering
avoid plotting the feature dendrogram
--no_sclustering avoid clustering samples
--no_plot_sclustering
avoid plotting the sample dendrogram
--flinkage FLINKAGE Linkage method for feature clustering [default
average]
--slinkage SLINKAGE Linkage method for sample clustering [default average]
Heatmap options:
--dpi DPI Image resolution in dpi [default 150]
-l, --log_scale Log scale
--title TITLE Title of the plot
--title_fontsize TITLE_FONTSIZE
Font size of the title
-s, --sqrt_scale Square root scale
--no_slabels Do not show sample labels
--minv MINV Minimum value to display in the color map [default
None meaning automatic]
--maxv MAXV Maximum value to display in the color map [default
None meaning automatic]
--no_flabels Do not show feature labels
--max_slabel_len MAX_SLABEL_LEN
Max number of chars to report for sample labels
[default 15]
--max_flabel_len MAX_FLABEL_LEN
Max number of chars to report for feature labels
[default 15]
--flabel_size FLABEL_SIZE
Feature label font size [default 10]
--slabel_size SLABEL_SIZE
Sample label font size [default 10]
--fdend_width FDEND_WIDTH
Width of the feature dendrogram [default 1 meaning
100% of default heatmap width]
--sdend_height SDEND_HEIGHT
Height of the sample dendrogram [default 1 meaning
100% of default heatmap height]
--metadata_height METADATA_HEIGHT
Height of the metadata panel [default 0.05 meaning 5%
of default heatmap height]
--metadata_separation METADATA_SEPARATION
Distance between the metadata and data panels.
[default 0.001 meaning 0.1% of default heatmap height]
--colorbar_font_size COLORBAR_FONT_SIZE
Color bar label font size [default 12]
--image_size IMAGE_SIZE
Size of the largest between width and eight size for
the image in inches [default 8]
--cell_aspect_ratio CELL_ASPECT_RATIO
Aspect ratio between width and height for the cells of
the heatmap [default 1.0]
-c {Accent,Blues,BrBG,BuGn,BuPu,Dark2,GnBu,Greens,Greys,OrRd,Oranges,PRGn,Paired,Pastel1,Pastel2,PiYG,PuBu,PuBuGn,PuOr,PuRd,Purples,RdBu,RdGy,RdPu,RdYlBu,RdYlGn,Reds,Set1,Set2,Set3,Spectral,YlGn,YlGnBu,YlOrBr,YlOrRd,afmhot,autumn,binary,bone,brg,bwr,cool,copper,flag,gist_earth,gist_gray,gist_heat,gist_ncar,gist_rainbow,gist_stern,gist_yarg,gnuplot,gnuplot2,gray,hot,hsv,jet,ocean,pink,prism,rainbow,seismic,spectral,spring,summer,terrain,winter,bbcyr,bbcry,bcry}, --colormap {Accent,Blues,BrBG,BuGn,BuPu,Dark2,GnBu,Greens,Greys,OrRd,Oranges,PRGn,Paired,Pastel1,Pastel2,PiYG,PuBu,PuBuGn,PuOr,PuRd,Purples,RdBu,RdGy,RdPu,RdYlBu,RdYlGn,Reds,Set1,Set2,Set3,Spectral,YlGn,YlGnBu,YlOrBr,YlOrRd,afmhot,autumn,binary,bone,brg,bwr,cool,copper,flag,gist_earth,gist_gray,gist_heat,gist_ncar,gist_rainbow,gist_stern,gist_yarg,gnuplot,gnuplot2,gray,hot,hsv,jet,ocean,pink,prism,rainbow,seismic,spectral,spring,summer,terrain,winter,bbcyr,bbcry,bcry}
--bottom_c BOTTOM_C Color to use for cells below the minimum value of the
scale [default None meaning bottom color of the scale]
--top_c TOP_C Color to use for cells below the maximum value of the
scale [default None meaning bottom color of the scale]
--nan_c NAN_C Color to use for nan cells [default None]
hclust2
>python2.7 hclust2/hclust2.py -h
$ python2.7 hclust2/hclust2.py -h
usage: hclust2.py [-h] [-i [INPUT_FILE]] [-o [OUTPUT_FILE]]
[--legend_file [LEGEND_FILE]] [-t INPUT_TYPE] [--sep SEP]
[--out_table OUT_TABLE] [--fname_row FNAME_ROW]
[--sname_row SNAME_ROW] [--metadata_rows METADATA_ROWS]
[--skip_rows SKIP_ROWS] [--sperc SPERC] [--fperc FPERC]
[--stop STOP] [--ftop FTOP] [--def_na DEF_NA]
[--f_dist_f F_DIST_F] [--s_dist_f S_DIST_F]
[--load_dist_matrix_f LOAD_DIST_MATRIX_F]
[--load_dist_matrix_s LOAD_DIST_MATRIX_S]
[--load_pickled_dist_matrix_f LOAD_PICKLED_DIST_MATRIX_F]
[--load_pickled_dist_matrix_s LOAD_PICKLED_DIST_MATRIX_S]
[--save_pickled_dist_matrix_f SAVE_PICKLED_DIST_MATRIX_F]
[--save_pickled_dist_matrix_s SAVE_PICKLED_DIST_MATRIX_S]
[--no_fclustering] [--no_plot_fclustering]
[--no_sclustering] [--no_plot_sclustering]
[--flinkage FLINKAGE] [--slinkage SLINKAGE] [--dpi DPI] [-l]
[--title TITLE] [--title_fontsize TITLE_FONTSIZE] [-s]
[--no_slabels] [--minv MINV] [--maxv MAXV] [--no_flabels]
[--max_slabel_len MAX_SLABEL_LEN]
[--max_flabel_len MAX_FLABEL_LEN]
[--flabel_size FLABEL_SIZE] [--slabel_size SLABEL_SIZE]
[--fdend_width FDEND_WIDTH] [--sdend_height SDEND_HEIGHT]
[--metadata_height METADATA_HEIGHT]
[--metadata_separation METADATA_SEPARATION]
[--colorbar_font_size COLORBAR_FONT_SIZE]
[--image_size IMAGE_SIZE]
[--cell_aspect_ratio CELL_ASPECT_RATIO]
[-c {Accent,Blues,BrBG,BuGn,BuPu,Dark2,GnBu,Greens,Greys,OrRd,Oranges,PRGn,Paired,Pastel1,Pastel2,PiYG,PuBu,PuBuGn,PuOr,PuRd,Purples,RdBu,RdGy,RdPu,RdYlBu,RdYlGn,Reds,Set1,Set2,Set3,Spectral,YlGn,YlGnBu,YlOrBr,YlOrRd,afmhot,autumn,binary,bone,brg,bwr,cool,copper,flag,gist_earth,gist_gray,gist_heat,gist_ncar,gist_rainbow,gist_stern,gist_yarg,gnuplot,gnuplot2,gray,hot,hsv,jet,ocean,pink,prism,rainbow,seismic,spectral,spring,summer,terrain,winter,bbcyr,bbcry,bcry}]
[--bottom_c BOTTOM_C] [--top_c TOP_C] [--nan_c NAN_C]
TBA
optional arguments:
-h, --help show this help message and exit
-i [INPUT_FILE], --inp [INPUT_FILE], --in [INPUT_FILE]
The input matrix
-o [OUTPUT_FILE], --out [OUTPUT_FILE]
The output image file [image on screen of not
specified]
--legend_file [LEGEND_FILE]
The output file for the legend of the provided
metadata
-t INPUT_TYPE, --input_type INPUT_TYPE
The input type can be a data matrix or distance matrix
[default data_matrix]
Input data matrix parameters:
--sep SEP
--out_table OUT_TABLE
Write processed data matrix to file
--fname_row FNAME_ROW
row number containing the names of the features
[default 0, specify -1 if no names are present in the
matrix
--sname_row SNAME_ROW
column number containing the names of the samples
[default 0, specify -1 if no names are present in the
matrix
--metadata_rows METADATA_ROWS
Row numbers to use as metadata[default None, meaning
no metadata
--skip_rows SKIP_ROWS
Row numbers to skip (0-indexed, comma separated) from
the input file[default None, meaning no rows skipped
--sperc SPERC Percentile of sample value distribution for sample
selection
--fperc FPERC Percentile of feature value distribution for sample
selection
--stop STOP Number of top samples to select (ordering based on
percentile specified by --sperc)
--ftop FTOP Number of top features to select (ordering based on
percentile specified by --fperc)
--def_na DEF_NA Set the default value for missing values [default None
which means no replacement]
Distance parameters:
--f_dist_f F_DIST_F Distance function for features [default correlation]
--s_dist_f S_DIST_F Distance function for sample [default euclidean]
--load_dist_matrix_f LOAD_DIST_MATRIX_F
Load the distance matrix to be used for features
[default None].
--load_dist_matrix_s LOAD_DIST_MATRIX_S
Load the distance matrix to be used for samples
[default None].
--load_pickled_dist_matrix_f LOAD_PICKLED_DIST_MATRIX_F
Load the distance matrix to be used for features as
previously saved as pickle file using hclust2 itself
[default None].
--load_pickled_dist_matrix_s LOAD_PICKLED_DIST_MATRIX_S
Load the distance matrix to be used for samples as
previously saved as pickle file using hclust2 itself
[default None].
--save_pickled_dist_matrix_f SAVE_PICKLED_DIST_MATRIX_F
Save the distance matrix for features to file [default
None].
--save_pickled_dist_matrix_s SAVE_PICKLED_DIST_MATRIX_S
Save the distance matrix for samples to file [default
None].
Clustering parameters:
--no_fclustering avoid clustering features
--no_plot_fclustering
avoid plotting the feature dendrogram
--no_sclustering avoid clustering samples
--no_plot_sclustering
avoid plotting the sample dendrogram
--flinkage FLINKAGE Linkage method for feature clustering [default
average]
--slinkage SLINKAGE Linkage method for sample clustering [default average]
Heatmap options:
--dpi DPI Image resolution in dpi [default 150]
-l, --log_scale Log scale
--title TITLE Title of the plot
--title_fontsize TITLE_FONTSIZE
Font size of the title
-s, --sqrt_scale Square root scale
--no_slabels Do not show sample labels
--minv MINV Minimum value to display in the color map [default
None meaning automatic]
--maxv MAXV Maximum value to display in the color map [default
None meaning automatic]
--no_flabels Do not show feature labels
--max_slabel_len MAX_SLABEL_LEN
Max number of chars to report for sample labels
[default 15]
--max_flabel_len MAX_FLABEL_LEN
Max number of chars to report for feature labels
[default 15]
--flabel_size FLABEL_SIZE
Feature label font size [default 10]
--slabel_size SLABEL_SIZE
Sample label font size [default 10]
--fdend_width FDEND_WIDTH
Width of the feature dendrogram [default 1 meaning
100% of default heatmap width]
--sdend_height SDEND_HEIGHT
Height of the sample dendrogram [default 1 meaning
100% of default heatmap height]
--metadata_height METADATA_HEIGHT
Height of the metadata panel [default 0.05 meaning 5%
of default heatmap height]
--metadata_separation METADATA_SEPARATION
Distance between the metadata and data panels.
[default 0.001 meaning 0.1% of default heatmap height]
--colorbar_font_size COLORBAR_FONT_SIZE
Color bar label font size [default 12]
--image_size IMAGE_SIZE
Size of the largest between width and eight size for
the image in inches [default 8]
--cell_aspect_ratio CELL_ASPECT_RATIO
Aspect ratio between width and height for the cells of
the heatmap [default 1.0]
-c {Accent,Blues,BrBG,BuGn,BuPu,Dark2,GnBu,Greens,Greys,OrRd,Oranges,PRGn,Paired,Pastel1,Pastel2,PiYG,PuBu,PuBuGn,PuOr,PuRd,Purples,RdBu,RdGy,RdPu,RdYlBu,RdYlGn,Reds,Set1,Set2,Set3,Spectral,YlGn,YlGnBu,YlOrBr,YlOrRd,afmhot,autumn,binary,bone,brg,bwr,cool,copper,flag,gist_earth,gist_gray,gist_heat,gist_ncar,gist_rainbow,gist_stern,gist_yarg,gnuplot,gnuplot2,gray,hot,hsv,jet,ocean,pink,prism,rainbow,seismic,spectral,spring,summer,terrain,winter,bbcyr,bbcry,bcry}, --colormap {Accent,Blues,BrBG,BuGn,BuPu,Dark2,GnBu,Greens,Greys,OrRd,Oranges,PRGn,Paired,Pastel1,Pastel2,PiYG,PuBu,PuBuGn,PuOr,PuRd,Purples,RdBu,RdGy,RdPu,RdYlBu,RdYlGn,Reds,Set1,Set2,Set3,Spectral,YlGn,YlGnBu,YlOrBr,YlOrRd,afmhot,autumn,binary,bone,brg,bwr,cool,copper,flag,gist_earth,gist_gray,gist_heat,gist_ncar,gist_rainbow,gist_stern,gist_yarg,gnuplot,gnuplot2,gray,hot,hsv,jet,ocean,pink,prism,rainbow,seismic,spectral,spring,summer,terrain,winter,bbcyr,bbcry,bcry}
--bottom_c BOTTOM_C Color to use for cells below the minimum value of the
scale [default None meaning bottom color of the scale]
--top_c TOP_C Color to use for cells below the maximum value of the
scale [default None meaning bottom color of the scale]
--nan_c NAN_C Color to use for nan cells [default None]
GraPhlAn
> graphlan.py -h
# graphlan.py -h
usage: graphlan.py [-h] [--format ['output_image_format']]
[--warnings WARNINGS] [--positions POSITIONS]
[--dpi image_dpi] [--size image_size] [--pad pad_in]
[--external_legends] [--avoid_reordering] [-v]
input_tree output_image
GraPhlAn 1.1.3 (5 June 2018) AUTHORS: Nicola Segata (nsegata@hsph.harvard.edu)
positional arguments:
input_tree the input tree in PhyloXML format
output_image the output image, the format is guessed from the
extension unless --format is given. Available file
formats are: png, pdf, ps, eps, svg
optional arguments:
-h, --help show this help message and exit
--format ['output_image_format']
set the format of the output image (default none
meaning that the format is guessed from the output
file extension)
--warnings WARNINGS set whether warning messages should be reported or not
(default 1)
--positions POSITIONS
set whether the absolute position of the points should
be reported on the standard output. The two
cohordinates are r and theta
--dpi image_dpi the dpi of the output image for non vectorial formats
--size image_size the size of the output image (in inches, default 7.0)
--pad pad_in the distance between the most external graphical
element and the border of the image
--external_legends specify whether the two external legends should be put
in separate file or keep them along with the image
(default behavior)
--avoid_reordering specify whether the tree will be reorder or not
(default the tree will be reordered)
-v, --version Prints the current GraPhlAn version and exit
> python2.7 graphlan/graphlan_annotate.py -h
# python2.7 graphlan/graphlan_annotate.py -h
usage: graphlan_annotate.py [-h] [--annot annotation_file] [-v]
input_tree [output_tree]
GraPhlAn annotate module 1.1.3 (5 June 2018) AUTHORS: Nicola Segata
(nsegata@hsph.harvard.edu)
positional arguments:
input_tree the input tree in Newick, Nexus, PhyloXML or plain
text format
output_tree the output tree in PhyloXML format containing the
newly added annotations. If not specified, the input
tree file will be overwritten
optional arguments:
-h, --help show this help message and exit
--annot annotation_file
specify the annotation file
-v, --version Prints the current GraPhlAn version and exit
入力ファイル
fasta、fastq、tar.bz2、gzなど。
実行方法
テストデータダウンロード。ここでは1MB程度のgzがダウンロードされる。
curl -O https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014459-Stool.fasta.gz
curl -O https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014464-Anterior_nares.fasta.gz
curl -O https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014470-Tongue_dorsum.fasta.gz
curl -O https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014472-Buccal_mucosa.fasta.gz
curl -O https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014476-Supragingival_plaque.fasta.gz
curl -O https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014494-Posterior_fornix.fasta.gz
ダウンロードしたデータを移動させる。
mkdir metaphlan2_analysis #新しくディレクトリを作る。
mv *fasta.gz metaphlan2_analysis/ #ダウンロードしたファイルを移動させる
cd metaphlan2_analysis/ #移動
fastaデータを1つだけ解析するには以下のように打つ。
metaphlan2.py SRS014476-Supragingival_plaque.fasta.gz --input_type fasta --nproc 4 > SRS014476-Supragingival_plaque_profile.txt
終わると2つのファイルが出来る。
- SRS014476-Supragingival_plaque.fasta.gz.bowtie2out.txt 1行1リード形式でマーカー遺伝子とのマッチング結果がまとめられている。中間ファイルだが、どのような生物のマーカー遺伝子とhitしたのか調べる時に使える。
- SRS014476-Supragingival_plaque_profile.txt 解析結果。同定できたtaxisonomyがプリントされている。
中身を確認する。
今回は4データある。それぞれランする。ここではスレッドを12指定。fastaはgzip圧縮して拡張子をfasta.gzにする。
metaphlan2.py SRS014464-Anterior_nares.fasta.gz --input_type fasta --nproc 12 > SRS014464-Anterior_nares_profile.txt
metaphlan2.py SRS014470-Tongue_dorsum.fasta.gz --input_type fasta --nproc 12 > SRS014470-Tongue_dorsum_profile.txt
metaphlan2.py SRS014472-Buccal_mucosa.fasta.gz --input_type fasta --nproc 12 > SRS014472-Buccal_mucosa_profile.txt
metaphlan2.py SRS014494-Posterior_fornix.fasta.gz --input_type fasta --nproc 12 > SRS014494-Posterior_fornix_profile.txt
公式サイトではforで回す例も記載されている。
4つの結果をマージする(*1)。
python2.7 metaphlan2/utils/merge_metaphlan_tables.py *_profile.txt > merged_abundance_table.txt
ここではワイルドカードを使っているが、順番にファイルを指定しても良い。処理は一瞬で終わる。
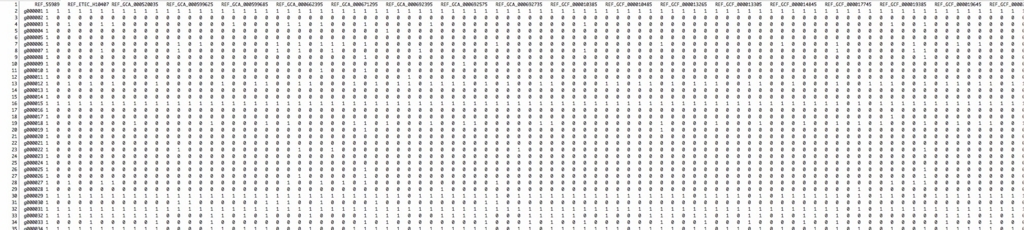
出力のmerged_abundance_table.txtを確認。
less -S merged_abundance_table.txt |head -10
ID SRS014459-Stool_profile SRS014464-Anterior_nares_profile SRS014470-Tongue_dorsum_profile SRS014472-Buccal_mucosa_profile SRS014476-Supragingival_plaque_profile SRS014494-Posterior_fornix_profile
#SampleID Metaphlan2_Analysis Metaphlan2_Analysis Metaphlan2_Analysis Metaphlan2_Analysis Metaphlan2_Analysis Metaphlan2_Analysis
k__Bacteria 100.0 16.82175 100.0 100.0 100.0 100.0
k__Bacteria|p__Actinobacteria 0.0 14.07759 0.85102 0.0 90.32561 0.0
k__Bacteria|p__Actinobacteria|c__Actinobacteria 0.0 14.07759 0.85102 0.0 90.32561 0.0
k__Bacteria|p__Actinobacteria|c__Actinobacteria|o__Actinomycetales 0.0 14.07759 0.85102 0.0 90.32561 0.0
k__Bacteria|p__Actinobacteria|c__Actinobacteria|o__Actinomycetales|f__Actinomycetaceae 0.0 0.0 0.85102 0.0 0.0 0.0
k__Bacteria|p__Actinobacteria|c__Actinobacteria|o__Actinomycetales|f__Actinomycetaceae|g__Actinomyces 0.0 0.0 0.85102 0.0 0.0 0.0
k__Bacteria|p__Actinobacteria|c__Actinobacteria|o__Actinomycetales|f__Actinomycetaceae|g__Actinomyces|s__Actinomyces_graevenitzii 0.0 0.0 0.85102 0.0 0.0 0.0
k__Bacteria|p__Actinobacteria|c__Actinobacteria|o__Actinomycetales|f__Actinomycetaceae|g__Actinomyces|s__Actinomyces_graevenitzii|t__Actinomyces_graevenitzii_unclassified 0.0 0.0 0.85102 0.0 0.0 0.0
species情報だけ抜き出す(この操作を行なわず上のファイルを直接hclustに使うと、階級を無視してクラスタリングを行ってしまうため、ここではspeciesレベルに階級を揃えている)。
grep -E "(s__)|(^ID)" merged_abundance_table.txt | grep -v "t__" | sed 's/^.*s__//g' > merged_abundance_table_species.txt
出力を確認。
head merged_abundance_table_species.txt
ID SRS014459-Stool_profile SRS014464-Anterior_nares_profile SRS014470-Tongue_dorsum_profile SRS014472-Buccal_mucosa_profile SRS014476-Supragingival_plaque_profile SRS014494-Posterior_fornix_profile
Actinomyces_graevenitzii 0.0 0.0 0.85102 0.0 0.0 0.0
Corynebacterium_matruchotii 0.0 0.0 0.0 0.0 58.21595 0.0
Corynebacterium_pseudodiphtheriticum 0.0 14.07759 0.0 0.0 0.0 0.0
Rothia_dentocariosa 0.0 0.0 0.0 0.0 32.10966 0.0
Bacteroides_cellulosilyticus 3.82206 0.0 0.0 0.0 0.0 0.0
Bacteroides_massiliensis 10.61295 0.0 0.0 0.0 0.0 0.0
Bacteroides_ovatus 4.08051 0.0 0.0 0.0 0.0 0.0
Bacteroides_stercoris 12.82765 0.0 0.0 0.0 0.0 0.0
Parabacteroides_distasonis 3.7393 0.0 0.0 0.0 0.0 0.0
hclust2を使いヒートマップを描く。ここではhclust2をダウンロードして、相対パスで実行している。
#hgコマンドがないなら sudo apt update && apt install mercurial
hg clone https://bitbucket.org/nsegata/hclust2
python2.7 hclust2/hclust2.py -i merged_abundance_table_species.txt \
-o abundance_heatmap_species.png \
--ftop 25 \
--f_dist_f braycurtis \
--s_dist_f braycurtis \
--cell_aspect_ratio 0.5 -l \
--flabel_size 6 \
--slabel_size 6 \
--max_flabel_len 100 \
--max_slabel_len 100 \
--minv 0.1 \
--dpi 300
hclust2はbrewで導入できる(今回はエラーになったため直接本体を叩いた)。

ヒートマップが描かれた。黒はその菌がゼロに近いことを意味し、赤-->黄色になると非常に多い(優占種である)ことを意味する。今回の6サンプルは、データによって菌種に共通するものはないことがわかる。ダウンサンプリングされたデータなので、レアなゲノムのデータが閾値以下のゼロに近い値になってしまったのかもしれない。また、データベースにな菌が多いデータでも、アサイン不可能なリードはたくさん出てくる(例えばrelative abundanceの99%が"unknown"など)。
サーバーで解析したなら、 図をscpでローカルに落として確認してください。
系統樹を描く。
GraPhlAnの入力ファイルを作る。
hg clone https://bitbucket.org/nsegata/graphlan
python2.7 graphlan/export2graphlan/export2graphlan.py \
--skip_rows 1,2 \
-i merged_abundance_table.txt \
--tree merged_abundance.tree.txt \
--annotation merged_abundance.annot.txt \
--most_abundant 100 \
--abundance_threshold 1 \
--least_biomarkers 10 \
--annotations 5,6 \
--external_annotations 7 \
--min_clade_size 1
graphlanもbrew/condaで導入できる(brew install biobakery/biobakery/graphlan )。ここではエラーが出たので、直接ダウンロードして使用している。
cladogramを作成。
#xmlファイル作成
python2.7 graphlan/graphlan_annotate.py \
--annot merged_abundance.annot.txt \
merged_abundance.tree.txt merged_abundance.xml
#xmlを使い系統樹描画
python2.7 graphlan/graphlan.py --dpi 300 \
merged_abundance.xml merged_abundance.png \
--external_legends

公式サイトでは他にもチュートリアルがある(リンク)。
引用
MetaPhlAn2 for enhanced metagenomic taxonomic profiling
Duy Tin Truong, Eric A Franzosa, Timothy L Tickle, Matthias Scholz, George Weingart, Edoardo Pasolli, Adrian Tett, Curtis Huttenhower & Nicola Segata.
Nature Methods 12, 902–903 (2015)
MetaPhlAn2 for enhanced metagenomic taxonomic profiling. - PubMed - NCBI
Compact graphical representation of phylogenetic data and metadata with GraPhlAn.
Asnicar F1, Weingart G2, Tickle TL3, Huttenhower C4, Segata N1.
PeerJ. 2015 Jun 18;3:e1029. doi: 10.7717/peerj.1029. eCollection 2015.
PanPhlAnは以前紹介しました。
追記
*1
condaで導入して使うと、helpメッセージは出るものの、マージができず空のファイルができてしまうエラがーがあった。hg loneして使用した。
hg clone https://bitbucket.org/biobakery/metaphlan2
python2.7 metaphlan2/utils/merge_metaphlan_tables.py *_profile.txt > merged_abundance_table.txt