ウェブの導入により、研究成果の伝統的な印刷出版物のソフトウェアベースの拡張が可能になった。a)研究論文をより簡単にコピー及び配布できるよりソフトウエアベースでpublishできる。 b)研究によって収集されたデータは、再利用および統合的で遡った(retrospective )分析のために、他の人がより容易に利用できるようにすることができる。 c)研究データを分析し視覚化するための方法とソフトウェアツールは、より強力かつアクセスしやすくなった。ウェブの導入によって促進されたこれらの発展は、生物医学研究の進歩にとって重要であったが、研究結果がどのようにデジタルで伝達されるかを改善するための多くの作業が残されている。ウェブの導入によりバイオインフォマティクス分野も促進されており、毎年何百もの新しいツールやデータベースが公開されている。同時に、biological sampleから大量の情報を抽出するhigh-content のバイオテクノロジーの進歩によるデータ収集の急速な成長は、この豊富なデータを管理するための集約型生物医学レポジトリの開発につながった。例えば、遺伝子発現トランスクリプトミクスとエピゲノミクスデータのためのOmnibus(GEO)(論文より ref.1)がある。実際、生物医学および生物学研究のすべての分野をカバーする多くのリポジトリが現在存在している。これらのリポジトリは、DataMed(ref.2)やFAIRSharing(ref.3)などのサイトで検索を行うよう索引付けされている。同様に、OMICtools(ref.4)などのバイオインフォマティクスツールを集約して索引付けするためのいくつかの取り組みが行われている。
集約型の生物医学データリポジトリのデータセットランディングページから開始してpublication-ready な図を生成したり、インタラクティブな視覚化グラフィックを生成する方法があるが、現在ほとんどの集約型生物医学リポジトリは、その中に含まれるデータセットを操作できるツールから切り離されている。バイオインフォマティクス分析のプロセスを合理化する努力がなされているが、最近まで、データセットを処理するために適用されたほとんどのバイオインフォマティクスツールは、分析を実行したユーザーには引き続き他のユーザーと共有できる永続URLを提供しない。しかし、特定の集約型データリポジトリに含まれるデータセットから生成され、特定のバイオインフォマティクスツールによって分析されるような、事前に生成された ‘缶詰型’ の分析結果を表示するウェブページは徐々に蓄積されている。 GeneMANIA(ref.5)、Enrichr(ref.6,7)、Clustergrammer(ref.8)、L1000CDS2(ref.9)などのソフトウェアツールは、分析のために遺伝子シグネチャーを提出するユーザー、または相互作用ネットワークを構築するユーザーに、これらのツール、他者と共有できる永続URLを提供する。これに加えて、Galaxy(ref.10)のようなプラットフォームは、Web上で共有できる複雑なバイオインフォマティクスワークフローやパイプラインを簡単に作成できるインターフェイスを提供する。同様に、JupyterやR Markdown(ref.11)のようなインタラクティブなノートブックは、バイオインフォマティクスの分析をウェブ上で体系的に共有するためのプラットフォームである。再現性のある生物医学的および生物学的研究結果を作成し、伝達し、共有するこれらの新しい永続的方法は、我々が「缶詰め分析( ‘canned analysis’)」と呼ぶ新しい形の出版物と考えることができる。
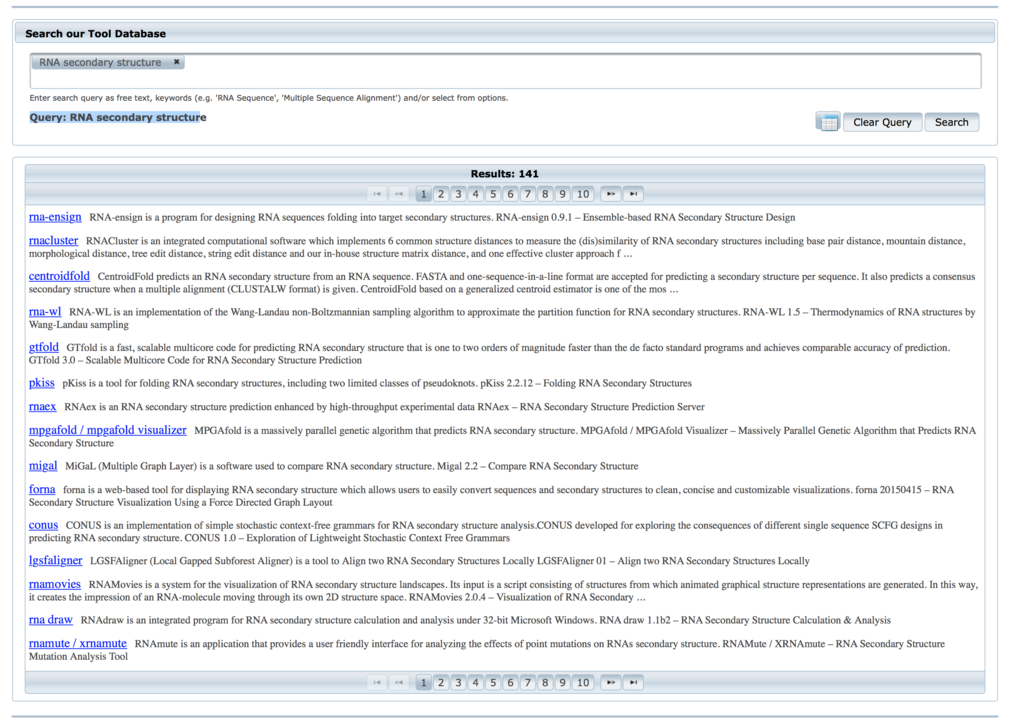
ここでは、初期状態で31,473のcanned analysisを揃えたcanned analysisのバイオインフォマティクス分析プラットフォームDatasets2Toolsを紹介する。31,473のcanned analysisは、選択されたツールを使用して6,431のデータセットに適用され、永続的なcanned analysisのURLが提供される。Datasets2Toolsは、4つのリーディングバイオインフォマティクスジャーナルBMC Bioinformatics、Bioinformatics、Database、そして Nucleic Acids Researchに掲載されている4901のソフトウェアツールのインデックスも作成している。Datasets2Toolsを使用すると、関連するデータセット、ツール、およびcanned analysisのデジタルオブジェクトをすばやく見つけることができる。著者らは、Datasets2Toolsがコミュニティへの貢献を通じて拡大することを期待している。 Datasets2Toolsは、Google Chromeブラウザの拡張機能とApplication Programming Interface(API)としても提供されている(リンク)。
Datasets2Toolsのユニークな機能の1つは、検索でき、アクセスでき、相互運用でき、再利用可能な(FAIR)原則に準拠する、データセット、ツール、およびcanned analysisを評価する機能である(ref.12)。 DataSet2Toolsに含まれる3種類のデジタルオブジェクト(データセット、ツール、およびcanned analysis)の検出可能性、アクセシビリティ、相互運用性、および再利用性に関するさまざまな側面に関する9つのYesまたはNoの質問に答えることができる。評価はデータベースに保存され、各データセット、ツール、または缶詰分析の近くに記章として表示される。 Datasets2Tools内の索引付けされたすべてのバイオインフォマティクスツールは出版物に由来しているため、Altmetric Attention ScoresおよびPlumXアセスメントならびに各ツールのPubMed引用はツールのカードの近くに表示される。このツール・メトリックの可視化は、ユーザーがバイオインフォマティクス・ツールをよりよく識別し、ランク付けするのに役立つ。 Datasets2Toolsのコンポーネントと機能の概要を論文図1に示す(pubmed)。
ヘルプページ
http://amp.pharm.mssm.edu/datasets2tools/help
概要
Datasets2Toolsは 30,000以上のバイオインフォマティクス解析と、6,000以上のデータセット、4,000以上の計算ツールの索引付けを行っているデジタルオブジェクトの発見と評価のためのプラットフォーム。 これらには、豊富なエンリッチメント分析、遺伝子相互作用ネットワーク、インタラクティブなデータの可視化、 マイクロアレイ、RNA-seq、プロテオミクスなどのデータセットの計算ツールを提供している。 ユーザーは、webページのインタラクティブな検索インターフェイスを使用してこれらのオブジェクトを検索できる。缶詰分析というのは、バイオインフォマティクス分析の結果を、見つけやすく、アクセス可能で、相互運用可能で、再利用可能な方法で保存するように設計された新しいタイプのデジタルオブジェクトと定義されている(詳細な定義は論文とhelp参照)。
使用方法
GEO accesion number(GEOはNGSやアレイのレポジトリで分析もできる)による検索。

ツール検索
http://amp.pharm.mssm.edu/datasets2tools/search?object_type=tool
例えば、NCBI GEOのこのデータ(ID: GSE39509)を調べたいとする(マウスのtumorのRNA seq)。
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE39509
IDで検索。

hitした。クリックする。

詳細が表示される。

Toolsタブではこのデータの分析に使われたツールが表示される。そのツールに飛んで、詳細を調べたり、他にこのツールが使われた解析にさらにリンクすることもできる。

Viewを押すと、GSE39509のデータから作られたヒートマップに飛ぶ。

マウスのホイールで拡大縮小できる。これだけでも論文よりずっと便利(論文のヒートマップは格好だけはいいが、拡大しても字が潰れて見えなかったり、データの価値が損なわれていることがある)。

左のメニューから、縦方向と列方向のソート方法を変えたり、ヒートマップの濃さを調整できる。また、ブラウザで表示できるSVG形式、または行列のテキストファイルとしてデータをダウンロードできる。
まずは興味あるキーワードでラフに検索してみて下さい。そこからさらに絞り込めます。

ユーザーがデータをアップロードすることも可能。
http://amp.pharm.mssm.edu/datasets2tools/contribute
chrome extensionとしても供給されている(ダイレクトリンクできる)。

おまけ
Ma'ayan LabのGithubレポジトリには他にもいくつかのツールが登録されている。そのうちの1つBioToolBayは、Datasets2Toolsと同じく、4つの主要バイオインフォマティクスジャーナルにアクセスされた論文のツールを検索できるHPらしい。
http://amp.pharm.mssm.edu/biotoolbay
ユニークリソース、可視化ツール、データベースなど、などで分類されている。
例えば可視化ツール一覧。まだ下にスクロールできる。

ツールをクリックすれば詳細を確認できる。

感じたこと
バイオインフォマティクスに限らないが、論文の図表で提示されたデータの多くは、通常、パースしたりフィルタリングされ、その上、場合によっては他のデータと比較可能なように正規化された、編集済みデータである(ただし生のデータが出される研究もたくさんある)。このようなデータセットは、著者らが適切に解釈できると判断した手順で処理されており、読み手が論文内で主張されていることを理解するには必要十分になっている。しかし、角度を変えて分析したい時には少し困る。このブログで書くと釈迦に説教になってしまうが、一般に、競合する研究者やその研究成果を基礎にしてより新しい研究を行いたい研究者がデータドリブンで新しい現象を見つけ出そうとする時、往往にして、false positiveのリスクを取ってでも最大限の情報を抽出したいと思うものである(特にomic関係でよくある。統計のcut-off valueは重要な役目を持つが、値自体に意味は薄いので、cut-offの向こうには大事な遺伝子が潜んでいる可能性がある)。具体的には、論文とともに公共レポジトリにdepositされた生データをダウンロードし(しばしば無い場合すらある)、寸暇を惜しんで自ら同じようなセットセットを作り上げたりする。これには、意外にバカにならないくらいの時間がかかったりする。しかし、たとえその論文のmethodsを注意深く読みながらデータの再解析を実行したとしても、環境の違いで再現できないことがしばしばある(理由は無数に考えられる)。バージョンを統一してもできない場合があり、それが意図的なものではないかと疑義が立つことすらあるが、それは別の問題なので、触れるだけにとどめる。
このような問題は、オリジナルデータは一切編集できないが、見かけ上だけフィルタリングやパース条件を変えられるインタラクティブなデータベースサイトに図表データがアップロードされていたり、depositされたデータを瞬時に可視化できるデータベースがあれば低減できる(NCBI GEO自体もそうです)。Datasets2Toolsは、公開されたデータを素早く、ユーザーが理解できる形で分析、再利用できることを目標に構築されている(FAIR Principles )。
注
紛らわしい文章を書きましたが、Datasets2ToolsはNCBI GEOにアクセッションされたデータから図を作っています。公開されているデータの中に、論文の著者らが直接アップしたものがどれだけあるのかは不明です。
引用
Datasets2Tools, repository and search engine for bioinformatics datasets, tools and canned analyses
Torre D, Krawczuk P, Jagodnik KM, Lachmann A, Wang Z, Wang L, Kuleshov MV, Ma'ayan A
Sci Data. 2018 Feb 27;5:180023.