随時更新
2019 1/23 リンク修正
2020 4/17 samtoolsについてmultiqcと連携する例を追記、4/18 help更新、インストール方法追加
2024/02/24 誤字修正 &インストールコマンド修正(conda => mamba )
samとbamのハンドリングに関するツールを紹介する。
追記
--2017--
- 8/20 samblaster samblasterでduplicationリードにタグをつける
- 8/29 BBTools 其の1、其の2
- 9/27 bamに塩基置換やindel変異を起こす bamに塩基置換やindel変異を起こすbamsurgeon
- 12/21 sambamba 高速なbam/samの解析ツール Sambamba
- 12.28 TopHatのunmapped.bamを修復する TopHat-Recondition
--2018--
- 1/19 Qualimap2 複数サンプルのマッピングを同時比較できるGUIツール Qualimap2
- 2/13 分析レポート出力 bamの分析に使うバイオインフォマティクスのツールキット goleft
- 2/21 各chromosomeのカバレッジ BBtoolsを使い各クロモソームのカバレッジを計算する
- 2/28 RNA seqのbam品質管理 RNA seqのクオリティチェックツール QoRTs
- 2/28 RNA seqのbam操作と品質管理 RNA seqのクオリティコントロールを行う RSeQC
- 3/9 bam操作 NGSUtilsその1 bamutils
- 3/31 SAMTools互換のsam高速処理 SAMTools互換で高速なsam,bam,cram処理ツールelprep
- 4/20 ファイルの単純分割 samやfastqの単純分割
- 4/28 構造変化部位の可視化 構造変化が起きた部位のマッピング状況を出力
- 5/16 realignmnet コード領域のリアライメントによってコールを改善ABRA
- 5/25 bamからprimerトリム bamのプライマートリミング BAMClipper
- 5/27 bamのプロセシング 高速なbam処理ツール biobambam2
- 6/6 bamの領域ごとのカバレッジ WGS、WESのカバレッジを素早く計算する mosdepth
- 6/6 bamをVCFコール周辺に限定 BAMを感心対象のみにするVariantBam
- 6/9 マルチマッピング補正 マルチマッピングを補正する MMR
- 6/20 bamの分析 bamの分析ツール Alfred
- 6/22 sam/bamの分析 シンプルなfastq、sam、bamの分析ツール fastqp
- 6/27 同じサンプルか判定 同じサンプルに由来するかvariant callingから判定する BAM-matcher
- 6/28 bam/samのカバレッジ bam/samのカバレッジなどを計算する pysamstats
- 6/29 bam/fastqの管理ツール bam, fastqのユーティリティツール EA-Utils
- 7/5 bam/samのカバレッジ 詳細なリードカウント情報を出力する bam-readcount
- 7/6 bamのvisualization statistics bamの可視化分析ツール bam.iobio.io
- 7/7 Picard Toolsのbam分析コマンドを自動実行 bamを分析し結果を統合する picardmetrics
- 8/1 bamとfastqが同一サンプルかチェック bamとfastqの整合性を確認する BamHash
- 8/4 sam/bamがmalformedではないか分析 PicardのValidateSamFile
- 9/16 BAMを複雑な条件でフィルタリング BAMを様々な条件でフィルタリングできる BAMQL
- 11/3 シーケンシングデータのハプロタイプを可視化 リードを分類する HapFlow
- 12/21 bamをクリーニング gencore
2019
- 1/14 リードの抽出とリアライメント リアライメントを素早く実行する Bazam
- 4/15 bamのデプスをコンソールで表示 bamのカバレッジを素早く確認bamcov
- 5/8 コンセンサス配列call deep bamからコンセンサス出力ConsensusFixer
- 6/16 bamを扱うcライブラリ bamM bamファイルを扱う bamM
- 8/6 fasta/fastq/bamのユーティリティツール fxtools
2020
- coverageトラックの視覚化 カバレッジトラックを視覚化する SparK
- bamを操作する包括的なツールキット(下でも紹介) BamDeal
2021
- バリアント領域のアラインメント画像を自動作成(たくさんの領域の自動処理にも対応) BamSnap
- ポジションに沿ってカバレッジプロットを生成 tinycov
-
ホストゲノムにunmapのリードを回収 Bowtie 2を使って素早くホスト由来リード除去
2022
- 高効率なカバレッジ計算ツール BamToCov
- BAMカバレッジ、polymorphic サイト率など計算する CMSeq
- ゲノムのBAMファイルを転写産物の BAMに変換してsalmonで扱う mudskipper
- BAMインデックス(bam.bai)ファイルを細かく切り刻む chopBAI
- 効率的な圧縮器 Genozip TM
2023
- 包括的なフォーマットコンバーター Bioconvert
picard-tools
homebrewやbiocondaでインストールできる。
#homebrew
brew install picard-tools
#bioconda
mamba install -c bioconda picard -y
BamIndexStats リファレンスにアラインされたリードの数。
picard BamIndexStats I=output_sorted.bam > bam_index_stats.tsv
I=でbamを指定。
>のリダイレクトで出力ファイル名を指定。
クロモソームごとのアライメントされたリードの数を出力できる。
Lambda_NEB length= 48502 Aligned= 4613 Unaligned= 0
CollectAlignmentSummaryMetrics アライメントされたリードの平均サイズ。
picard CollectAlignmentSummaryMetrics I=R1R2_sorted.bam OUTPUT=aln_sum_metric.tsv
CollectInsertSizeMetrics インサートサイズの分析。
picard CollectInsertSizeMetrics INPUT=R1R2_sorted.bam OUTPUT=insert_size_metric.tsv H=insert_size_metric.pdf
図が出力される。

QualityScoreDistribution クオリティスコアの分析。
picard QualityScoreDistribution INPUT=R1R2_sorted.bam OUTPUT=quality_score_dist.tsv CHART_OUTPUT=quality_score_dist.pdf
図が出力される。
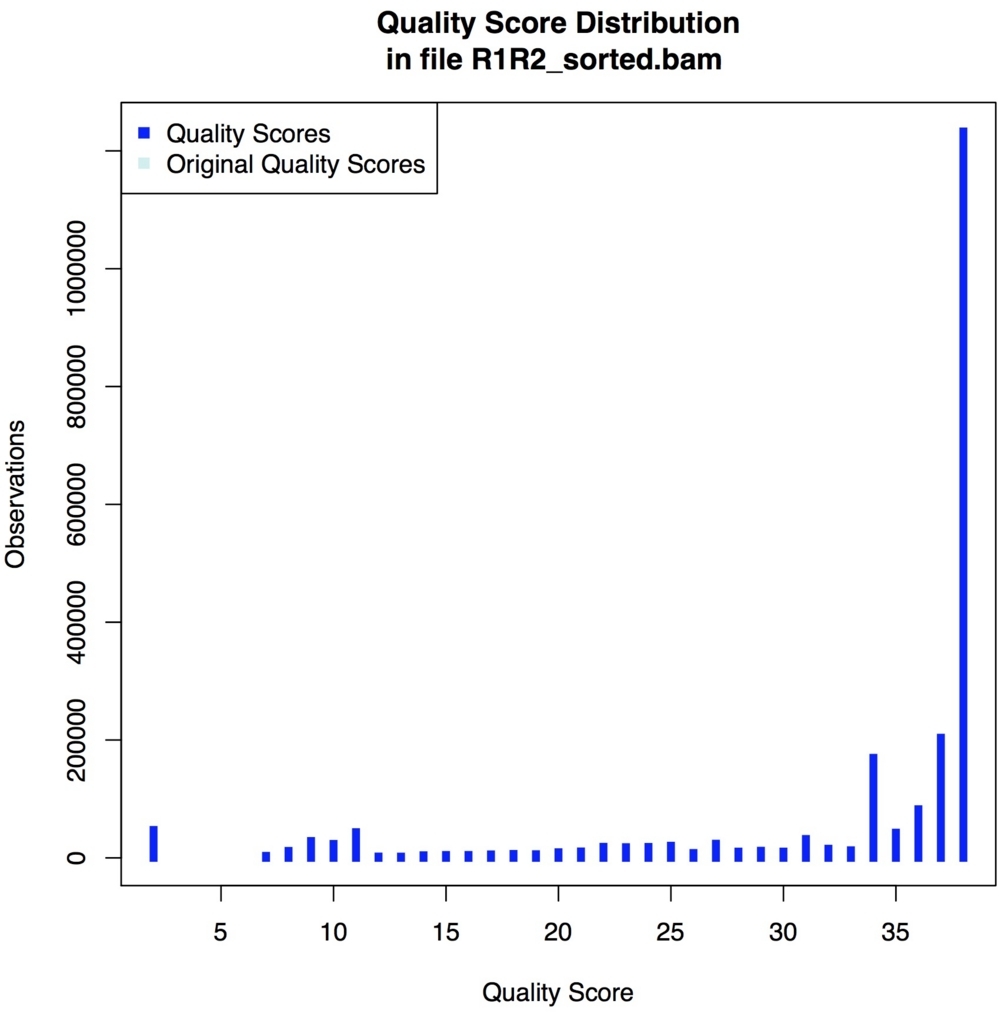
gatkのBQSR補正したbamを使った場合、補正前のスコアがOriginal quality scoreで表示され、補正前後のクオリティの変化を比較できる。
SamToFastq fastqの抽出
picard SamToFastq INPUT=R1R2_sorted.bam F=R1.fq F2=R2.fq
ペアリードをFとF2で指定する。シングルならF=で指定。 ペアリードの片方だけアンマップのペアや、セカンドアライメントのペア、ペアでないリードだけ返すようなオプションもある。https://gsl.hudsonalpha.org/information/software/bam2fastqにはunmapのリードを返せるとあるが、そのようなオプションはないように見える。
(下に載せた"bamtools split"でunmapped.bamを作り、それからリードを取り出すことでunmapリードを回収できる。)
samtools
インストール
#bioconda
mamba install -c bioconda -y samtools
#homebrew
brew install samtools
$ samtools
Program: samtools (Tools for alignments in the SAM format)
Version: 1.9 (using htslib 1.9)
Usage: samtools <command> [options]
Commands:
-- Indexing
dict create a sequence dictionary file
faidx index/extract FASTA
fqidx index/extract FASTQ
index index alignment
-- Editing
calmd recalculate MD/NM tags and '=' bases
fixmate fix mate information
reheader replace BAM header
targetcut cut fosmid regions (for fosmid pool only)
addreplacerg adds or replaces RG tags
markdup mark duplicates
-- File operations
collate shuffle and group alignments by name
cat concatenate BAMs
merge merge sorted alignments
mpileup multi-way pileup
sort sort alignment file
split splits a file by read group
quickcheck quickly check if SAM/BAM/CRAM file appears intact
fastq converts a BAM to a FASTQ
fasta converts a BAM to a FASTA
-- Statistics
bedcov read depth per BED region
depth compute the depth
flagstat simple stats
idxstats BAM index stats
phase phase heterozygotes
stats generate stats (former bamcheck)
-- Viewing
flags explain BAM flags
tview text alignment viewer
view SAM<->BAM<->CRAM conversion
depad convert padded BAM to unpadded BAM
flagstat - シンプルなアライメントレポートを出力
samtools flagstat -@ 4 input.bam
#multiqc(link)で統合レポート
samtools flagstat -@ 4 sample1.bam > sample1_flagstats
samtools flagstat -@ 4 sample2.bam > sample2_flagstats
samtools flagstat -@ 4 sample3.bam > sample3_flagstats
multiqc .
出力結果の例
3153604 + 0 in total (QC-passed reads + QC-failed reads)
0 + 0 secondary
0 + 0 supplementary
0 + 0 duplicates
18472 + 0 mapped (0.59% : N/A)
3153604 + 0 paired in sequencing
1576802 + 0 read1
1576802 + 0 read2
5708 + 0 properly paired (0.18% : N/A)
16576 + 0 with itself and mate mapped
1896 + 0 singletons (0.06% : N/A)
0 + 0 with mate mapped to a different chr
0 + 0 with mate mapped to a different chr (mapQ>=5)
properly pairedは、同じクロモソームに適切な向きと位置でアライメントされたペアリードの数になる。詳細に興味がある人はsamtoolsのマニュアルを参考にしてください。
http://www.htslib.org/doc/samtools.html
レポート結果が正しいかどうかは簡単なカウントで判断できる。
samtools view -@ 4 input.bam |wc #1行目と同じ数値になる。
stats -アライメントレポートを出力
samtools stats -@ 4 input.bam
#multiqc(link)で統合レポート
samtools stats -@ 4 sample1.bam > sample1_stats
samtools stats -@ 4 sample2.bam > sample2_stats
samtools stats -@ 4 sample3.bam > sample3_stats
multiqc .
idxstats - BAM indexからのレポート
samtools idxstats input.bam
#multiqc(link)で統合レポート
samtools idxstats sample1.bam > sample1_idxstats
samtools idxstats sample2.bam > sample2_idxstats
samtools idxstats sample3.bam > sample3_idxstats
multiqc .
depth - アライメントされたリードのデプスレポート
samtools depth input.bam > output
#zero coverage positionも含める
samtools depth -a input.bam > output
- -a output all positions (including zero depth)
- -a -a (or -aa) output absolutely all positions, including unused ref. sequences
- -l <int> read length threshold (ignore reads shorter than <int>) [0]
- -d/-m <int> maximum coverage depth [8000]. If 0, depth is set to the maximum
integer value, effectively removing any depth limit. - -q <int> base quality threshold [0]
- -Q <int> mapping quality threshold [0]
全ポジションのカバレッジを出力したい時に使える。depthコマンドを使えばpileupのカバレッジフィールドをパースする必要がない。
$ head -n 5 depth
chr 1 20
chr 2 20
chr 3 20
chr 4 22
chr 5 22
chr 6 22
chr 7 22
chr 8 22
chr 9 22
chr 10 23
merge - sam/bamのマージ
samtools merge -@ 8 output.bam input1.bam input2.bam input3.bam
2つ以上のbam、samを1つにまとめる。splitコマンドでクロモソームごとに分割し、別のクラスターでランして、後で統合するような用途にも使えそうである。
split - sam/bamの分割
samtools split
samtools fasta input.bam > input.fa
fastq - bam/samからfastqを抽出
#シングルエンド
samtools fastq -@ 8 single.bam > single.fq
#または
samtools fastq -@ 8 single.bam -s input.fq
#ペアエンド
samtools fastq -@ 8 pair.bam -1 pair_1.fq -2 pair_2.fq -n
- -1 write paired reads flagged READ1 to FILE
- -2 write paired reads flagged READ2 to FILE
- -0 write paired reads flagged both or neither READ1 and READ2 to
- -s write singleton reads to FILE [assume single-end]
- -n don't append /1 and /2 to the read name
- -N always append /1 and /2 to the read name
- FILE
ペアエンドの順番は考慮されない。ペアエンドの順番を維持したい場合は"bam bam2FastQ"コマンド(下の方で紹介)を使う。もしくは一度fastqを出力し、それからBBtoolsのrepairコマンドを使ってペアの同期を回復する。↓
ペアリードファイルの修復(BBTools)。
#pair_1.fq、pair_2.fqを修復
repair.sh -Xmx20g in1=pair_1.fq in2=pair_2.fq out1=fixed1.fq.gz out2=fixed2.fq.gz outs=singletons.fq repair
アンマップのリードだけSPAdesを使ってアセンブルする。
spades.py -1 fixed1.fq.gz -2 fixed2.fq.gz -s singletons.fq -o unmap-spades -k auto -t 8
=> blastnやblastxでどんな生物由来の配列かを推定する。
reheader - bamのヘッダーを変更
samtools reheader header.sam input.bam > output.bam
input.bamのヘッダーをheader.samのヘッダー情報で置換する。bam=>sam=>bamと変換するより圧倒的に早い。例えば以下のようにして使う。
#header情報だけsamに変換
samtools view -H input.bam > header.sam
# viなどでheader.bamを編集。例えばSO:unsortedをSO:sortedに修正。
# sample名をつけ忘れていたなら,@RGのSM:部分を編集する => SM:sample1
#元のbamのヘッダーを編集したheaderで置換。
samtools reheader header.sam input.bam > reheaer.bam
#headerが交換されたことを確認。
samtools view -H reheaer.bam |head
rmdup - PCR duplicateを除く。
samtools rmdup input.bam output.bam
ペアリードに使える。ペアリードがFRの向きで同じクロモソームの同じ位置にアライメントされているリードが2つ以上あると適用される。duplicate判定されるとマッピングクオリティがベストのものだけ残し、あとのペアは排除される。ペアの片方しかアライメントされていないものには適用されない。duplicateがあるかどうかは、fastqcでfastqを分析すれば調べられる。"-s"でシングルエンドに対応。
addreplacerg - リードグループ情報を追加したり、消したりする。
samtools addreplacerg
phase - heterozygous SNPsのコール。
samtools phase
sort - リードを 染色体のポジション順にソート(coordinate sort)。
samtools sort -@ 8 -m 2G pair.bam -o pair_sorted.bam
- -@ Set number of sorting and compression threads [1]
- -m Set maximum memory per thread; suffix K/M/G recognized [768M]
- -n Sort by read name
デフォルトではメモリーを768Mまでしか使わない。メモリーがもっとあるなら、-mで指定することで、特に大きなゲノムのソートが速くなる。上では2G/thread指定して、スレッド数を12にしている。入力はbamとsamに対応している。samを指定すると、ソートしたbamを出力してくれる。
"sam"からポジションソート済みbamを作成
samtools sort -@ 8 -m 8G -O BAM pair.sam -o pair_sorted.bam
- -O < FORMAT> Specify output format (SAM, BAM, CRAM)
view
bam <=> sam変換やフィルタリング
#samからbamに変換
samtools view -@ 10 -bS input.sam > output.bam
#bamからsamに変換
samtools view -@ 8 -h input.bam > output.sam
#bamの中身を見る。headerは見れない。
samtools view -@ 8 input.bam |less
#bamのheaderだけ見る。ソートされているか
samtools view -@ 8 -H input.bam |less
#50%ランダムサンプリング
samtools view -@ 8 -s 0.5 -b input.bam > random0.5.bam
#マッピングされたリードだけbamとして返す
samtools view -@ 8 -h -b -F 4 input.bam > mapped.bam
#マッピングされなかったリードだけbamとして返す
samtools view -@ 8 -h -b -f 4 input.bam > mapped.bam
#chr1とchr2のマッピングだけ取り出しbamとして返す
samtools view -@ 8 -b input.bam chr1 chr2 > chr1-2.bam
#chr1の100-300のマッピングだけ取り出しbamとして返す
samtools view -@ 8 -b input.bam chr1:100-300 > chr1_100-300.bam
#chr1の100-300のマッピングだけ取り出しペアエンドfastqとして返す
samtools view -b input.bam chr1:100-300 |samtools fastq -1 pair1.fq -2 pair2.fq -
#elprepには、マッピング時にbedで指定された領域のリードだけ残すか捨てるオプション"--filter-non-overlapping-reads bed-file"があります。exome解析などに使えるかと思います。
- -b output BAM
- -C output CRAM requires -T)
- -h include header in SAM output
- -S ignored (input format is auto-detected)
- -T Reference sequence FASTA FILE [null]
- -s FLOAT integer part sets seed of random number generator [0]; rest sets fraction of templates to subsample [no subsampling]
- -F INT only include reads with none of the bits set in INT set in FLAG [0]
- -f INT have all of the FLAGs present
- -@ Number of additional threads to use [0]
他にも以下のような使い方がある。
samからmappingされたリードだけbamに変換。
samtools view -@ 8 -bS -F 4 input.sam > output.bam
samからmappingされたリードだけbamに変換して、さらにfastq出力(unmapを除き、mapされたリードだけfastqで回収)。
samtools view -@ 8 -bS -F 4 input.sam |samtools fastq -@ 10 - > output.fq
bamからchr1にmappingされたリードだけ抽出して、さらにfastq出力。
samtools view -@ 8 -b input.bam chr1 > chr1.bam
samtools index chr1.bam
samtools fastq -@ 8 chr1.bam > chr1_mapped.fq
mapping quality (MAPQ) が1以上のリードだけ出力(mapping qualityについての情報: リンク)。
samtools view -@ 8 -bSq 1 input.sam > filtered.bam
マッピングされなかったリードとマルチマッピングのリードを除く。
samtools view -bS -F 0x904 input.sam > filtered.bam
index
bamファイルのindexing
samtools index sorted.bam
- -b Generate BAI-format index for BAM files [default]
- -c Generate CSI-format index for BAM files
quickcheck - 追記 bamがおかしくなっていないか、カレントの全てのbamを調べる。
samtools quickcheck -v *.bam > bad_bams.fofn && echo 'all ok' || echo 'some files failed check, see bad_bams.fofn'
bamがおかしくなっていないか(download中に切れたことを気づかないまま進めたり、変換中におちたbamを使ったりなど)。 "||"をつかうことで、エラーがある時だけ||以降が実行されるようにしている。
bamがcoordinateソート(chr順&ポジション順)されているかどうか。ソートされていれば、 SO:coordinateが表示される。
samtools view -H iput.bam | grep @HD
sample名取得。
samtools view -H iput.bam | grep '^@RG' | sed "s/.*SM:\([^\t]*\).*/\1/g" | uniq
GNU parallelsを使った複数のbamの並列処理。同時処理数を"-j"オプションで6つまでに制限している。samtools sortコマンド自体も4スレッドの並列処理をしている。
ls *.bam | parallel -j 6 "samtools sort -@ 4 {} > {.}_sorted.bam; samtools index {.}_sorted.bam"
splitした小さなbamなどを並列処理する時に一番高速化が期待できます。ただし、samtoolsのコマンドはI/O intensitiveなので、同時処理数が増えすぎるとかえって速度が遅くなったり、最悪、コンピュータがフリーズする恐れがあります。注意して使ってください。parallelのあとに"--dry-run"をつけると(parallel --dry-run)、コマンド内容は実行せず、実行コマンドだけ表示されます。まず--dry-runをつけて実行してください。時間短縮を考えるなら、elprep splitなどでbamを分割し、timeをつけて実行時間を測定し、最速の組み合わせを検討することをお勧めします。I/OやCPUなど環境の違いによって最速の組み合わせは変化するはずです。
minimap2でmapping、coordinate sortしたbamを出力。スレッドは12、メモリは2G / thread。リードグループ(@RG)のサンプル名はsample1。
minimap2 -R "@RG\tID:X\tLB:Y\tSM:sample1\tPL:ILLUMINA" -t 12 -ax sr \
ref.fa pair1.fq.gz pair2.fq.gz \
|samtools sort -@ 12 -m 2G -O BAM - > sorted.bam \
&& samtools index sorted.bam
minimap2でmapping、elprepでフィルタリングしてbam出力。elprep以外は上と同じだが、
minimap2 -ax sr -R "@RG\tID:X\tLB:Y\tSM:sample1\tPL:ILLUMINA" -t 12 \
-ax sr ref.fa pair1.fq.gz pair2.fq.gz \
|elprep filter /dev/stdin /dev/stdout \
--mark-duplicates --remove-duplicates --filter-mapping-quality 1 \
--clean-sam --nr-of-threads 12 \ --sorting-order coordinate \
|samtools sort -@ 12 -m 2G -O BAM - > sorted.bam \
&& samtools index sorted.bam
elprepの mark duplicatesとsortはin memoryで動作するため、メモリ要求量はデータサイズに直接比例します(samファイルサイズの10倍近くのメモリが必要)。メモリが足りなければ、--mark-duplicatesと--remove-duplicatesを外すか、samのsplitを行ってから順次フィルタリングするよう変更して下さい。
optional fieldのMD tagをつける。
samtools calmd -@ 12 -b input.bam ref.fasta > input.md.bam
samtools index input.md.bam
例えばchr2のギャップが検出された領域100-150のリードだけ抽出してfastaに変換、mafftでマルチプルアラインメントしてコンセンサス配列consensus.fastaを出力。mafft(リンク)とEMBOSSのcons(consensus call)を使う。
samtools view -@ 10 -b sort.bam chr2:100-150 \
|samtools fasta -@ 10 - > gapped.fasta && \
mafft --auto gapped.fasta > output.maf && \
cons output.maf consensus.fasta
#注意 *1
#小改良。karretでerror correctionしてからマルチプルアラインメント、最後にnucdiff(紹介)でSNV、indelをコールする(リファレンスはref.faと記載)
samtools view -@ 10 -b sort.bam chr2:100-150 \
|samtools fasta -@ 10 - > gapped.fasta && \
|karect -correct -threads=8 -matchtype=hamming -celltype=haploid -inputfile=gapped.fasta -kmer=9 &&
|mafft --auto gapped.fasta > output.maf && \
|cons output.maf consensus.fasta && \
|nucdiff --vcf yes ref.fa onsensus.fasta outdir unmatched
リードではなく、リファレンスゲノム配列の特定の染色体のポジションの配列だけ取り出す。
samtools
#chr2のリファレンス配列だけ取り出して保存する
samtools faidx genome.fa chr2 > chr2.fasta
#chr3の15000-20000のリファレンス配列だけ取り出して保存する
samtools faidx genome.fa chr3:15000-20000 > chr3_15000-20000.fasta
#数が多い時はリストファイルを指定する(テキストに1行に1つずつ指定)
samtools faidx genome.fa -r region_list > chr3_15000-20000.fasta
bamtools
bamファイルを使い色々なことができるツール。samtoolsの機能と重複するものが多いが、よりデータ解析よりに作られている。
インストール
#bioconda
mamba install -c bioconda bamtools -y
#homebrew
brew install bamtools
$ bamtools -h
usage: bamtools [--help] COMMAND [ARGS]
Available bamtools commands:
convert Converts between BAM and a number of other formats
count Prints number of alignments in BAM file(s)
coverage Prints coverage statistics from the input BAM file
filter Filters BAM file(s) by user-specified criteria
header Prints BAM header information
index Generates index for BAM file
merge Merge multiple BAM files into single file
random Select random alignments from existing BAM file(s), intended more as a testing tool.
resolve Resolves paired-end reads (marking the IsProperPair flag as needed)
revert Removes duplicate marks and restores original base qualities
sort Sorts the BAM file according to some criteria
split Splits a BAM file on user-specified property, creating a new BAM output file for each value found
stats Prints some basic statistics from input BAM file(s)
See 'bamtools help COMMAND' for more information on a specific command.
convert - bamtoolsの一番基本的な使い方はbamからのformat変換である。
bamtools convert -format <format> -in input.bam -out output
formatの部分に出力するフォーマットを記載する。pileup、bedのほかfastaやfastqに直接変換することも可能になっている(bedtoolsのbamtofastqと同じ)。ただしbamtoolsはシングルスレッドで動くので、samの出力はマルチスレッド化したsamtools viewが速いと思われる。
stats - bamの情報を出力できる。
bamtools stats -in input.bam
bamtools statsの出力
**********************************************
Stats for BAM file(s):
**********************************************
Total reads: 525231
Mapped reads: 524170 (99.798%)
Forward strand: 262861 (50.0467%)
Reverse strand: 262370 (49.9533%)
Failed QC: 0 (0%)
Duplicates: 0 (0%)
Paired-end reads: 525231 (100%)
'Proper-pairs': 517998 (98.6229%)
Both pairs mapped: 523577 (99.6851%)
Read 1: 262603
Read 2: 262628
Singletons: 593 (0.112903%)
samtoolsのflagstatの出力
525231 + 0 in total (QC-passed reads + QC-failed reads)
0 + 0 secondary
1683 + 0 supplementary
0 + 0 duplicates
524170 + 0 mapped (99.80% : N/A)
523548 + 0 paired in sequencing
261774 + 0 read1
261774 + 0 read2
517042 + 0 properly paired (98.76% : N/A)
521908 + 0 with itself and mate mapped
579 + 0 singletons (0.11% : N/A)
846 + 0 with mate mapped to a different chr
791 + 0 with mate mapped to a different chr (mapQ>=5)
大きな差はないが、個人的にはbamtools statの方が分かりやすい表記だと思う。
追記
#multiqcで統合
bamtools stats -in sample1.bam > sample1_bamtools
bamtools stats -in sample2.bam > sample2_bamtools
bamtools stats -in sample3.bam > sample3_bamtools
multiqc .
count - アライメントされたリード数のカウント。
bamtools count -in input.bam
sort - bamをname順に並べ替える。
bamtools sort -in input.bam -out input_sort.bam -byname
リード名順に並べ替えられる。samtools view -Hでヘッダーを確認すると
$ samtools view -H input_sort.bam |head 1
@HD VN:1.3 SO:queryname
になっており、名前順にソートされていることを確認できる。(-HはSAMheaderのみprintするsamtools viewのオプション)。samtools sort -nと同じと考えられる。
index - bamのindexを作る。
bamtools index -in input_sort.bam
samtools indexと同じである。~.baiが出力される。
coverage - アライメントされたリード数のカウント。
bamtools convert -in input.bam -out coverage.txt
version2.4.1ではなぜかsortしてないというエラーメッセージが出て検証できていない(bamtools sortとindexをかけていてもエラーになってしまう)。
filter - bamのフィルタリング。
bamtools filter -in input.bam -out output.bam -length 50
様々なパラメータでbamをフィルタリングできる。上記では50bpのリードのみ出力される。ただし~以上や~以下、また複数指定もできない(正直、使い勝手はイマイチである)。以下のようなフィルターがある。
- -alignmentFlag keep reads with this *exact* alignment flag (for more detailed queries, see below)
- -insertSize keep reads with insert size that matches pattern -length <int> keep reads with length that matches pattern
- -mapQuality keep reads with map quality that matches pattern -name <string> keep reads with name that matches pattern
- -queryBases keep reads with motif that matches pattern
- -tag keep reads with this key=>value pair
header - ヘッダー情報を出力。
bamtools header -in input.bam
bamのheaderを出力する。samtools view -Hと同じである。
merge - 複数のbamを1つにマージする。3つ以上も可。
bamtools merge -in input1.bam -in input2.bam -out merge.bam
random - bamを指定数ランダムサンプリングして出力。
bamtools random -in input1.bam -out output.bam -n 10000
この例だと10000リードランダム出力する。bamtools formatやsamtools view -hでsamに戻し、リード数をカウントしてチェックする (wc -l input.sam)。
split - ユーザー定義の情報に従いbamを分割する。
bamtools split -in input.bam -mapped
上記ではリファレンスにマッピングされたリード(> input.MAPPED.bam)、マッピングされていないリード(> input.UNMAPPED.bam)に分けて出力される。利用できる定義は
- -mapped split mapped/unmapped alignments
- -paired split single-end/paired-end alignments
- -reference split alignments by reference
- -tag <tag name> splits alignments based on all values of TAG encountered (i.e. -tag RG creates a BAM file for each read group in original BAM file)
クロモソームごとに分割するなら、 -referenceのフラグをつける。
bamtools split -in input.bam -reference
input_chrromosome1.bam、input_chromosome2.bam...unmapped.bamのようにリファレンスのFASTAのヘッダー名付のbamが出力される。
他にもいくつかの機能を持つ。bamtools <command> -hで各コマンドの全機能は確認できる。
elprep(紹介)
インストール
#bioconda
mamba install -c bioconda -y elprep
# elprep
elprep version 4.1.5 compiled with go1.12.7 - see http://github.com/exascience/elprep for more information.
2020/04/18 03:07:51 Incorrect number of parameters.
Print command details:
[--help]
[--help-extended]
Available commands: filter, sfm, vcf-to-elsites, bed-to-elsites, fasta-to-elfasta
filter/sfm parameters:
elprep [filter | sfm] sam-file sam-output-file
[--replace-reference-sequences sam-file]
[--filter-unmapped-reads]
[--filter-unmapped-reads-strict]
[--filter-mapping-quality mapping-quality]
[--filter-non-exact-mapping-reads]
[--filter-non-exact-mapping-reads-strict]
[--filter-non-overlapping-reads bed-file]
[--replace-read-group read-group-string]
[--mark-duplicates]
[--mark-optical-duplicates file]
[--optical-duplicates-pixel-distance nr]
[--remove-duplicates]
[--remove-optional-fields [all | list]]
[--keep-optional-fields [none | list]]
[--sorting-order [keep | unknown | unsorted | queryname | coordinate]]
[--clean-sam]
[--bqsr recal-file]
[--bqsr-reference elfasta]
[--quantize-levels nr]
[--sqq list]
[--max-cycle nr]
[--known-sites list]
[--nr-of-threads nr]
[--timed]
[--log-path path]
[--intermediate-files-output-prefix name] (sfm only)
[--intermediate-files-output-type [sam | bam]] (sfm only)
[--tmp-path path]
[--single-end] (sfm only)
[--contig-group-size nr] (sfm only)
vcf-to-elsites parameters:
elprep vcf-to-elsites vcf-file elsites-file
[--log-path path]
bed-to-elsites parameters:
elprep bed-to-elsites bed-file elsites-file
[--log-path path]
fasta-to-elfasta parameters:
elprep fasta-to-elfasta fasta-file elfasta-file
[--log-path path]
filter 以下の作業をワンライナーで行う。
- アンマップリードを完全に除く(--filter-unmapped-reads-strict)。
- duplicationのタグを付け(--mark-duplicates)duplicationリードを捨てる(--remove-duplicates)。
- mapping qualityがゼロのリード(bwaならmultiple mappingはゼロ)を捨てる(--filter-mapping-quality)
- samのcleaning(--clean-sam)。
elprep filter input.sam output.sam --mark-duplicates --remove-duplicates --filter-mapping-quality 0 --clean-sam
split インプット (sam/bam/cram)をクロモソームごとに分割する。unmapリードは別出力する。
elprep split input.sam output/ --output-prefix "split-sam" --output-type sam --nr-of-threads 8 --single-end
ソートやindexファイルがなくても動作する。シングルエンドのinputは--single-endをつける。デフォルトでは入力をペアエンドとして扱う。
merge splitで分割した複数のインプット (sam/bam/cram)をマージする。
elprep merge input/ output.sam --nr-of-threads 8
シングルエンドのinputは--single-endをつける。デフォルトでは入力をペアエンドとして扱う。bamとcram出力にも対応している。
VariantBam(紹介)
インストール
git clone --recursive https://github.com/jwalabroad/VariantBam.git
cd VariantBam
./configure
make
cd src/
#bioconda (link)
mamba install -c biocondavariantbam -y
> variant
# variant
Usage: variant <input.bam> [OPTIONS]
Description: Filter a BAM/SAM/CRAM/STDIN according to hierarchical rules
General options
-h, --help Display this help and exit
-v, --verbose Verbose output
-x, --no-output Don't output reads (used for profiling with -q)
-r, --rules JSON ecript for the rules.
-k, --proc-regions-file Samtools-style region string (e.g. 1:1,000-2,000) or BED/VCF of regions to process. -k UN iterates over unmapped-unmapped reads
-Q, --mark-as-qc-fail Flag reads that don't pass VariantBam with the failed QC flag, rather than deleting the read.
Output options
-o, --output Output file to write to (BAM/SAM/CRAM) file instead of stdout
-C, --cram Output file should be in CRAM format
-b, --bam Output should be in binary BAM format
-T, --reference Path to reference. Required for reading/writing CRAM
-s, --strip-tags Remove the specified tags, separated by commas. eg. -s RG,MD
-S, --strip-all-tags Remove all alignment tags
-Z, --write-trimmed Output the base-quality trimmed sequence rather than the original sequence. Also removes quality scores
Filtering options
-q, --qc-file Output a qc file that contains information about BAM
-m, --max-coverage Maximum coverage of output file. BAM must be sorted. Negative values enforce a minimum coverage
-p, --min-phred Set the minimum base quality score considered to be high-quality
Region specifiers
-g, --region Regions (e.g. myvcf.vcf or WG for whole genome) or newline seperated subsequence file.
-G, --exclude-region Same as -g, but for region where satisfying a rule EXCLUDES this read.
-l, --linked-region Same as -g, but turns on mate-linking
-L, --linked-exclude-region Same as -l, but for mate-linked region where satisfying this rule EXCLUDES this read.
-P, --region-pad Apply a padding to each region supplied with the region flags (specify after region flag)
Command line rules shortcuts (to be used without supplying a -r script)
--min-clip Minimum number of quality clipped bases
--max-nbases Maximum number of N bases
--min-mapq Minimum mapping quality
--min-del Minimum number of deleted bases
--min-ins Minimum number of inserted bases
--min-length Minimum read length (after base-quality trimming)
--motif Motif file
-R, --read-group Limit to just a single read group
-f, --include-aln-flag Flags to include (like samtools -f)
-F, --exclude-aln-flag Flags to exclude (like samtools -F)
chr1の1-1,100,000にマッピングされたリードのみbam出力する。
variant input.bam -g 'chr1:1-1,100,000' --min-mapq 10 -b -o mini.bam -v
sam出力なら-bを外す。mini.bamが出力される。
VCFでコールされたポジション周囲100-bpのリードのみbam出力する。
variant input.bam -l input.vcf -P 100 -b -o mini.bam -v
sub-samplingしてmax-coverageを100に減らす(マッパーによっては、リピート領域にMAPQ=0のリードが多数マッピングされる)。
variant input.bam -m 100 -o mini.bam -v -b
clippingされたハイクオリティなリードのみbam出力する。
variant input.bam --min-phred 4 --min-clip 5 -b -o mini.bam -v
goleft(紹介)
インストール
#bioconda
mamba install -c bioconda goleft -y
$ goleft
goleft Version: 0.2.0
covstats : coverage stats across bams by sampling
depth : parallelize calls to samtools in user-defined windows
depthwed : matricize output from depth to n-sites * n-samples
indexcov : quick coverage estimate using only the bam index
indexsplit : create regions of even coverage across bams/crams
samplename : report samplename(s) from a bam's SM tag
covstats bamをサンプリングしてカバレッジとインサートサイズをレポートする。
goleft covstats pair.bam
カバレッジ、インサートサイズとそのSDなどが出力される。

insert sizeは-> <-の内側サイズが計算される。250x2でinner inset sizeが100なら、outer inset sizeは600。
depth 一定のウィンドウサイズでbamのカバレッジを計算する。
goleft depth --reference ref.fa --prefix output input.bam
indexcov bam.baiからのカバレッジの超高速な推定。30サンプルを30秒で解析可能。複数のbamをディレクトリに準備してランする。
goleft indexcov --directory goleft_outdir/ *.bam
関係ないファイルがあるとエラーになるのでbamとbaiだけ集める。htmlで結果は出力される。ヒトゲノムでも早ければ1サンプル数秒で解析できる。
出力ディレクトリにできるindex.htmlが全サンプルのまとめ

multiqcでレポート作成 。
goleft indexcov --directory goleft_outdir *.bam
multiqc .
mosdepth(紹介)
インストール
#bioconda (link) (linux)
mamba create -n mosdepth -y
conda activate mosdepth
mamba install -c bioconda mosdepth -y
chr1のデプスを計算する。
mosdepth --by capture.bed output exome.bam -Q 0 -t 10 -c chr1
- -Q mapping quality threshold [default: 0]
- -t number of BAM decompression threads [default: 0]
- -c chromosome to restrict depth calculation.
multiqcで統合レポートを作成する。全スレッド使用。MQ≥1。
mosdepth --by gencode.v33.annotation.bed sample1.mosdepth sample1.bam -Q 1
mosdepth --by gencode.v33.annotation.bed sample2.mosdepth sample2.bam -Q 1
mosdepth --by gencode.v33.annotation.bed sample3.mosdepth sample3.bam -Q 1
multiqc .
biobambam2(紹介)
インストール
#bioconda (link)
mamba install -c bioconda biobambam -y
duplicateにタグをつけ、coordinateソートも行って出力する。
bamsormadup < input.bam level=1 threads=8 inputformat=bam SO=coordinate outputformat=bam indexfilename=output.bam.bai > output.bam
処理の大半が並列化されており、I/Oがリミットになりうるため、I/Oの高速なSSDなどのストレージ使用が推奨されている。threadsフラグがなければ利用できる全ての論理コアを使う。
ペアエンドを照合し、secondary hitは除く。samtools collateと同様の機能(samtools1.8 manual)。 level=0だと非圧縮になる。
bamcollate2 <input.bam collate=1 level=1 exclude=SECONDARY > out.bam
bamを再圧縮する。
bamrecompress <input.bam level=9 numthreads=8 > output.bam
ポジションソートしてbam出力する。
./bamsort SO=coordinate < input.bam inputthreads=8 level=1 outputformat=bam > out.bam
bamからペアエンドfastqを出力。
bamtofastq < out.bam F=1.fq F2=2.fq
Sambamba(紹介)
インストール
#bioconda
mamba install -c bioconda Sambamba -y
# sambamba -h
sambamba 0.6.6
Usage: sambamba [command] [args...]
Available commands: 'view', 'index', 'merge', 'sort',
'flagstat', 'slice', 'markdup', 'depth', 'mpileup'
To get help on a particular command, just call it without args.
Leave bug reports and feature requests at
https://github.com/lomereiter/sambamba/issues
view bamの基本情報
sambamba view --reference-info input.bam
- -I, --reference-info output to stdout only reference names and lengths in JSON
view samの基本情報
sambamba view -S --reference-info input.bam
- -S specify that input is in SAM format
bamのリファレンス"chr19"に適切にアライメントされるリードだけsam出力。(カウントなら-cをつけ、-o以下を消す)
sambamba view -F "proper_pair" input.bam chr19 -t 8 -f sam -o output.sam
- -o specify output filename
sort(リンク) ソート。
指定がなければindexが自動でつくcoordinate-sorted。
sambamba sort input.bam -o sorted.bam -t 20 -p -l 6
- -t use specified number of threads
- -p show progressbar in STDERR
- -l level of compression for sorted BAM, from 0 to 9(指定がなければ"6"くらいのサイズになる)
名前でソート。(indexは付けられない)
sambamba sort input.bam -o sorted.bam -t 20 -p -l 5
flagstat(リンク) bamの情報表示。
bamの情報表示。
sambamba flagstat input.bam -t 8 -p
sambamba flagstat input.bam
509280 + 0 in total (QC-passed reads + QC-failed reads)
0 + 0 secondary
538 + 0 supplementary
0 + 0 duplicates
456058 + 0 mapped (89.55%:N/A)
508742 + 0 paired in sequencing
254371 + 0 read1
254371 + 0 read2
451458 + 0 properly paired (88.74%:N/A)
454670 + 0 with itself and mate mapped
850 + 0 singletons (0.17%:N/A)
0 + 0 with mate mapped to a different chr
0 + 0 with mate mapped to a different chr (mapQ>=5)
表示された情報については上のflagstatのリンク先参照。bamのBGZFフォーマットしか対応していないのでsamはviewで変換してから使う。
index(リンク) bamのindexファイル作成
sambamba index input.bam -t 12 -p
merge(リンク) bamのマージ。
圧縮率最大の設定で、12スレッド使い3つのbamをマージ。
sambamba merge merge.bam input1.bam input2.bam nput3.bam -t 12 -p -l 9
- -l Specify compression level of output file as a number from 0 to 9
mpileup(リンク) samtoolsのmpileupのparallelバージョン。
20スレッド使い、pileupを実行。samtoolsの条件は--samtoolsをつけて書く。
sambamba mpileup input.bam -t 12 --samtools samtools -gu -S -D -d 1000 -L 1000 -m 3 -F 0.0002
mpileup出力 (manual) からリファレンスと異なる塩基の数を数える (引用)。pileup出力の5列目に全バリアント情報があるので、ここを取り出してパースする。
#5フィールド目を取り出して小文字を全て大文字に変換(tr)、1行1文字ずつにして(fold -w 1)ソートし(sort)、ユニークな文字数をカウント(uniq -c)
cut -f 5 sambamba-pileup_output | tr '[a-z]' '[A-Z]' | fold -w 1 | sort | uniq -c > counts
> cat counts
17026 !
947 "
178 #
2048447 $
78 %
78 &
122 '
117 (
101 )
4231 *
3801 +
157425826 ,
3737 -
157527525 .
203 /
80 0
6055 1
1157 2
330 3
249 4
123 5
90 6
250 7
88 8
99 9
118 :
109 ;
88 <
108 =
84 >
105 ?
88 @
95195 A
88 B
98141 C
82 D
90 E
70 F
97752 G
91 H
79 I
104 J
104 K
91 L
99 M
97 N
107 O
93 P
2117 Q
93 R
105 S
94768 T
94 U
296 V
1017 W
151 X
690 Y
2549 Z
231 [
990 \
2010030 ]
2041594 ^
5フィールド目は
- ^ この位置がリードによってカバーされる最初の位置である場合
- ドット(.)、カンマ(,) 塩基が参照塩基と一致
- ACGTN acgtn 塩基が参照塩基とミスマッチしている
- > < 参照スキップ (CIGAR "N ")
- * */# 参照塩基の削除(CIGAR "D")
- $ リードでカバーされる最後の位置の場合
読み取った塩基の後に挿入があった場合、"\+[0-9]+[ACGTNacgtn*#]+"にマッチするテキスト:"+"文字の後に挿入の長さを示す整数が続き、その後に挿入された配列が表示される。パッドは "*"で表示され、--reverse-delが使用された場合は、逆鎖のパッドが "#"で表示される。
この読み取り塩基の後に削除がある場合、「-[0-9]+[ACGTNacgtn]+」に一致するテキスト:「-」文字の後に削除された参照塩基が同様に表される。
Samblaster(紹介)
インストール
#bioconda
mamba install -c bioconda samblaster -y
samblaster: Version 0.1.25
Author: Greg Faust (gf4ea@virginia.edu)
Tool to mark duplicates and optionally output split reads and/or discordant pairs.
Input sam file must contain sequence header and be grouped by read ids (QNAME).
Input typicallly contains paired-end data, although singleton data is allowed with --ignoreUnmated option.
Output will be all alignments in the same order as input, with duplicates marked with FLAG 0x400.
Usage:
For use as a post process on an aligner (eg. bwa mem):
bwa mem <idxbase> samp.r1.fq samp.r2.fq | samblaster [-e] [-d samp.disc.sam] [-s samp.split.sam] | samtools view -Sb - > samp.out.bam
bwa mem -M <idxbase> samp.r1.fq samp.r2.fq | samblaster -M [-e] [-d samp.disc.sam] [-s samp.split.sam] | samtools view -Sb - > samp.out.bam
For use with a pre-existing bam file to pull split, discordant and/or unmapped reads without marking duplicates:
samtools view -h samp.bam | samblaster -a [-e] [-d samp.disc.sam] [-s samp.split.sam] [-u samp.umc.fasta] -o /dev/null
For use with a bam file of singleton long reads to pull split and/or unmapped reads with/without marking duplicates:
samtools view -h samp.bam | samblaster --ignoreUnmated [-e] --maxReadLength 100000 [-s samp.split.sam] [-u samp.umc.fasta] | samtools view -Sb - > samp.out.bam
samtools view -h samp.bam | samblaster --ignoreUnmated -a [-e] [-s samp.split.sam] [-u samp.umc.fasta] -o /dev/null
Input/Output Options:
-i --input FILE Input sam file [stdin].
-o --output FILE Output sam file for all input alignments [stdout].
-d --discordantFile FILE Output discordant read pairs to this file. [no discordant file output]
-s --splitterFile FILE Output split reads to this file abiding by paramaters below. [no splitter file output]
-u --unmappedFile FILE Output unmapped/clipped reads as FASTQ to this file abiding by parameters below. [no unmapped file output].
Requires soft clipping in input file. Will output FASTQ if QUAL information available, otherwise FASTA.
Other Options:
-a --acceptDupMarks Accept duplicate marks already in input file instead of looking for duplicates in the input.
-e --excludeDups Exclude reads marked as duplicates from discordant, splitter, and/or unmapped file.
-r --removeDups Remove duplicates reads from all output files. (Implies --excludeDups).
--addMateTags Add MC and MQ tags to all output paired-end SAM lines.
--ignoreUnmated Suppress abort on unmated alignments. Use only when sure input is read-id grouped,
and either paired-end alignments have been filtered or the input file contains singleton reads.
-M Run in compatibility mode; both 0x100 and 0x800 are considered chimeric. Similar to BWA MEM -M option.
--maxReadLength INT Maximum allowed length of the SEQ/QUAL string in the input file. [500]
Primarily useful for marking duplicates in files containing singleton long reads.
--maxSplitCount INT Maximum number of split alignments for a read to be included in splitter file. [2]
--maxUnmappedBases INT Maximum number of un-aligned bases between two alignments to be included in splitter file. [50]
--minIndelSize INT Minimum structural variant feature size for split alignments to be included in splitter file. [50]
--minNonOverlap INT Minimum non-overlaping base pairs between two alignments for a read to be included in splitter file. [20]
--minClipSize INT Minumum number of bases a mapped read must be clipped to be included in unmapped file. [20]
-q --quiet Output fewer statistics.
bwa memのアライメントの過程でduplicationにタグをつける。
bwa index -a is input.fa
bwa mem -t 12 -R "@RG\tID:X\tLB:Y\tSM:Z\tPL:ILLUMINA" input.fa *.fastq | samblaster |samtools view -@ 12 -Sb - |samtools sort -@ 12 - > samp.sorted.bam
discordant-readとsplit-readは別ファイルに出力。
bwa mem -t 12 -R "@RG\tID:X\tLB:Y\tSM:Z\tPL:ILLUMINA" input.fa *.fastq | samblaster -e -d samp.disc.sam -s samp.split.sam > output.sam
- -e Exclude reads marked as duplicates from discordant, splitter, and/or unmapped file.
- -d FILE Output discordant read pairs to this file. [no discordant file output]
- -s FILE Output split reads to this file abiding by paramaters below. [no splitter file output]
duplication-readは全出力から除く。
bwa mem -t 12 -R "@RG\tID:X\tLB:Y\tSM:Z\tPL:ILLUMINA" input.fa *.fastq | samblaster -r -e -d samp.disc.sam -s samp.split.sam > output.sam
- -r Remove duplicates reads from all output files.
注意;bwa memで-M(mark shorter split hits as secondary)をつけている時は、samblasterにも-Mをつけてランを行う。
BamDeal(紹介)
BamDeal convert
bam2soap - bam/sam => SOAP bam
BamDeal convert bam2soap -InFile in.bam -OutPut out_SOAP.bam
soap2bam - SOAP bam => bam/sam
BamDeal convert soap2bam -InSoap in_SOAP.bam -OutBam out.bam -Dict Ref.fa
bam2fq - bam/sam => fastq
BamDeal convert bam2fq -i in.bam -o out.fq
=> out.fq.gzが出力される
bam2fa - bam/sam => fasta
BamDeal convert bam2fa -i in.bam -o out.fa
=> out.fa.gzが出力される
BamDeal modify
bamFilter -低クオリティなリードをフィルタリング
#Q15以下のリード、30bp以下のリード、duplicateリードをフィルタリング
BamDeal modify bamFilter -i in.bam -o out.bam -q 15 -l 30 -d
- -q the quality to filter reads, default [15]
- -l the length to filter reads, default [30]
- -s the beginning of interval containing the 1-based leftmost mapping position of first matching base, default [0]
- -e the end of interval containing the 1-based leftmost mapping position of first matching base, default [1e9]
- -c specify the chromosome to output, default [all chromosomes]
- -d remove the duplicate read
bamSplit - クロモソームごとにbamを分離
mkdir output_dir
BamDeal modify bamSplit -i in.bam -o output_dir -q 0
- -r reset output files headers by remove the chromosomes not in the output files
bamAssign - ユーザー指定の組み合わせでbamを分離(詳細はBamDeal modify bamAssign -h参照)
mkdir out_dir
BamDeal modify bamAssign -l list -o output_dir
-
-a list indicating how to assign chromosomes to outputs
-
-r reset output files headers by remove the chromosomes not in the output files
bamCat - bamをマージ
bamCat -i A.bam B.bam -o merged.bam -s
-
-s output sort bam file when all inputs were sorted
bamRand - bamをランダムサンプリング
#10 percent
BamDeal modify bamRand -i in.bam -p 0.1 -o out.bam
- -p probability with which each read would be outputed, default [0.1]
- -s random seed, default [time]
bamSubChr - bamからリストで指定したchrを削除、または追加
#リストのchrを除去
bamSubChr -i in.bam -d delete.list -o out.bam -r
#リストのchrを追加
bamSubChr -i in.bam -k keep.list -o out.bam -r
#unmapのリードを除去
bamSubChr -i in.bam> -o AAA -u
- -k list of chromosomes to be kept
- -d list of chromosomes to be deleted
- -u remove unmapped reads
- -r reset output headers by remove the chr(s) not in the out files
bamShiftQ - Phred qualityのタイプを修正
#リードのPhred qualityをASCII33に修正して出力
bamShiftQ -i in.bam -o out.bam -p 1
#リードのPhred qualityをASCII64に修正して出力
bamShiftQ -i in.bam -o out.bam -p 2
- -p phred quality in output BAM, [1]: ASCII+33 or [2]: ASCII+64, default [1]
- -q the quality to filter reads, default [10]
- -l the length to filter reads, default [30]
bamLimit - 大きなbamを分割
#最大1000000リードずつ分割
mkdir out_dir
BamDeal modify bamLimit -i in.bam -o out_dir/ -n 1000000
- -n max read number for each bam[1000000000]
BamDeal statistics
Coverage - Coverage/Depth/GC分布を調べる
BamDeal statistics Coverage -i in.bam -r ref.fasta -o outprefix
- -r input reference FASTA to get Depth-GC wig
-
-b list of the regions of which the coverage and mean of depth would be given
- -d Filter the duplicated read
BasesCount - 全ポジションのATGC全てのデプスカウント
#3つのbamを調べる。q10以上
BamDeal statistics BasesCount -i A.bam B.bam C.bam -o outprefix -q 10
DeteCNV - CNV/Deletionを検出
BamDeal statistics DeteCNV -i A.bam B.bam -r ref.fasta -m 1000 -o outprefix
-
-f <float> depthRatio to judge breakpoint of merge adjacent[0.45]
- -c for each chromosome, use its own mean of depth into calculation default would use the mean of depth of the whole genome
-
-m <int> set the minimum length of CNV, default [1800]
-
-p <float> p-value of CNV depth bias, default [0.02]
DeteSV - ペアエンドのインサートサイズ情報を使ってSVを検出
BamDeal statistics DeteSV -i in.bam -r ref.fasta -o outprefix -m 1800
LowDepth - low depthの領域を検出
BamDeal statistics LowDepth -i in.bam -o out.bed -q 10 -s 1000
- -o output bed region file
- -x set the minimum value of low depth,default[2]
- -s the length to filter short region, default [1000]
BamDeal visualize
StatQC - クオリティレポート出力(Rのggplot2とreshapeパッケージが必要*1)
BamDeal visualize StatQC -i in.bam -o outdir
#複数bamをlist指定
BamDeal visualize StatQC -i in.bam -o outdir
分析結果のテキストとPDFが出力される。
DepthCov - リードデプス出力
BamDeal visualize DepthCov -i in.bam -o out
DepthCov - リードデプス-GCプロット出力
BamDeal visualize DepthCov -i in.bam -r ref.fasta -o outprefix -k -q 10
DepthSlide - ゲノムのchr、ポジション順のマンハッタンプロット出力
BamDeal visualize DepthSlide -i in.bam -r ref.fasta -o outprefix
Alfred(紹介)
BAM統計、フィーチャーカウント、フィーチャーアノテーションのツール。
インストール
#bioconda (errorはこちらを参照)
mamba install -c bioconda alfred -y
リファレンスのFASTAと.bamを指定する。
alfred qc -r ref.fa -o qc.tsv.gz input.bam
#結果の可視化
#なければ本体をcloneして取ってくる
git clone https://github.com/tobiasrausch/alfred.git
#可視化
Rscript alfred/R/stats.R qc.tsv.gz
出力例(direct link)。
bamM(紹介)
インストール
#bioconda
mamba create -n bamM -y python=2.7
conda activate bamM
mamba install -c bioconda bamm -y
# bamm
...::: BamM :::...
Working with the BAM, not against it...
-------------------------------------------------------------------------
version: 1.7.3
-------------------------------------------------------------------------
bamm make -> Make BAM/TAM files (sorted + indexed)
bamm parse -> Get coverage profiles / linking reads / insert types
bamm extract -> Extract reads / headers from BAM files
bamm filter -> Filter BAM file reads
USE: bamm OPTION -h to see detailed options
bamm make mappingしてbamを作成
ペアエンド、シングルエンド、インターリーブのfastqをそれぞれ指定する。20スレッド指定する。unmapのリードは出力しない(default)。
bamm make -d ref.fasta -c pair_1.fastq.gz pair_2.fastq.gz \
-s unpaired.fastq.gz -i interleave.fq.gz \
-p mapped -t 20 --alignment_algorithm mem -f
bamが出力される。
bamm parse カバレッジやインサートサイズ計算
複数のbamのカバレッジ、インサートサイズを出力する。
bamm parse -c covs.tsv -l links.tsv -i inserts.tsv -t 8 -m pmean \
-b f1.bam f2.bam f3.bam
extract
bamからリードを出力する。出力はgzip圧縮される。
bamm extract -g ref.fasta -b mapped.bam -t 8
ペアエンドのbamを指定した場合、ペアエンドfastq、ペアエンドfastqでsingletonになったfastqの3つのファイルが出力される。ペアエンドfastqの順番は同期されている。
KneadData(紹介)
メタゲノムからヒトゲノムなどホストゲノムの汚染を除く作業を自動で行う。有名なhuttenhowerラボのツール。
(初回のみ)初めにリファレンスゲノムのインデックスを作成する。ヒトやマウスのゲノムならKneadDataのコマンドからbowite2のindex作成済みゲノムをダウンロードできる。
mkdir genome
kneaddata_database --download human_genome bowtie2 genome/ #3.44GBある
(初回のみ)indexを作成
bowtie2-build Homo_sapiens.fasta -o Homo_sapiens_db test
#作成したindexをディレクトリに収納する
mkdir genome_index
mv test* genome_index/
作成したindexのフォルダを指定してランする。
#single
kneaddata --i seq.fastq --reference-db genome_index/ --output kneaddata_output -t 20
#pair
kneaddata --i seq1.fastq --i seq2.fastq -db genome_index/ --output kneaddata_output -t 20
- -t number of threads [ Default : 1 ]
- -i input FASTQ file (add a second argument instance to run with paired input files)
- -o directory to write output files
- -d location of reference database (additional arguments add databases)
Trimmomaticがないと言われたら --trimmomatic フラグをつけて trimmomaticのディレクトリを指定する。面倒ならwgetでダウンロードしておいておけばいい。
wget http://www.usadellab.org/cms/uploads/supplementary/Trimmomatic/Trimmomatic-0.33.zip
unzip Trimmomatic-0.33.zip
#trimmomaticを指定してラン
kneaddata --input seq1.fastq --input seq2.fastq -db genome_index/ --output kneaddata_output -t 20 --trimmomatic Trimmomatic-0.33/
fastqp(紹介)
fastqp input.bam
複数のmetricsで分析が行われる。
BAMStats
bamのアライメント情報をGUIで確認できるツール。samtools statコマンドを利用してる。Sourceforgeからビルド済みのJAVA実行ファイルをダウンロードする。
http://bamstats.sourceforge.net
java -Xmx6g -jar BAMStats-GUI-1.25.jarをコマンドラインで叩くか、このファイルをダブルクリックするとアプリが起動する。
GUI版では下のような情報を見ることができる。
このように、平均や中央値でなく、分布でリードのアライメント状況を見ることができる。 bam/samを追加して複数データを同時比較することも可能である。
BAMStatsはコマンドライン版もある。ランするには以下のように打つ。
java -Xmx4g -jar BAMStats-1.25.jar -i input.bam
- -iでbam/samを指定する。
samstat
公式サイト SAMStat
fastq、sam、bamなどを分析できる。インストールは
tar -zxcf samstat.tgz
cd samstat
./configure
make clean
make
ビルドできたらsrc/のsamstatにパスを通す。
fastqの分析
samstat input.fq
~.htmlが出力される。


sam/bamの分析
samstat input.bam
~.htmlが出力される。

ショートリード向けに設計されているので、ロングリードを使うとエラーになる。
bcftools
非常にたくさんのコマンドがある。よく使うだろうコマンドを紹介する。
インストール
#homebrew
brew install bcftools
#bioconda
mamba install -c bioconda bcftools -y
# bcftools
Program: bcftools (Tools for variant calling and manipulating VCFs and BCFs)
Version: 1.9 (using htslib 1.9)
Usage: bcftools [--version|--version-only] [--help] <command> <argument>
Commands:
-- Indexing
index index VCF/BCF files
-- VCF/BCF manipulation
annotate annotate and edit VCF/BCF files
concat concatenate VCF/BCF files from the same set of samples
convert convert VCF/BCF files to different formats and back
isec intersections of VCF/BCF files
merge merge VCF/BCF files files from non-overlapping sample sets
norm left-align and normalize indels
plugin user-defined plugins
query transform VCF/BCF into user-defined formats
reheader modify VCF/BCF header, change sample names
sort sort VCF/BCF file
view VCF/BCF conversion, view, subset and filter VCF/BCF files
-- VCF/BCF analysis
call SNP/indel calling
consensus create consensus sequence by applying VCF variants
cnv HMM CNV calling
csq call variation consequences
filter filter VCF/BCF files using fixed thresholds
gtcheck check sample concordance, detect sample swaps and contamination
mpileup multi-way pileup producing genotype likelihoods
roh identify runs of autozygosity (HMM)
stats produce VCF/BCF stats
Most commands accept VCF, bgzipped VCF, and BCF with the file type detected
automatically even when streaming from a pipe. Indexed VCF and BCF will work
in all situations. Un-indexed VCF and BCF and streams will work in most but
not all situations.
Consensus fasta
minimap2を使いfastqをリファレンスにmappingしてbamを作成、そのbamからリファレンスと異なる部位を修正したコンセンサス配列を作成する。
ここではnanoporeのlong readを使っている。
#mapping to the ref
minimap2 -t 8 -ax map-ont ref.fa long.fq \
|samtools sort -@ 8 -O BAM - > mapping.bam \
&& samtools index -@ 8 mapping.bam
#pileup and consensus calling
samtools mpileup -C50 -uf ref.fa mapping.bam | bcftools call -c -Oz -o calls.vcf.gz
#indexing
bcftools index calls.vcf.gz
#export consensus fasta
cat ref.fa | bcftools consensus calls.vcf.gz > consensus.fa
samtools mpileup
- -C, --adjust-MQ INT adjust mapping quality; recommended:50, disable:0 [0]
- -f faidx indexed reference sequence file
bcftools call
- -c, --consensus-caller the original calling method (conflicts with -m)
- -o, --output <file> write output to a file [standard output]
- -O, --output-type <b|u|z|v> output type: 'b' compressed BCF; 'u' uncompressed BCF; 'z' compressed VCF; 'v' uncompressed VCF [v]
fass
インストール
#bioconda
mamba install -c bioconda bamutil -y
$ bam -h
Set of tools for operating on SAM/BAM files.
Version: 1.0.14; Built: Tue Jun 26 23:14:05 PDT 2018 by distiller
Tools to Rewrite SAM/BAM Files:
convert - Convert SAM/BAM to SAM/BAM
writeRegion - Write a file with reads in the specified region and/or have the specified read name
splitChromosome - Split BAM by Chromosome
splitBam - Split a BAM file into multiple BAM files based on ReadGroup
findCigars - Output just the reads that contain any of the specified CIGAR operations.
Tools to Modify & write SAM/BAM Files:
clipOverlap - Clip overlapping read pairs in a SAM/BAM File already sorted by Coordinate or ReadName
filter - Filter reads by clipping ends with too high of a mismatch percentage and by marking reads unmapped if the quality of mismatches is too high
revert - Revert SAM/BAM replacing the specified fields with their previous values (if known) and removes specified tags
squeeze - reduces files size by dropping OQ fields, duplicates, & specified tags, using '=' when a base matches the reference, binning quality scores, and replacing readNames with unique integers
trimBam - Trim the ends of reads in a SAM/BAM file changing read ends to 'N' and quality to '!' or softclipping the ends (resulting file will not be sorted)
mergeBam - merge multiple BAMs and headers appending ReadGroupIDs if necessary
polishBam - adds/updates header lines & adds the RG tag to each record
dedup - Mark Duplicates
dedup_LowMem - Mark Duplicates using only a little memory
recab - Recalibrate
Informational Tools
validate - Validate a SAM/BAM File
diff - Diff 2 coordinate sorted SAM/BAM files.
stats - Stats a SAM/BAM File
gapInfo - Print information on the gap between read pairs in a SAM/BAM File.
Tools to Print Information In Readable Format
dumpHeader - Print SAM/BAM Header
dumpRefInfo - Print SAM/BAM Reference Name Information
dumpIndex - Print BAM Index File in English
readReference - Print the reference string for the specified region
explainFlags - Describe flags
Additional Tools
bam2FastQ - Convert the specified BAM file to fastQs.
Dummy/Example Tools
readIndexedBam - Read Indexed BAM By Reference and write it from reference id -1 to maxRefId
Usage:
bam <tool> [<tool arguments>]
The usage for each tool is described by specifying the tool with no arguments.
fastqの抽出
bam bam2FastQ --in input.bam --outBase outprefix
ペアエンドは同期されて出力される。同期できなかったリードはシングルで出力される。
NGSUtils - bamutils
#bioconda (link) linux (テストした時は導入できなかった)
mamba create -n ngsutils -y python=2.7
conda activate ngsutils
mamba install -c bioconda ngsutils -y
keepbest(リンク) best mappingを抽出。
bamutils keepbest input.bam output.bam
export(リンク) 指定した情報を全リードから出力
bamutils export -name input.bam > output #リード名を出力
bamutils export -ref input.bam > output #mappingされたクロモソーム名を出力
expressed(リンク) マッピングされた領域をBED形式で出力。(samtools indexでbam.baiファイルを準備しておく必要がある)
bamutils expressed input.bam > output.bed
filter(リンク) 指定した条件を満たすリードだけbamで出力。
#chr1にマッピングされており、リード長が100以上でペアエンドが-> <-の向きに両方マップングされているリードだけbamで出力。
bamutils filter input.bam output.bam -minlen 100 -properpair -includeref chr1
merge(リンク) bamをマージ(best matchを選択してくる)。
bamutils merge output.bam input1.bam input2.bam
pair(リンク)別々にマッピングされているペアエンドについて、指定条件内でペアを再度調べる。
bamutils pair output.bam input1.bam input2.bam
removevlipping(リンク)hard-clipされた領域を捨てる。
bamutils removeclipping input.bam output.bam
renamepair(リンク)一部アライナーはペアエンドのリード名に/1や/2をつけてbsmを作り、これが下流の解析でエラーになる。これを消す(ペアエンドは同じリード名になる)。
bamutils renamepair input.bam output.bam
tag(リンク)tagをbamに追加する。
bamutils tag -xs input.bam output.bam
check(リンク)破損のチェック。
bamutils check input.bam
cleancigar(リンク)CIGAR情報の修復。
bamutils cleancigar input.bam
stats(リンク)bamを分析。
bamutils stats input.bam
$ bamutils stats input.bam
Calculating Read stats...
Done! (0:36)
out.bam
Reads: 1083286
Mapped: 1083286
Unmapped: 0
Flag distribution
[0x001] Multiple fragments 1083286 100.00%
[0x002] All fragments aligned 888871 82.05%
[0x008] Next unmapped 58635 5.41%
[0x010] Reverse complimented 542299 50.06%
[0x020] Next reverse complimented 539537 49.81%
[0x040] First fragment 1083286 100.00%
[0x800] Supplementary 1 0.00%
Template length: 568.90 +/- 824.04
Reference counts count
1 1083286
bedファイルを出力するにはbedtoolsを使う。
bamをbedに変換する(bed)。
bedtools bamtobed -i reads.bam > reads.bed
bedtoolsについては以下にまとめています。
bam/samが増えてきていちいちコマンドを実行するのが面倒になったら、スクリプトを組む事を考える。ただし簡単な作業ならparallelを使い並列化した方が簡単で高速な場合も多い。
例えば特定のディレクトリにbamが複数あり、それぞれについてpicardを使いマッピングスコアを分析する。
find *bam | parallel 'Picard QualityScoreDistribution INPUT={} OUTPUT={}.quality_score_dist.tsv CHART_OUTPUT={}.quality_score_dist.pdf'
bamそれぞれについてpdfファイルとtsvファイルが出力される。
findでbamファイルを全て拾い、parallelコマンドに渡している。parallelコマンドが' 'で囲まれた部位を並列実行する。ただし負荷はファイル分増加するので気をつけること。
fasta、fastqなどを扱うなら、seqkitが高速で使い易いと思います。ランダムサインプリング、逆相補鎖に変換、ソート、重複の除去、指定した範囲だけ抽出など、なんでもできます。
以下にまとめています。
引用
BCFtools/RoH: a hidden Markov model approach for detecting autozygosity from next-generation sequencing data.
Narasimhan V1, Danecek P1, Scally A2, Xue Y1, Tyler-Smith C1, Durbin R1.
Bioinformatics. 2016 Jun 1;32(11):1749-51. doi: 10.1093/bioinformatics/btw044. Epub 2016 Jan 30.
https://www.ncbi.nlm.nih.gov/pubmed/26826718
The Sequence Alignment/Map format and SAMtools.
Li H1, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R; 1000 Genome Project Data Processing Subgroup.
Bioinformatics. 2009 Aug 15;25(16):2078-9. doi: 10.1093/bioinformatics/btp352. Epub 2009 Jun 8.
https://www.ncbi.nlm.nih.gov/pubmed/19505943
SAMStat: monitoring biases in next generation sequencing data.
Lassmann T1, Hayashizaki Y, Daub CO.
Bioinformatics. 2011 Jan 1;27(1):130-1. doi: 10.1093/bioinformatics/btq614. Epub 2010 Nov 18.
https://www.ncbi.nlm.nih.gov/pubmed/21088025
BamTools: a C++ API and toolkit for analyzing and managing BAM files.
Barnett DW1, Garrison EK, Quinlan AR, Strömberg MP, Marth GT.
Bioinformatics. 2011 Jun 15;27(12):1691-2. doi: 10.1093/bioinformatics/btr174. Epub 2011 Apr 14.
BamTools: a C++ API and toolkit for analyzing and managing BAM files. - PubMed - NCBI
VariantBam: filtering and profiling of next-generational sequencing data using region-specific rules.
Wala J, Zhang CZ, Meyerson M, Beroukhim R
Bioinformatics. 2016 Jul 1;32(13):2029-31.
biobambam: tools for read pair collation based algorithms on BAM files
German Tischler, Steven Leonard
Source Code Biol Med. 2014; 9: 13.
elPrep: High-Performance Preparation of Sequence Alignment/Map Files for Variant Calling.
Herzeel C, Costanza P, Decap D, Fostier J, Reumers J.
PLoS One. 2015 Jul 16;10(7):e0132868.
parallel
samtools
http://davetang.org/wiki/tiki-index.php?page=SAMTools
bamのsplit
https://www.biostars.org/p/46327/
Biostars
https://www.biostars.org/p/247903/
Tip and tricks for BAM files
https://github.com/IARCbioinfo/BAM-tricks
参考
*1
このまま実行しても、大半のコンセンサスはnになってしまうと思います。flagで取り出すリードを調節したり、エラーコレクションを挟んだりする必要があるかなと思います。