2019/10/24 MetaCycの結果追記
2021 1/27 誤字修正
ゲノムアノテーションは、文字通り、アセンブリされたDNA分子の特徴の注釈である。そのような特徴は、そもそも、タンパク質をコードする遺伝子[「オープンリーディングフレーム」(ORF)]およびリボソームまたはトランスファーRNA分子をコードする遺伝子を含む遺伝子である。アノテーションは、このような機能の識別と、各機能に、考えられる生物学的機能に関するヒントの意味のあるリストを提供することで構成される。アノテーションは通常、ゲノムまたはメタゲノムデータの自動計算処理の最終ステップであり、生物学の始まりとなる。
背景と研究の質問に応じて、生物学者は異なるアノテーションのニーズを持つ。たとえば、研究が単一の微生物を対象とする場合、そのゲノムの詳細な遺伝子ごとのアノテーションが望まれる。一方、研究が完全な生態系を対象とする場合、関連するメタゲノムの機能的可能性の高レベルの要約が目的かもしれない。これらの例には、アノテーションの別の開始点も表示される。前者の場合、単一のほぼ完全な全ゲノム配列で構成されている。 2番目のケースでは、さまざまな品質の多数のMAG、ビニングされていないメタゲノムコンティグ、または数十億ものアセンブルされていないリードで構成されている場合がある。
メタゲノムデータの処理においてアノテーションを他の計算ステップと区別するのは、アノテーションツールのベンチマークが存在しないということである。つまり、これらのツールをランキングし、勝者を客観的に宣言することは簡単ではない。最適なアノテーションパイプラインの選択は、データ、研究の質問、利用可能な計算リソース、およびアノテーションソフトウェアのヒントとその提示方法を理解する必要がある研究者の背景に依存する。
実際には、ゲノムアノテーションのオプションには、オンラインプラットフォームとスタンドアロンパイプラインの2種類がある。オンラインプラットフォームの例として、IMG(Chen et al、2017)、MG-RAST(Keegan et al、2016)、MicroScope(Vallenet et al、2017)、Mgnify(Mitchell et al、2018)、およびEdge( Li et al、2017)などがある。プラットフォームを選択すると、ローカルコンピューティングインフラストラクチャや、面倒なツールとデータベースのインストールと更新の必要性を回避しながら、オンラインコラボレーション機能を活用できる。これらのプラットフォームはデータリポジトリでもあるため、プラットフォームは公開後にデータを共有するためのアクセッション番号を提供する場合がある。
ただし、プラットフォームは特定の研究質問に必要なアノテーションのタイプを提供しなかったり、ツールとデータベースの最新版の組み込みが遅くなる事もある。そのような要因が重要な場合は、スタンドアロンパイプラインを選択するのがよいだろう。 PythonやPerlなどのスクリプト言語に堪能なサイエンティストは、独自のパイプラインを最初から作成することさえある。アセンブルされたコンティグ、scaffolds、または全ゲノム配列のアノテーションに使用可能なスタンドアロンパイプラインの例は、Prokka(Seemann、2014)、DFAST-core(Tanizawa et al、2018)、およびPGAP(Tatusova et al。、2016)がある。 Prokkaは非常に高速なゲノムアノテーションパイプラインである。その中核となる概念は、一部のデータベースまたはツールが他のものよりも優れたまたはより多くの情報を提供することである。良いデータベースへの肯定的な「ヒット」で遺伝子にアノテーションが付けられると、追加の検索を実行する必要はない。 DFASTは、より高速な類似検索ツール(GHOSTX)を使用することで、このアプローチに追加する。クエリゲノムとユーザーが追加したカスタムリファレンスゲノムを含む可能性のあるリファレンスゲノムのより大きなセットとの間の相互のベストブラストヒットに基づいたオルソログの割り当てを推測する。したがって、十分にアノテーションが付けられたリファレンスゲノムからアノテーションを転送することが特に有用である。 NCBIはPGAPを使用して、submitされた全ゲノム配列にアノテーションを付ける。洗練された遺伝子予測アルゴリズムと、その原核生物タンパク質クラスターセットへの遺伝子アサインを組み合わせている(Klimke et al、2009)。機関の「ゴールドスタンダード」アノテーションとして、アノテーションの標準と規約、品質管理、および実行中のdue diligence(wiki)を重視している。
ここでは、metagenome-assembled genomes(MAG)用に開発された拡張可能なスタンドアロンアノテーションパイプラインであるMetaErgを紹介する。ゲノム中心のメタゲノムデータには、3つの大きな課題がある。 1つ目は、アセンブリ品質が比較的低く、「外来」遺伝子によるMAGの汚染が予想されることである。この課題は、単純に機能を推測し、各遺伝子を分類学的に分類するために必要なものよりもはるかに大きなデータベースに対して高速の類似性検索を実行することによって対処される。これにより、潜在的に人為的なハイブリッドビンまたはコンティグの検出が可能になる。 2つ目は、ユーザーが多くのアノテーション付きゲノムを同時に理解する必要がある可能性が高いことである。この課題は、エンコードされた生物学的機能およびパスウェイに関する迅速な推論を可能にするために、データを視覚化および要約することにより対処される。 3番目は、多くの環境微生物について、意味のある/近いリファレンスゲノムがまだ利用できないことである。この課題は、さまざまなツールやデータベースから派生した各遺伝子に関する包括的な情報を常に提供し、機能を割り当てるだけでなく、リファレンス遺伝子との類似性が低い遺伝子に対して実際に可能なことによって対処される。
MetaErgは、メタゲノムでアセンブリされたコンティグにアノテーションを付けるための統合され完全自動化されたパイプラインである。多数のオープンソースツールを統合し、そのモジュラー設計により、柔軟なワークフロー、新しい機能の追加、および容易なリファクタリングが可能になる。 MetaErgの実装は、コマンドラインインターフェイス、入力DNAシーケンスのフィルタリングおよびフォーマット用の入力データ前処理モジュール、生物学的特徴および要素を予測するための構造アノテーションモジュール、rRNA遺伝子とORFを分類系統に分類し、注釈結果をさまざまな要約レポートで表示し、HTMLとJavaScriptを使用して視覚化するプレゼンテーションモジュール、遺伝子推定のための機能的アノテーションモジュールを含む5つの主要モジュール(論文図1)で構成される。(一部略) 最小限必要な入力として、事前にアセンブリされたFASTA形式のDNA配列ファイルを受け入れる。 パイプラインのオプションパラメータのデフォルト値は、メタゲノム解析用に最適化されている。 経験豊富なユーザーは、コマンドラインインターフェイスを介してプログラムと対話し、遺伝子予測とデータベース検索パラメーターのカスタマイズ、特定のツールと機能の有効化または無効化、データフィルターのしきい値の設定、出力ディレクトリの指定を行うことができる。(以下略)
If you are looking for a standalone metagenome annotation pipeline with gene prediction, gene annotation, taxon assignment, pathway prediction, and visualization, our MetaErg pipeline is now published at https://t.co/5wxKtkPPz5 pic.twitter.com/FJqQ7rmJsM
— Xiaoli Dong (@xiaoliidong) October 15, 2019
インストール
オーサーらの準備したdockerイメージをpullしてテストした(ホストOSはmacos10.14)。
依存
- ARAGORN: a program to detect tRNA genes and tmRNA genes in nucleotide sequences
- BLAST+ executables: The Basic Local Alignment Search Tool (BLAST) finds regions of local similarity between sequences.
- DIAMOND: a new high-throughput program for aligning DNA reads or protein sequences against a protein reference database
- Hmmer: HMMER is used for searching sequence databases for sequence homologs, and for making sequence alignments.
- MinCED: a program to find Clustered Regularly Interspaced Short Palindromic Repeats (CRISPRs) in full genomes or environmental datasets such as assembled contigs from metagenomes.
- MinPath: a parsimony approach for biological pathway reconstructions using protein family predictions, achieving a more conservative, yet more faithful, estimation of the biological pathways for a query dataset.
- Prodigal: Fast, reliable protein-coding gene prediction for prokaryotic genomes.
- SignalP: The program predicts the presence of signal peptides and the location of their cleavage sites in proteins from Archaea, Gram-positive Bacteria, Gram-negative Bacteria and Eukarya.
- TMHMM: a method for predicting transmembrane helices based on a hidden Markov model
本体 Github
#dockerhub (link) (*3)
docker pull xiaolidong/docker-metaerg
>docker run --rm -it xiaolidong/docker-metaerg metaerg.pl -h
$ docker run --rm -it xiaolidong/docker-metaerg metaerg.pl -h
Option h is ambiguous (help, hmmcutoff, hmmevalue)
Name:
Metaerg.pl 1.2.0 by Xiaoli Dong <xdong@ucalgary.ca>
Synopsis:
Metagenome annotation
Usage:
metaerg.pl [options] <contigs.fasta>
General:
--help This help
--version Print version and exit
input:
--dbdir [X] metaerg searching database directory (default '')
--mincontiglen [N] Minimum contig size [NCBI needs 200] (default '200')
gene prediction:
--gcode [N] translation table to use for gene predication (default '11')
--gtype [X] single or metagenome: [arc|bac|euk|meta] (default 'meta')
--minorflen [N] Minimum orf length (default '180')
--sp Enable signal peptide and cleavage site predication using signalp, it is slow when it is enabled (default OFF)
--tm Enable transmembrane helics predication using tmhmm, it is slow when it is enabled (default OFF)
Outputs:
--prefix [X] Filename output prefix (default '')
--outdir [X] Output folder [auto] (default '')
--force Force overwriting existing output folder (default OFF)
--locustag [X] Locus tag prefix (default 'metaerg.pl')
--increment [N] Locus tag counter increment (default '1')
Computation:
--cpus [N] Number of CPUs to use (default '8')
diamond cutoff:
--evalue [n.n] Similarity e-value cut-off (default '1e-05')
--identity [n.n] identity (default '20')
--coverage [n.n] coverage (default '70')
hmmsearch cutoff:
--hmmcutoff [X] hmm search trusted score threshold: [--cut_ga|--cut_nc|--cut_tc] (default '--cut_ga')
--hmmevalue [n.n] custom hmm db search threshold (default '1e-5')
abundance input:
--depth [X] contig coverage depth file in format of:contigid contigLe depth (default '')
> docker run --rm -it xiaolidong/docker-metaerg fastaContig2Gff.pl
Name:
by Xiaoli Dong <xdong@ucalgary.ca>
Synopsis:
extract subset of the gff file for the input contigs
Usage:
perl /NGStools/metaerg/bin/fastaContig2Gff.pl
-c <fasta format contig file>
-g <gff format metagenome annotation file generated using meta-annotator>
> docker run --rm -it xiaolidong/docker-metaerg output_reports.pl
Name:
output_reports.pl by Xiaoli Dong <xdong@ucalgary.ca>
Synopsis:
Generate all all type of reports based on the features in the gff file
Usage:
perl /NGStools/metaerg/bin/output_reports.pl
-g <input gff file: all.gff file produced by metaerg>
-f <fasta format contig file, the features were assocaited with this input file>
options:
-o <output dir, default will be the current directory>
-d <database dir, default will be inside of the metaerg directory>
(base) kamisakakazumanoMac-mini:WT kazu$
データベースの準備
ディレクトリworking_dirを作り、その中にデータベースを構築する。
mkdir working_dir && cd working_dir
docker run --shm-size 2g --rm -u $(id -u):$(id -g) -it -v $PWD:/data/ xiaolidong/docker-metaerg setup_db.pl -o /data -v 132
docker runのオプション
- --shm-size <bytes> Size of /dev/shm
- -u Username or UID (format: <name|uid>[:<group|gid>])
- -i Keep STDIN open even if not attached
- -t Allocate a pseudo-TTY
- -v list Bind mount a volume
--shm-sizeは一時ファイル領域の /dev/shmのサイズを指定するもの。defaultよりかなり増やしている。-uでホストユーザーID(uid):イメージ側のプライマリグループID(gid)と指定している。
/working_dirに/dbと/tmpができる。

構築されるデータベースについてはGithubに まとめられている。
実行方法
contigsのFASTA(gzip圧縮未対応)とデータベース(/db)、出力を指定する(*1)。
docker run --shm-size 2g --rm -u $(id -u):$(id -g) -it -v $PWD:/data \
-w /data xiaolidong/docker-metaerg metaerg.pl \
--dbdir db --outdir my_metaerg_output contig.fasta
- --cpus Number of CPUs to use (default '8')
- --outdir Output folder [auto] (default '')
- --dbdir metaerg searching database directory (default '')
出力

index.htmlから各結果にジャンプできる(*2)。
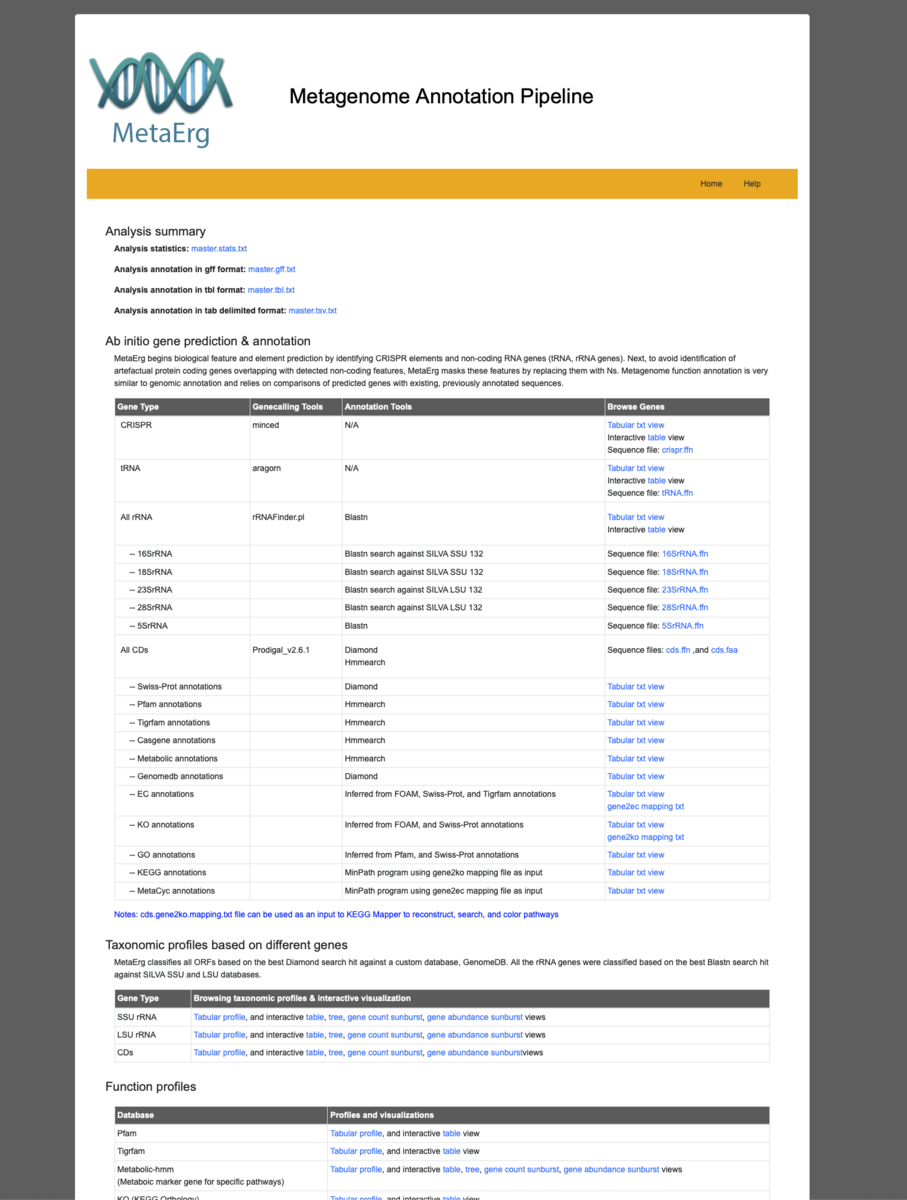
data/all.gffが全アノテーションを統合したgffファイルになる。

Ab initio gene prediction & annotation
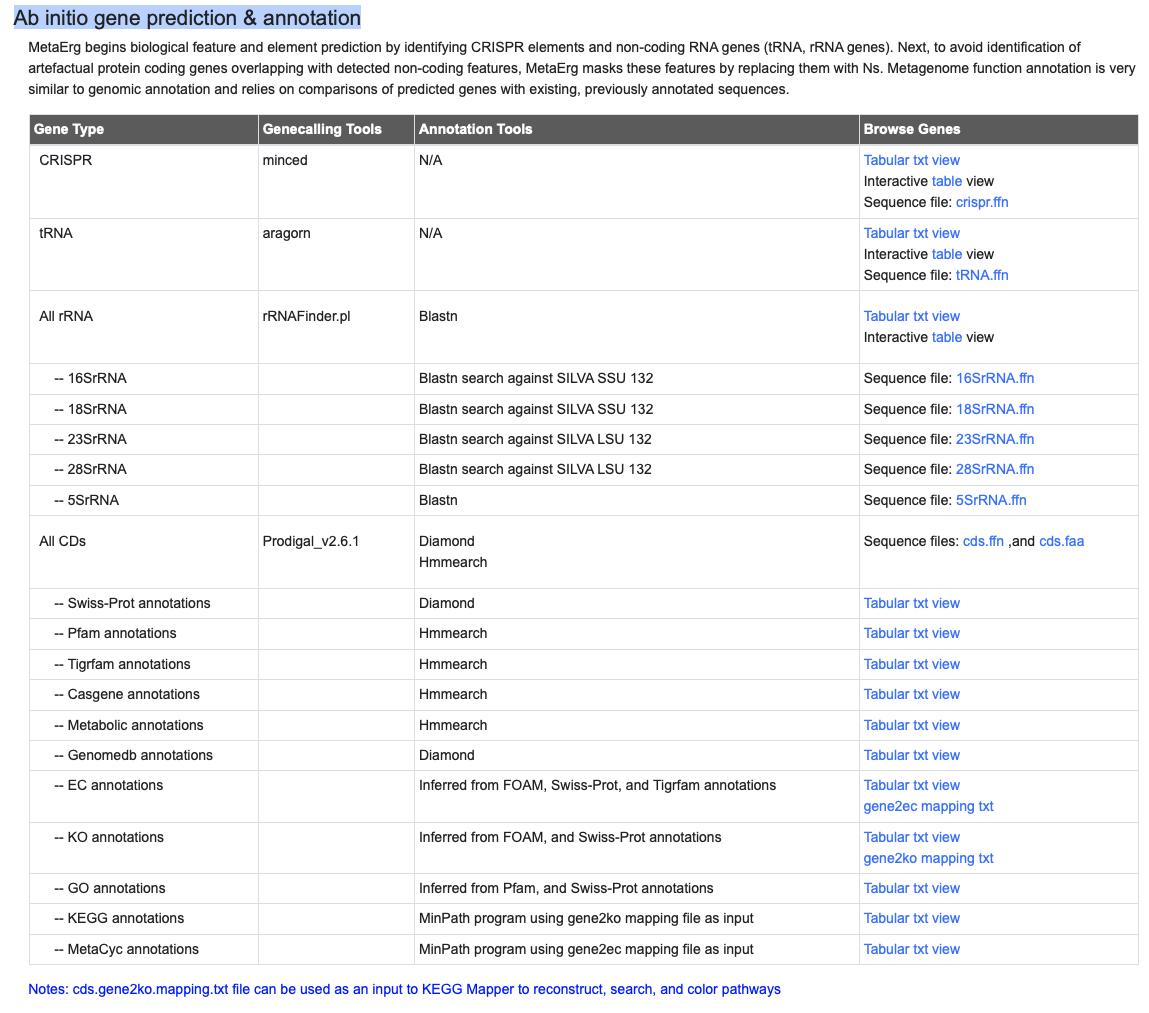
Taxonomic profiles based on different genes

taxonomic profiles of SSU rRNA
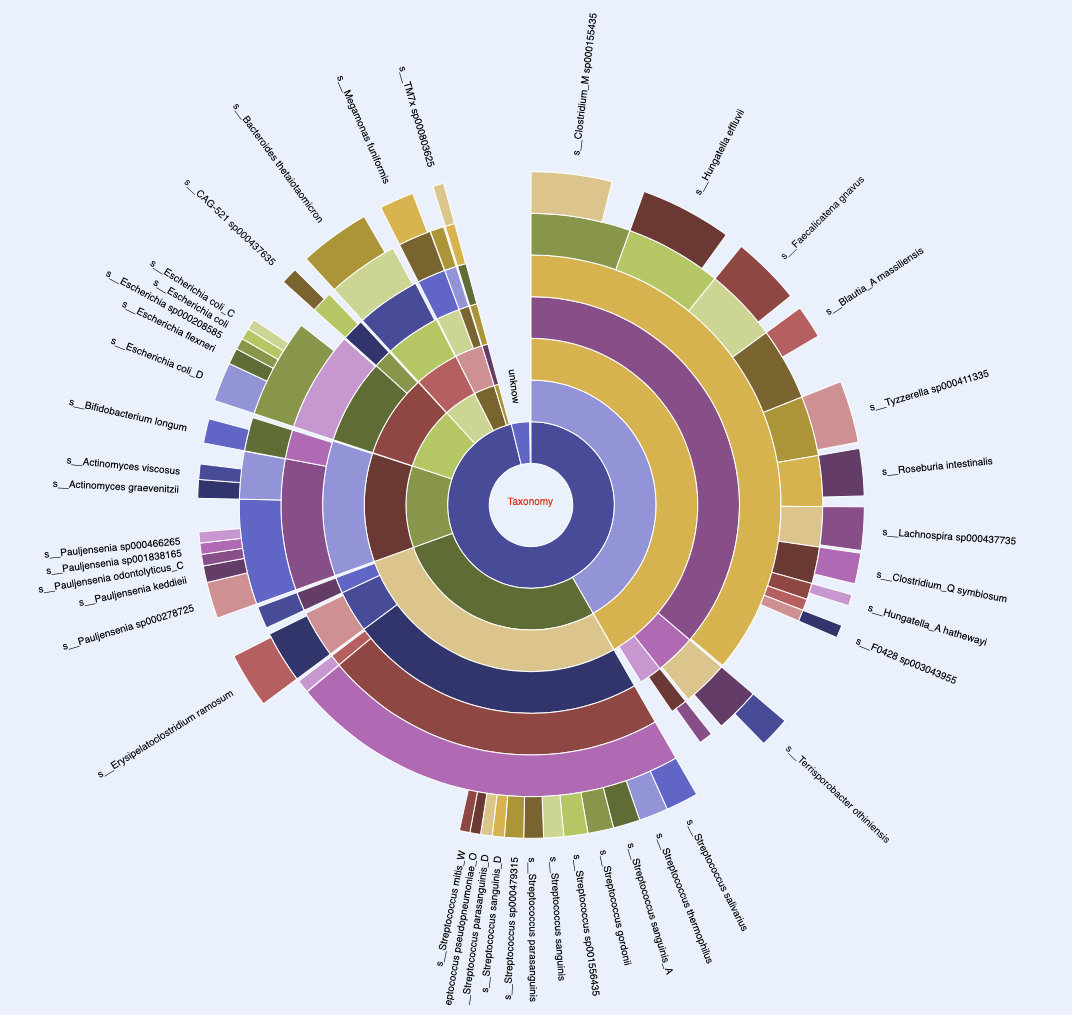
Function profiles
- Enzyme profile
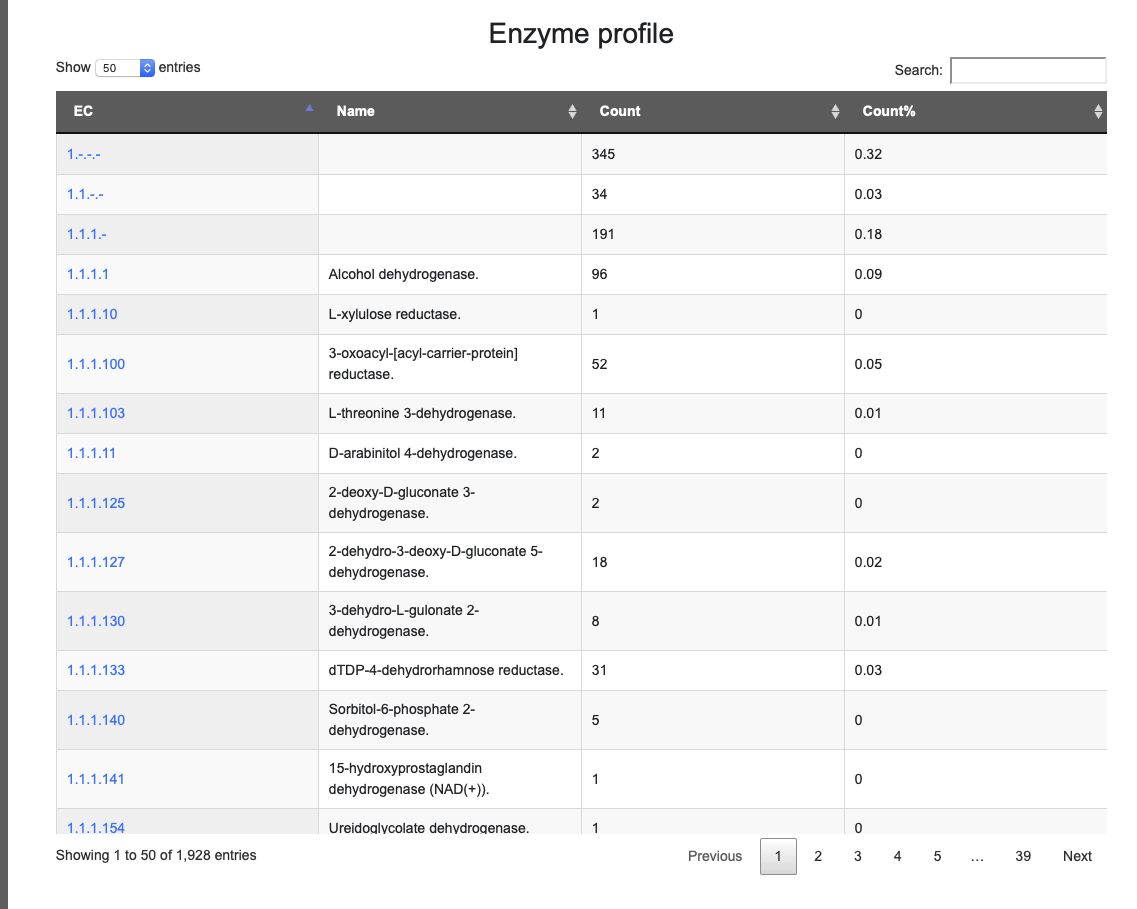
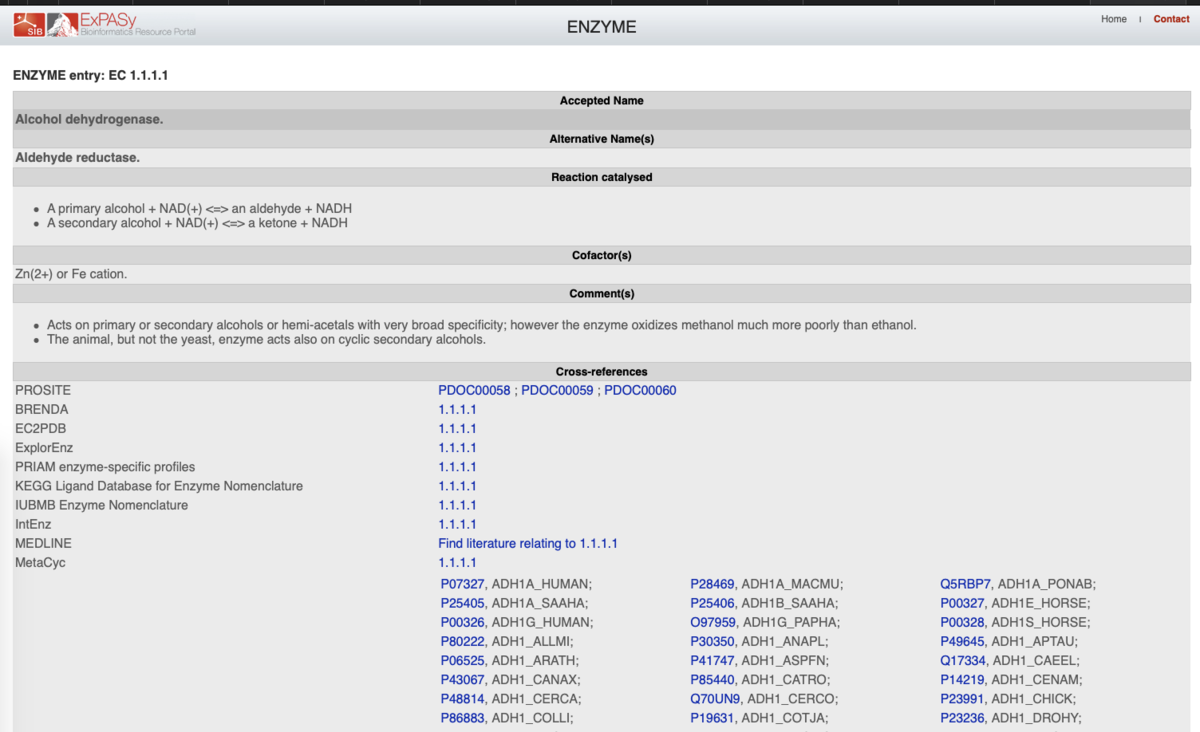
swissprotのENZYME にリンクしている。
Alcohol dehydrogenase.
- GO (Gene Ontology) profile
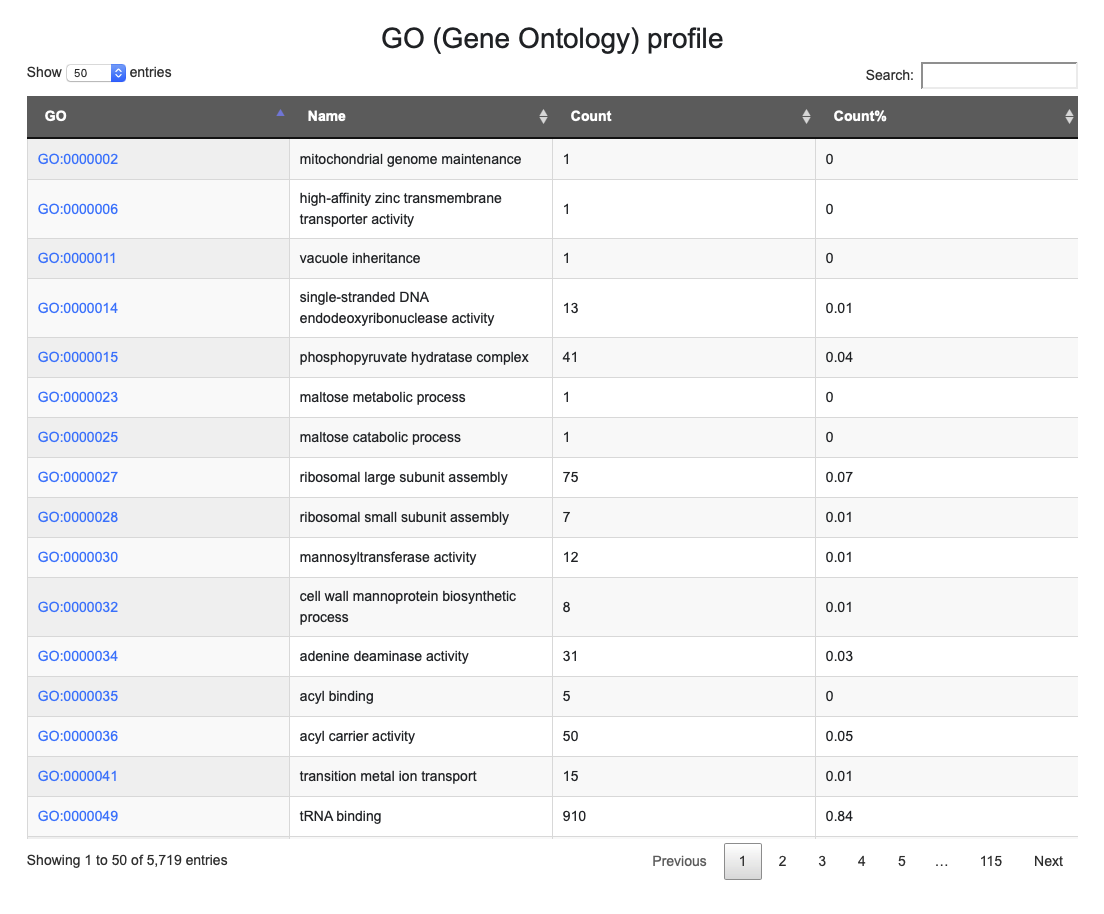
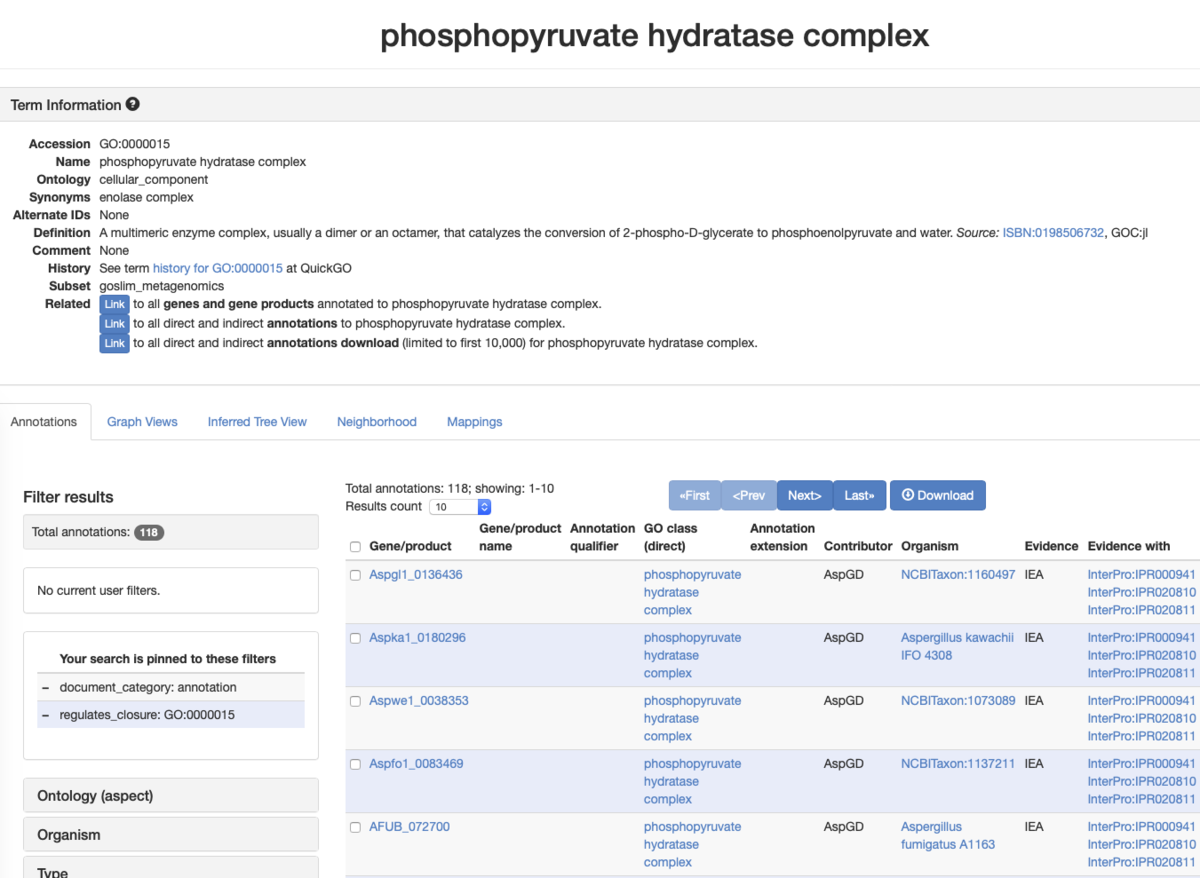
AMiGO2にリンクしている。
- Protein family distribution: TIGRFAMS
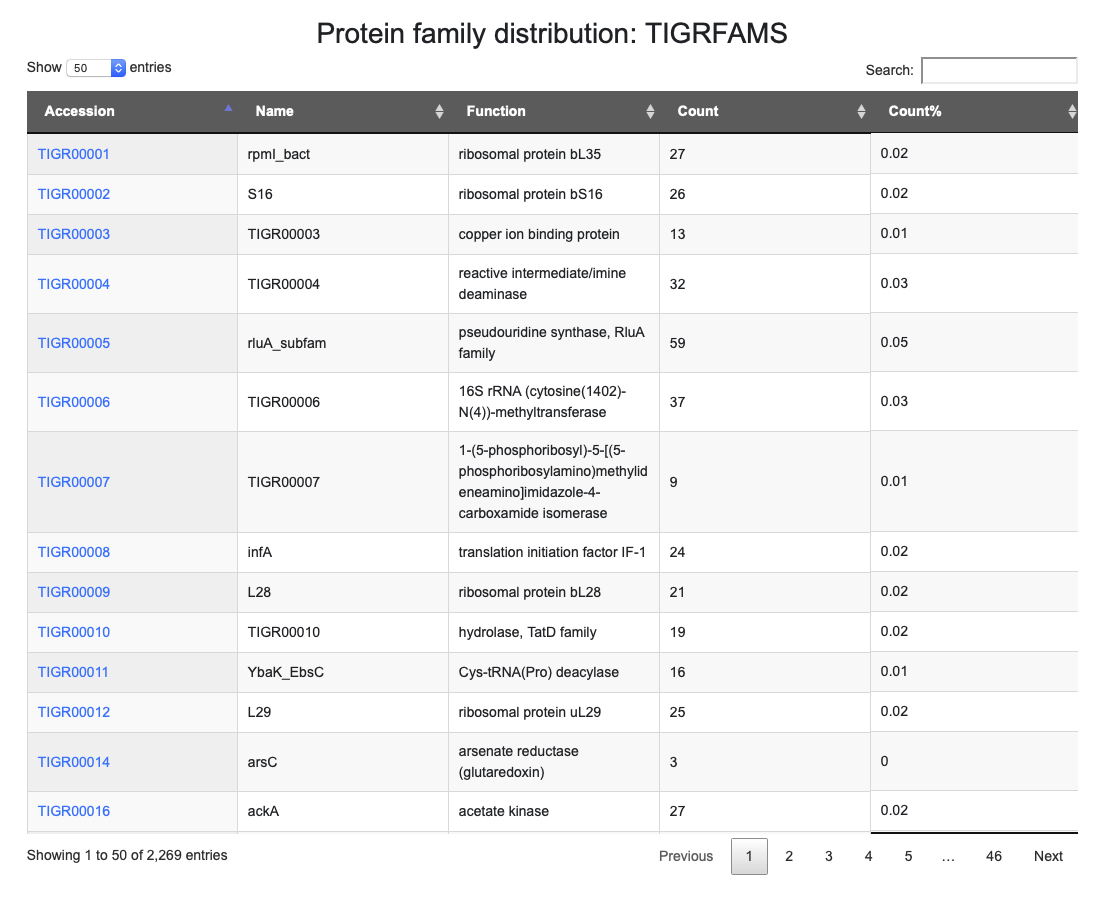

Pathway profiles

- KEGG pathway

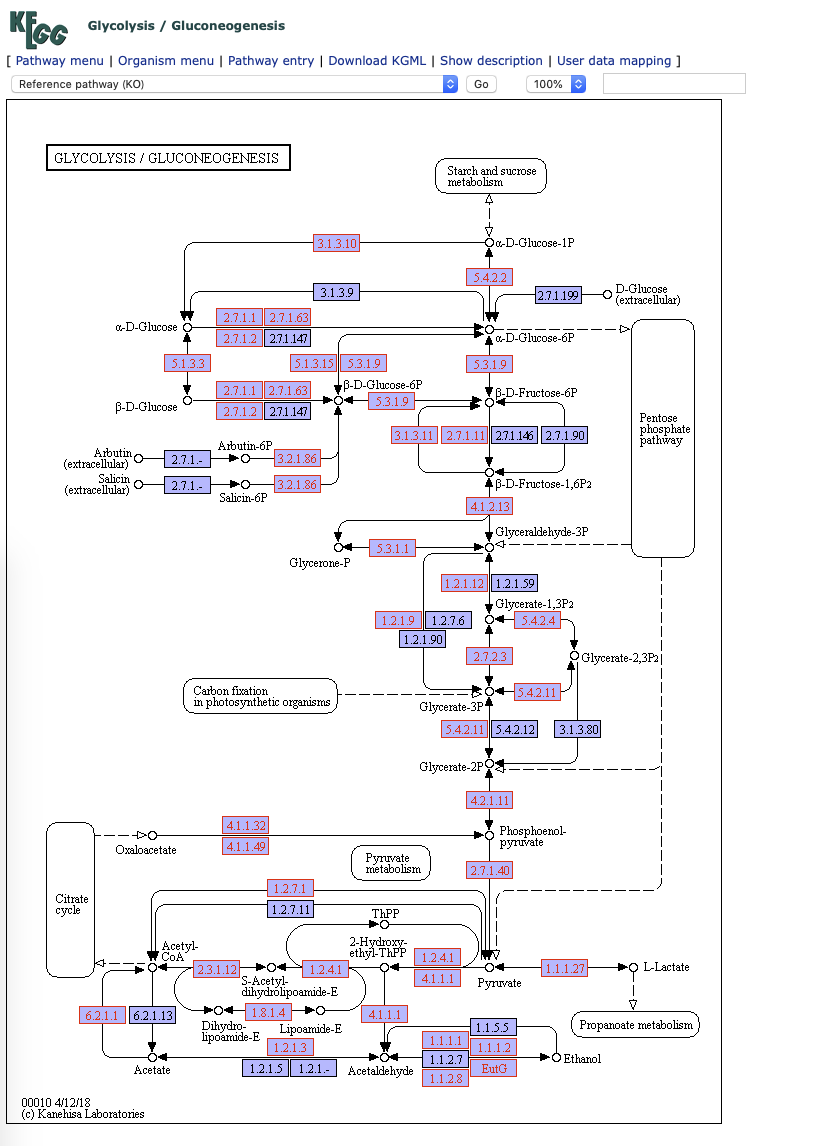
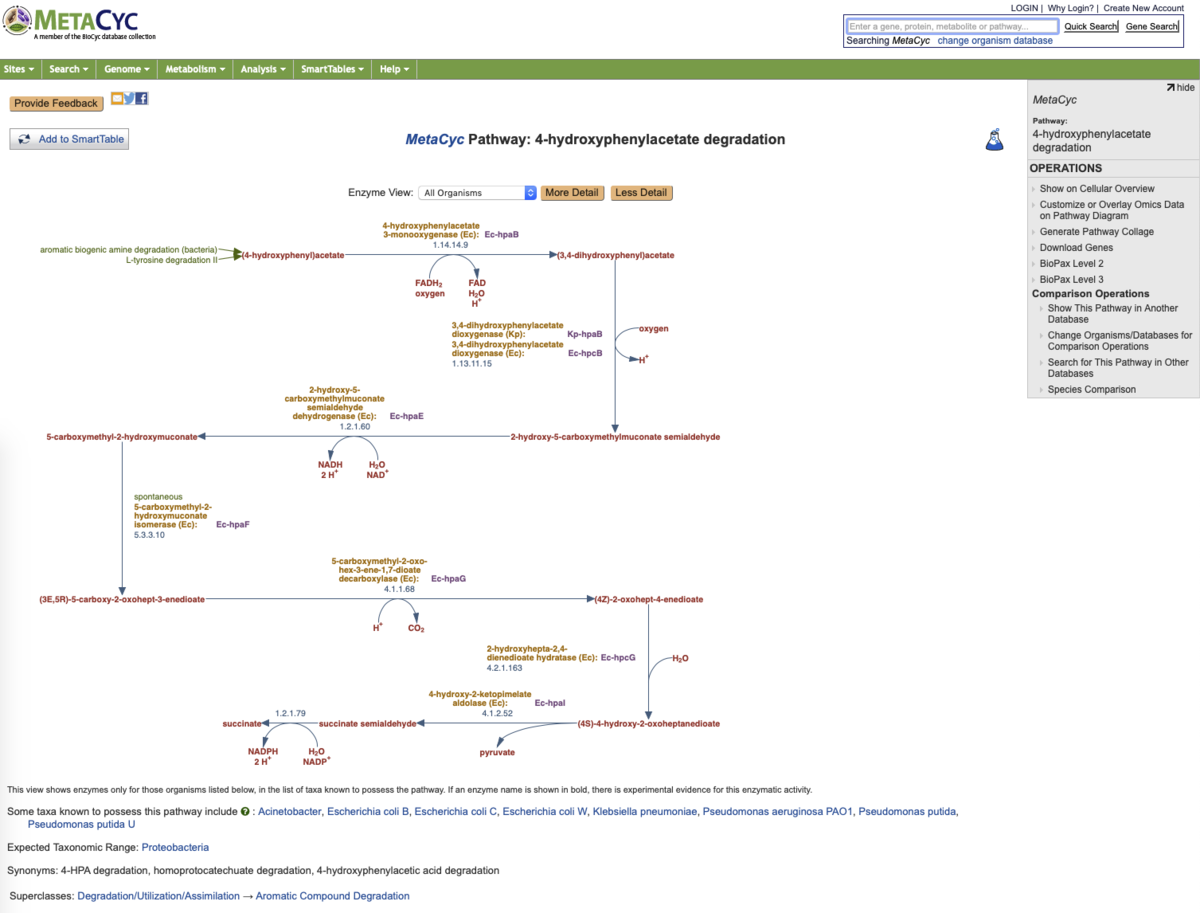
- MetaCyc Pathway
MinPath Predicted MetaCyc Pathway

MetaCyc AROMATIC-COMPOUNDS-DEGRADATION;
別の方法でbinningして得たcontigのsubsetの情報だけ取り出すスクリプトも含まれている。出力がresult、result/data/にall.gffがあるとして、metagenome assemblyのsubsetがmybin.fastaとすると、docker imageを使う場合は次のように実行。
docker run --shm-size 2g --rm -u $(id -u):$(id -g) -it -v $PWD:/data \
-w /data xiaolidong/docker-metaerg fastaContig2Gff.pl \
-c mybin.fasta -g result/data/all.gff > mybin.gff
このあと、さらにreportファイルを作成する。
binned fastaが複数ファイルから構成される場合、別のスクリプトを使います。Githubで確認して下さい。前もってcontigsを長さでフィルタリングするユーティリティスクリプトの説明もあります。
引用
An Integrated Pipeline for Annotation and Visualization of Metagenomic Contigs
Xiaoli Dong, Marc Strous
Front. Genet., 15 October 2019
*1 -w /dataにしておけば、ログイン後の初期パスが/dataになる。-itv $PWD:/dataを合わせて使うと、各ファイルをフルパス指定する必要はない。cpuスレッド指定のoptionがあるが、指定すると値にかかわらずprocess途中でerrorになるデータがあった。指定せずランした。
*2 ローカルファイルにリンクしているので、出力のディレクトリ構成をいじるとアクセスできなくなる。注意する。
*3 ライセンスが理由でdocker imagesには SignalP とTMHMMが含まれていない。
関連