大規模並列シーケンシング、または次世代シーケンシング(NGS)は、従来のSangerシーケンシングと比較してスループットの大幅な増加と低存在量のバリエーションを検出するこれまでにない能力により、遺伝的バリエーションの探索方法に革命をもたらした。シーケンスベースあたりのコストを大幅に削減しているが、これらの新しい技術はエラーを起こしやすいので、NGSデータから遺伝的変異を識別することは依然としてかなりの挑戦である(ref.1)。これは特にheterogeneousなサンプルで当てはまり、このような場合、variant allelic fractions(VAF、合計リード数に対する変異対立遺伝子を持つシークエンシングリード数の比率)が二倍体ゲノムでの期待値(0%、50%、100%の二倍体遺伝子型についての3つの可能性のある値)から逸脱し、変異対立遺伝子のシーケンシングされたリードが全体のごく一部を占め、バックグラウンドエラー率に近づく。それにもかかわらず、そのようなheterogeneousなセッティングで低VAFをロバストに同定することは非常に有益である。例えば、腫瘍由来の分子フットプリントを同定するために無細胞DNAを分析すること。組織学的に正常な組織における体細胞変異の評価(ref.4)など。
次世代シークエンシングのエラー率は、DNA塩基対間でも、同じDNA位置での複数の塩基変化間でも異なることが知られている(ref.5,6)。 NGSエラーは、鋳型DNAのクオリティ、それに続く断片化、ライブラリー調製、ベースコール、または未処理リードのシーケンシングに続くアラインメント工程に起因する、シーケンシングプロセスの多くのステップに起因している。これらのエラーの中にはサンプル間で一貫して繰り返し発生する傾向があるものもあれば、より予測不可能な外観を持つものもある。 NGSが複数の情報源からのエラーから成っていることの正味の影響は、それらを区別したり訂正したりするのが非常に困難になるということである(ref.7)。この非常に多様なエラーパターンを考慮したバリアント同定は、たとえそれらの存在量が少ない時でさえも、真の配列変異をロバストに検出する我々の能力を改善するかもしれない。現在のほとんどのアルゴリズムは、シーケンスアーチファクトと真の変異を区別するため、サンプル間で独立して適用されるVAFの確率的モデルを使用する(ref.8)。一方、shearwaterMLのような低頻度突然変異を検出するために特別に設計されたメソッドは、サンプル間の誤差については、誤差率に関する事前のしきい値要件の制限がある。
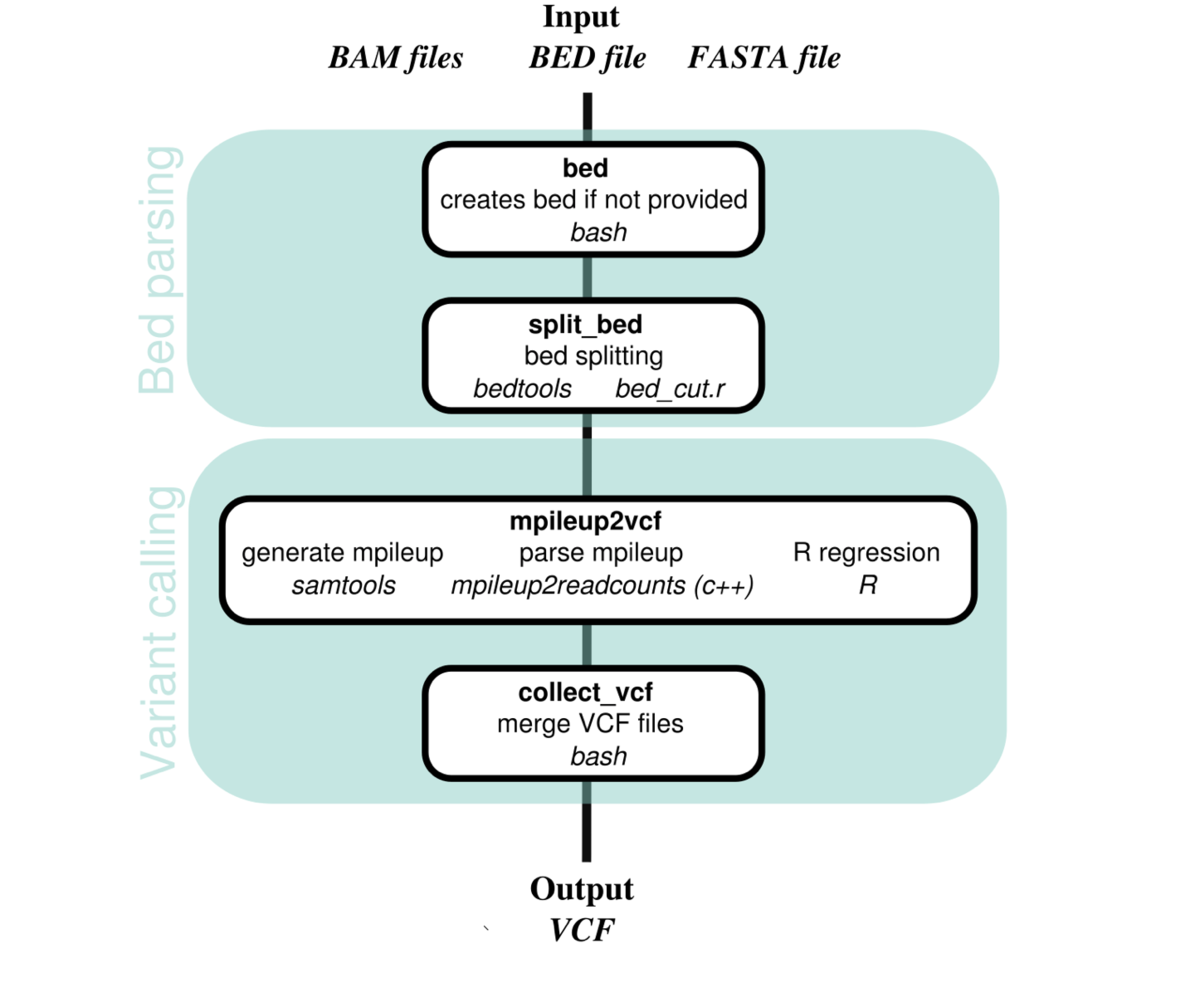
ここでは、同時に分析された複数のサンプルについての、潜在的な塩基の変化のエラーモデルを作成する方法について探索した。変異は、このロバストなエラーモデルに対する外れ値として同定される。 Needlestackと呼ばれるこのメソッドは、NGSデータに見られる可変エラーパターンと比較して動的にシーケンスバリアントを識別することを可能にし、レアバリアントをコールするのに特に適している。この方法をさらなるエラー訂正のための追加の実験室処理(ref.11)およびvery deep シーケンシングと組み合わせることによって、許容できるエラー発見率を維持しながら、1%をはるかに下回るVAFを確実に識別することができる。体細胞変異および生殖細胞変異検出の両方のための方法と複数の厳密な性能評価および比較を行った。ドメイン固有言語(DSL)のnextflow(ref.12)を使用した効率性と堅牢性、およびDockerとSingularityのイメージを提供することによる再現性に重点を置いてパイプラインを展開した。ソースコードはバージョン管理されており、GitHub(https://github.com/IARCbioinfo/needlestack)で無料で入手できる。
Our needlestack variant caller paper is available as a preprint in #bioRxiv
— Tiffany M. Delhomme (@tm_delhomme) May 23, 2019
Thanks to all contributors! including supervisor @m_foll and colleagues @gabriel_aurelie @nl_alcala 1/3 https://t.co/6zLJqjMIcE
Needlestackは、いくつかのサンプルを一緒に分析することでシーケンシングエラー分布を推定してバリアントを正確に同定するのに役立ち得るという考えに基づいた、NGSデータの超高感度なマルチサンプルバリアントコーラーである。circulating free DNAから得られた非常にディープなNGSデータを使用した体細胞変異コールのために最初に開発されたが、WESまたはWGSのような低カバレッジデータにも適用できる。
最近のモダンバリアントコーラー(Mutect2、Strelka2など)のように、needlestackはハプロタイプを決定するためにリードの再構成を実行していないことに注意する。このため、needlestackを実行する前に、ABRA2を使用することが推奨されている。
Githubより転載
インストール
依存
Needlestack works under most Linux distributions and Apple OS X.
そのほかの依存は割愛
Docker Hub
dockerイメージをpullするだけで使える。
docker pull iarcbioinfo/needlestack:latest
> nextflow run iarcbioinfo/needlestack --help
$ nextflow run iarcbioinfo/needlestack --help
N E X T F L O W ~ version 19.04.1
Launching `iarcbioinfo/needlestack` [tiny_tesla] - revision: 8883a4ad65 [master]
--------------------------------------------------------
_ _ _ _
| | | | | | |
_ __ ___ ___ __| | | ___ ___| |_ __ _ ___| | __
| _ \ / _ \/ _ \/ _` | |/ _ \/ __| __/ _` |/ __| |/ /
| | | | __/ __/ (_| | | __/\__ \ || (_| | (__| <
|_| |_|\___|\___|\__,_|_|\___||___/\__\__,_|\___|_|\_\
NEEDLESTACK v1.1: A MULTI-SAMPLE SOMATIC VARIANT CALLER
--------------------------------------------------------
Copyright (C) IARC/WHO
This program comes with ABSOLUTELY NO WARRANTY; for details see LICENSE.txt
This is free software, and you are welcome to redistribute it
under certain conditions; see LICENSE.txt for details.
--------------------------------------------------------
Usage:
nextflow run iarcbioinfo/needlestack [-with-docker] --bed bedfile.bed --input_bams BAM/ --ref reference.fasta --output_vcf VCF_name.vcf [other options]
Mandatory arguments:
--input_bams BAM BAM files directory or text file containing BAM paths in lines.
--ref REF_IN_FASTA Reference genome in fasta format.
--output_vcf VCF FILE NAME Name of the VCF file (output file).
OR
--input_vcf VCF FILE VCF file (basically from GATK pipeline) to annotate.
Options:
--nsplit INTEGER Split the region for calling in nsplit pieces and run in parallel.
--min_dp INTEGER Minimum median coverage (in addition, min_dp in at least 10 samples).
--min_ao INTEGER Minimum number of non-ref reads in at least one sample to consider a site.
--min_af INTEGER Minimum AF in at least one sample to consider a site.
--min_qval VALUE Qvalue in Phred scale to consider a variant.
--sb_type SOR or RVSB Strand bias measure.
--sb_snv VALUE Strand bias threshold for SNVs.
--sb_indel VALUE Strand bias threshold for indels.
--power_min_af VALUE Minimum allelic fraction for power computations.
--sigma_normal VALUE Sigma parameter for negative binomial modeling germline mutations.
--map_qual VALUE Samtools minimum mapping quality.
--base_qual VALUE Samtools minimum base quality.
--max_dp INTEGER Samtools maximum coverage before downsampling.
--output_folder OUTPUT FOLDER Output directory, by default input bam folder.
--bed BED FILE A BED file for calling.
--region CHR:START-END A region for calling.
--tn_pairs TEXT FILE A tab-delimited file containing two columns (normal and tumor sample name) for each sample in line.
--genome_release VALUE Reference genome for alignments plot
--plots VALUE Output PDF regression plots.
--min_af_extra_rob VALUE Minimum allelic fraction to exclude a sample at a position for extra-robust regression.
--min_prop_extra_rob VALUE Minimum proportion of samples having an allelic fraction to be excluded from extra-robust regression.
--mAX_prop_extra_rob VALUE Maximum proportion of samples having an allelic fraction to be excluded from extra-robust regression.
Flags:
--use_file_name Sample names are taken from file names, otherwise extracted from the bam file SM tag.
--all_SNVs Output all SNVs, even when no variant found.
--extra_robust_gl Perform an extra robust regression, basically for germline variants
--do_alignments Add alignment plots.
--no_labels Do not add labels to outliers in regression plots.
--no_indels Do not call indels.
--no_contours Do not add contours to plots and do not plot min(AF)~DP.
テストラン
テストデータを入手する(リンク)。
git clone --depth=1 https://github.com/IARCbioinfo/data_test
cd data_test/
データ
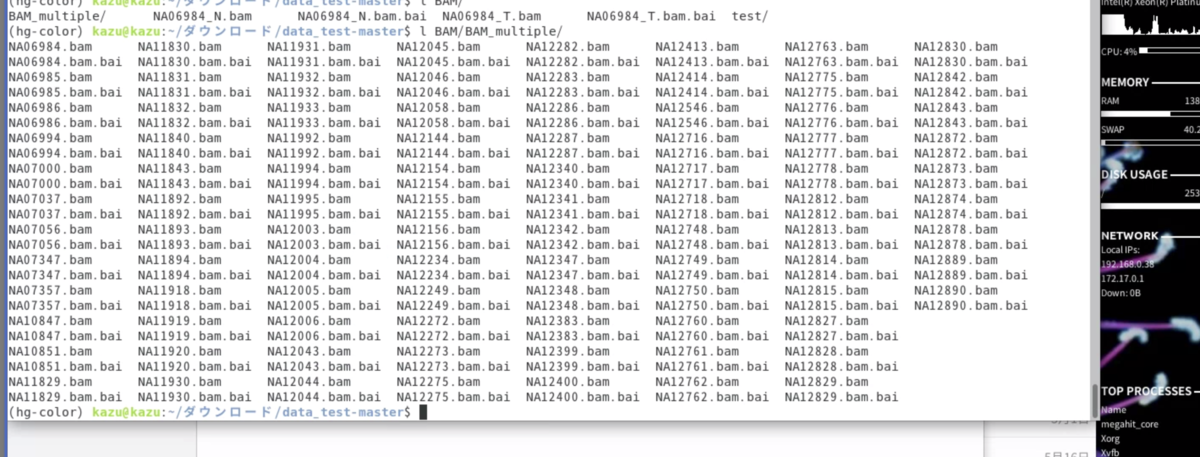
bamとbam.baiは以下のような構造になっている。エラーと区別するため20サンプル以上が推奨されている。
BAMディレクトリ、ターゲットのbed、リファレンス、出力を指定してランする。
nextflow run iarcbioinfo/needlestack -with-docker \
--bed BED/TP53_all.bed \
--input_bams BAM/BAM_multiple/ \
--ref REF/17.fasta \
--output_vcf all_variants.vcf
bedを指定しない場合、reference whole genomeでバリアントがコールされる。
"--do_alignments”フラグが使用されている場合、検出された各バリアントについてpdfが生成される。
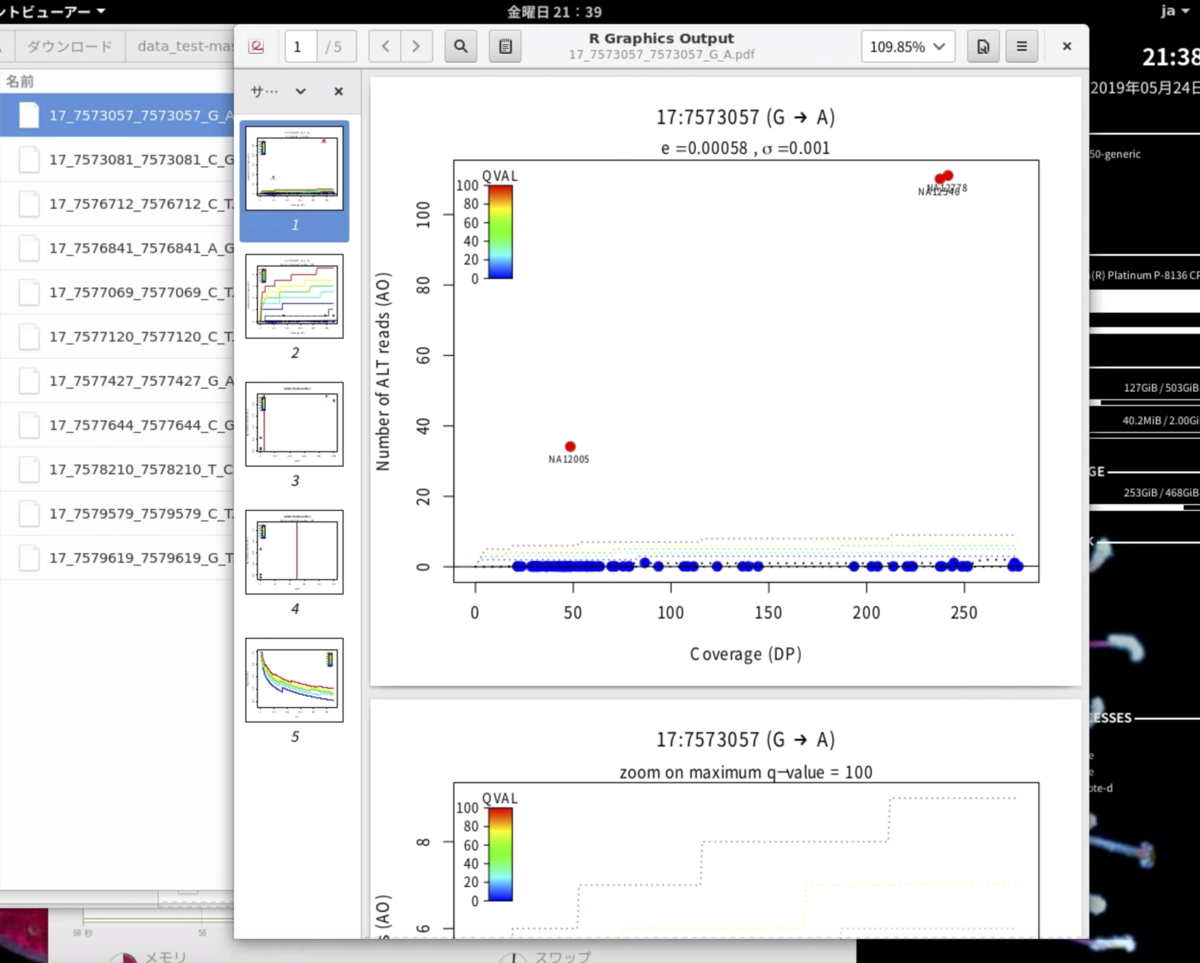
変異が回帰プロットと線形プロットで視覚化される(詳細はGithub参照)。
ランのパラメータが重要です。実際の使用時にはGithubのREAMEも読んでください。Needlestackはmatched tumor/normalの解析にも対応しています。
引用
Needlestack: an ultra-sensitive variant caller for multi-sample next generation sequencing data
Tiffany M. Delhomme, Patrice H. Avogbe, Aurélie Gabriel, Nicolas Alcala, Noemie Leblay, Catherine Voegele, Maxime Vallée, Priscilia Chopard, Amélie Chabrier, Behnoush Abedi-Ardekani, Valérie Gaborieau, Ivana Holcatova, Vladimir Janout, Lenka Foretová, Sasa Milosavljevic, David Zaridze, Anush Mukeriya, Elisabeth Brambilla, Paul Brennan, Ghislaine Scelo, Lynnette Fernandez-Cuesta, Graham Byrnes, Florence Le Calvez-Kelm, James D. McKay, Matthieu Foll
bioRxiv preprint first posted online May. 21, 2019
関連