2021 2/7 タイトル修正
2021 4/24,26 追記
次世代シーケンシングにより、シングルセルシーケンシングまたはメタゲノムシーケンシングテクノロジーを使用して、微生物環境のシーケンシングができる。しかし、両方の技術には、ゲノムの異なる領域または異なる種のゲノムのシーケンシングデプスが非常に不均一であるという問題がある。ほとんどの既存のゲノムアセンブラーは、通常、シーケンスの深さが均一であるという仮定を持っている。これらのアセンブラは、正しい長いコンティグを構築できない。
単一セルシーケンスまたは不均一なシーケンス深度を持つメタゲノムシーケンステクノロジーのリードをアセンブルするためのde Bruijnグラフアプローチに基づくIDBA-UDアルゴリズムを紹介する。問題に取り組むために、いくつかの重要な技術が採用されている。単純なしきい値を使用する代わりに、multiple depthrelative thresholdsを使用して、低デプス領域と高デプス領域の両方で誤ったk-merを削除する。ペアエンド情報を使用したローカルアセンブリの手法は、低デプスの短い繰り返し領域の分岐問題を解決するために使用される。プロセスを高速化するために、エラー修正ステップを実行して、信頼性の高いコンティグに位置合わせできる高デプス領域のリードを修正する。 IDBA-UDと既存のアセンブラ(Velvet、Velvet-SC、SOAPdenovo、Meta-IDBA)のさまざまなデータセットのパフォーマンスを比較すると、IDBA-UDはより長いコンティグをより高い精度で再構築できることがわかる。
IDBAは、第2世代のシーケンスリード用の基本的な反復de Bruijnグラフアセンブラである。 IDBAの拡張であるIDBA-UDは、ペアエンドリードを使用して低デプス領域をアセンブルし、コンティグのプログレッシブデプスを使用して高深度領域のエラーを減らすように設計されている。 これは汎用のアセンブラであり、特にシングルセルおよびメタゲノムシーケンスデータに適している。 IDBA-HybridはIDBA-UDの別の更新バージョンであり、リファレンスゲノムを利用してアセンブリ結果を改善できる。 IDBA-Tranは、RNA-Seqデータ用の反復de Bruijnグラフアセンブラである。

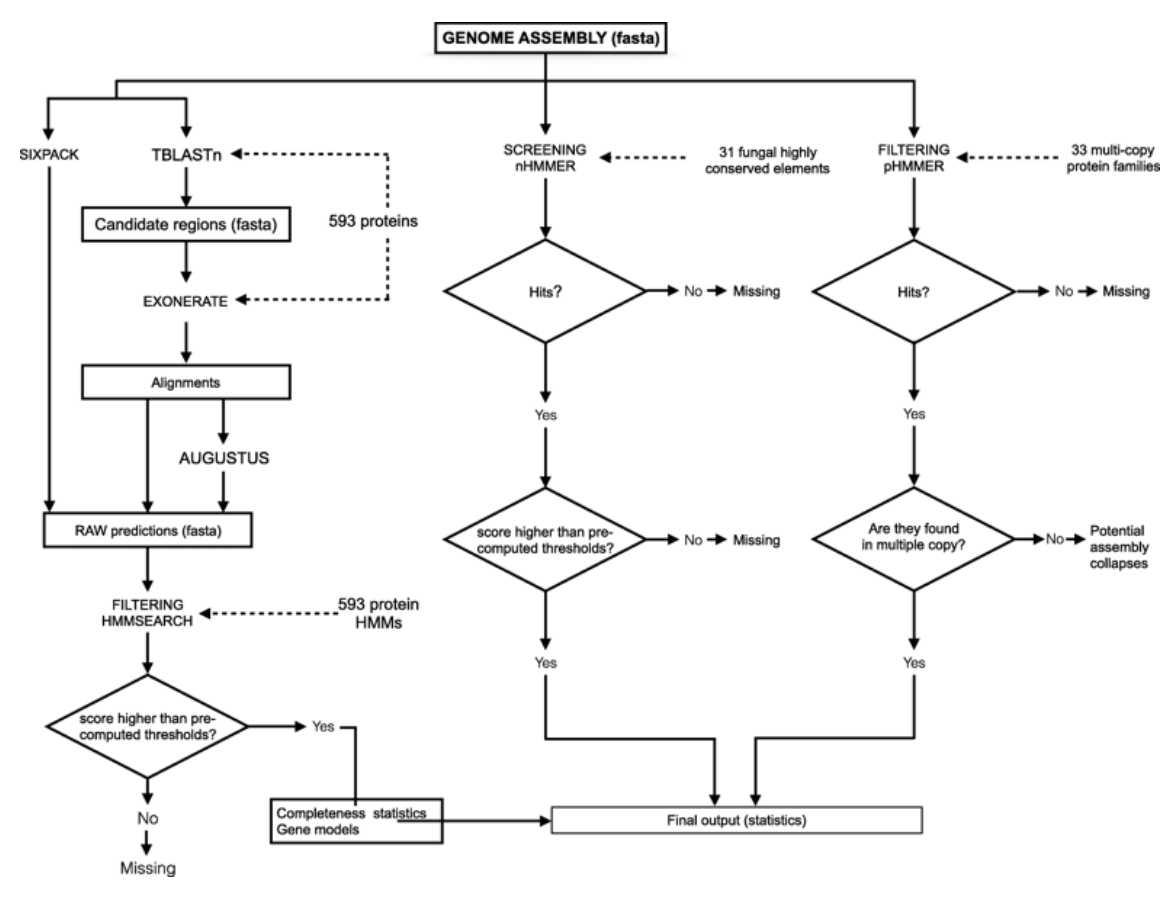
Flowchart of IDBA-UD. 論文より転載
HP
https://i.cs.hku.hk/~alse/hkubrg/projects/idba_ud/
インストール
macos10.14のminiconda3-4.0.5環境でテストした。
本体 Github
#bioconda (link)
conda install -c bioconda -y idba
> idba_ud
$ idba_ud
not enough parameters
IDBA-UD - Iterative de Bruijn Graph Assembler for sequencing data with highly uneven depth.
Usage: idba_ud -r read.fa -o output_dir
Allowed Options:
-o, --out arg (=out) output directory
-r, --read arg fasta read file (<=600)
--read_level_2 arg paired-end reads fasta for second level scaffolds
--read_level_3 arg paired-end reads fasta for third level scaffolds
--read_level_4 arg paired-end reads fasta for fourth level scaffolds
--read_level_5 arg paired-end reads fasta for fifth level scaffolds
-l, --long_read arg fasta long read file (>600)
--mink arg (=20) minimum k value (<=312)
--maxk arg (=100) maximum k value (<=312)
--step arg (=20) increment of k-mer of each iteration
--inner_mink arg (=10) inner minimum k value
--inner_step arg (=5) inner increment of k-mer
--prefix arg (=3) prefix length used to build sub k-mer table
--min_count arg (=2) minimum multiplicity for filtering k-mer when building the graph
--min_support arg (=1) minimum supoort in each iteration
--num_threads arg (=0) number of threads
--seed_kmer arg (=30) seed kmer size for alignment
--min_contig arg (=200) minimum size of contig
--similar arg (=0.95) similarity for alignment
--max_mismatch arg (=3) max mismatch of error correction
--min_pairs arg (=3) minimum number of pairs
--no_bubble do not merge bubble
--no_local do not use local assembly
--no_coverage do not iterate on coverage
--no_correct do not do correction
--pre_correction perform pre-correction before assembly
> fq2fa
$ fq2fa
not enough parameters
fq2fa - Convert Fastq sequences to Fasta sequences.
Usage: fq2fa tmp.fq tmp.fa [...]
fq2fa --paired tmp.fq tmp.fa
fq2fa --merge tmp_1.fq tmp_2.fq tmp.fa
Allowed Options:
--paired if the reads are paired-end in one file
--merge if the reads are paired-end in two files
--filter filter out reads containing 'N'
実行方法
1、fastqはマージして1つの"FASTA"として与える必要があるため、IDBAのラン前にペアエンドfastqをマージする。このコマンドではgzip圧縮fastqは受け付けない。解凍してから指定する。
fq2fa --merge --filter pair_1.fq pair_2.fq read.fa
read.faができる。
2、IDBAのラン
idba_ud -r read.fa -o out_dir
出力
k-merを変えながら繰り返しローカルアセンブリが行われ、最後にscaffoldingされる。

scaffold.faの配列には”N”が含まれる可能性がある。出力についてはこちらも参照(link)。
32スレッド指定、k値は20から120まで10ずつ増やす。 最低サイズ300bpとする。precorrection実行。
idba_ud -r read.fa --num_threads 32 --pre_correction -o out_dir --mink 20 --maxk 120 --step 10 --min_contig 300
-
--mink arg (=20) minimum k value (<=312)
-
--maxk arg (=100) maximum k value (<=312)
-
--step arg (=20) increment of k-mer of each iteration
-
--pre_correction perform pre-correction before assembly
- --min_contig arg (=200) minimum size of contig
長い配列を指定する。
idba_ud -l sequence.fa --num_threads 24 -o out_dir
- -l fasta long read file (>600)
引用
IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth
Peng Y, Leung HC, Yiu SM, Chin FY
Bioinformatics. 2012 Jun 1;28(11):1420-8
参考
https://www.researchgate.net/post/IDBA-UD_files-which_one_to_use
不明なoptionについてはレポジトリのissuesを確認してください。
https://github.com/loneknightpy/idba/issues
*1
IDBA-UDは、de Bruijn graphベースのアセンブラとしては珍しく、k-mer長kに偶数の値許容している。