2020 4/19 流れを修正
2020 4/21 biom出力とh5pyインストール追記
2020 ステップ2のコマンド修正
微生物群集の機能プロファイルは、通常、包括的なメタゲノムやメタトランスクリプトーム配列の検索を用いて作成されるが、これらの検索は時間がかかり、偽のマッピングが発生しやすく、多くの場合、群集レベルでの定量化には限界がある。著者らは、HUMAnN2を開発した。HUMAnN2は、宿主関連コミュニティと環境コミュニティの高速、正確、種分解機能プロファイリングを可能にする階層化された検索戦略である。HUMAnN2は、コミュニティの既知の種を同定し、そのパンゲノムにリードをアラインメントし、分類されていないリードの翻訳検索を行い、最終的に遺伝子ファミリーとパスウェイを定量化する。純粋な翻訳検索と比較して、HUMAnN2は高速で、より正確な遺伝子ファミリープロファイルを生成する。海洋の代謝における連鎖的変動、ヒト微生物パスウェイ間の生態学的貢献パターン、種のゲノム対転写の変動、株のプロファイリングを研究するためにHUMAnN2を適用した。さらに、異なる微生物群集タイプにまたがる生態学的コミュニティのパターンを説明するために「contributional diversity」を導入する。
HUMAnN 2.0は、メタゲノムまたはメタトランスクリプトームシーケンシングデータから、効率的かつ正確にコミュニティ内の微生物パスウェイの豊富さをプロファイリングするためのパイプラインである。このプロセスは機能プロファイリングと呼ばれ、微生物群集とそのメンバーの代謝ポテンシャルを記述することを目的としている。一般的に機能的プロファイリングは、「私の関心のあるコミュニティの微生物は何をしているのか」という質問に答えるものである。
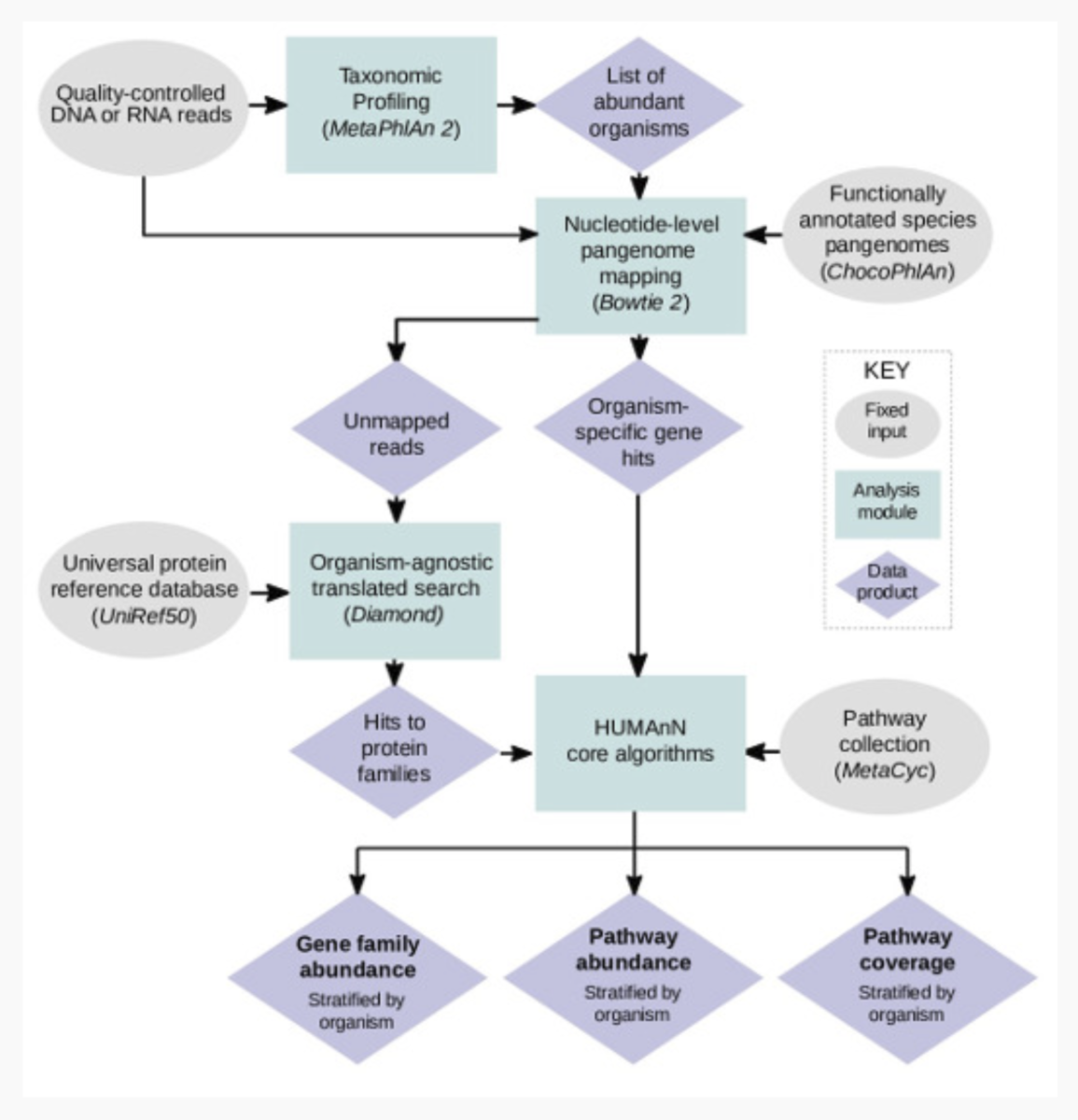
HUMAnN2の流れ。HPより転載。
詳細は論文のONLINE METHODSセクションを読んでください。
huttenhower Lab HP
https://huttenhower.sph.harvard.edu
humann2 HP
https://huttenhower.sph.harvard.edu/humann
Tutorial
https://bitbucket.org/biobakery/biobakery/wiki/humann2
インストール
condaでpython2.7環境を作って導入したがテストでエラーを吐いた(macos10.14)。公式dockerイメージを使ってテストした。
依存
- MetaPhlAn 2.0
- Bowtie2 (version >= 2.1) (see NOTE)
- Diamond (0.9.0 > version >= 0.8.22) (see NOTE)
- MinPath (see NOTE)
- Python (version >= 2.7)
- Memory (>= 16 GB)
- Disk space (>= 10 GB [to accommodate comprehensive sequence databases])
- Operating system (Linux or Mac)
#bioconda (link) 依存が多いため仮想環境が望ましい
conda create -n humann2 -y python=2.7
conda activate humann2
conda install -c bioconda -y humann2
#biom出力するならh5pyも必要
conda install -c anaconda -y h5py
#ツールの互換性の問題であえて古いバージョンを入れる
conda create -n humann2_0.9.9 -y python=2.7
conda activate humann2_0.9.9
conda install -c bioconda -y humann2==0.9.9
#公式docker image (dockerhub)
docker pull biobakery/humann2:2.8.2_cloud
#run
docker run -itv $PWD:/data/ biobakery/humann2:2.8.2_cloud
#biom出力するならh5pyも必要
pip install h5py
> humann2 -h
$ humann2 -h
usage: humann2 [-h] [--version] [-v] [-r] [--bypass-prescreen]
[--bypass-nucleotide-index] [--bypass-translated-search]
[--bypass-nucleotide-search] -i <input.fastq> -o <output>
[--nucleotide-database <nucleotide_database>]
[--annotation-gene-index <8>]
[--protein-database <protein_database>] [--evalue <1.0>]
[--search-mode {uniref50,uniref90}] [--metaphlan <metaphlan>]
[--metaphlan-options <metaphlan_options>]
[--o-log <sample.log>]
[--log-level {DEBUG,INFO,WARNING,ERROR,CRITICAL}]
[--remove-temp-output] [--threads <1>]
[--prescreen-threshold <0.01>] [--identity-threshold <50.0>]
[--translated-subject-coverage-threshold <50.0>]
[--translated-query-coverage-threshold <90.0>]
[--bowtie2 <bowtie2>] [--usearch <usearch>]
[--rapsearch <rapsearch>] [--diamond <diamond>]
[--taxonomic-profile <taxonomic_profile.tsv>]
[--id-mapping <id_mapping.tsv>]
[--translated-alignment {usearch,rapsearch,diamond}]
[--xipe {on,off}] [--minpath {on,off}] [--pick-frames {on,off}]
[--gap-fill {on,off}] [--output-format {tsv,biom}]
[--output-max-decimals <10>] [--output-basename <sample_name>]
[--remove-stratified-output]
[--remove-column-description-output]
[--input-format {fastq,fastq.gz,fasta,fasta.gz,sam,bam,blastm8,genetable,biom}]
[--pathways-database <pathways_database.tsv>]
[--pathways {metacyc,unipathway}]
[--memory-use {minimum,maximum}]
HUMAnN2 : HMP Unified Metabolic Analysis Network 2
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
-v, --verbose additional output is printed
-r, --resume bypass commands if the output files exist
--bypass-prescreen bypass the prescreen step and run on the full ChocoPhlAn database
--bypass-nucleotide-index
bypass the nucleotide index step and run on the indexed ChocoPhlAn database
--bypass-translated-search
bypass the translated search step
--bypass-nucleotide-search
bypass the nucleotide search steps
-i <input.fastq>, --input <input.fastq>
input file of type {fastq,fastq.gz,fasta,fasta.gz,sam,bam,blastm8,genetable,biom}
[REQUIRED]
-o <output>, --output <output>
directory to write output files
[REQUIRED]
--nucleotide-database <nucleotide_database>
directory containing the nucleotide database
[DEFAULT: /Users/kazu/anaconda3/envs/metaphylan2/lib/python2.7/site-packages/humann2/data/chocophlan_DEMO]
--annotation-gene-index <8>
the index of the gene in the sequence annotation
[DEFAULT: 8]
--protein-database <protein_database>
directory containing the protein database
[DEFAULT: /Users/kazu/anaconda3/envs/metaphylan2/lib/python2.7/site-packages/humann2/data/uniref_DEMO]
--evalue <1.0> the evalue threshold to use with the translated search
[DEFAULT: 1.0]
--search-mode {uniref50,uniref90}
search for uniref50 or uniref90 gene families
[DEFAULT: based on translated database selected]
--metaphlan <metaphlan>
directory containing the MetaPhlAn software
[DEFAULT: $PATH]
--metaphlan-options <metaphlan_options>
options to be provided to the MetaPhlAn software
[DEFAULT: "-t rel_ab"]
--o-log <sample.log> log file
[DEFAULT: temp/sample.log]
--log-level {DEBUG,INFO,WARNING,ERROR,CRITICAL}
level of messages to display in log
[DEFAULT: DEBUG]
--remove-temp-output remove temp output files
[DEFAULT: temp files are not removed]
--threads <1> number of threads/processes
[DEFAULT: 1]
--prescreen-threshold <0.01>
minimum percentage of reads matching a species
[DEFAULT: 0.01]
--identity-threshold <50.0>
identity threshold for alignments
[DEFAULT: 50.0]
--translated-subject-coverage-threshold <50.0>
subject coverage threshold for translated alignments
[DEFAULT: 50.0]
--translated-query-coverage-threshold <90.0>
query coverage threshold for translated alignments
[DEFAULT: 90.0]
--bowtie2 <bowtie2> directory containing the bowtie2 executable
[DEFAULT: $PATH]
--usearch <usearch> directory containing the usearch executable
[DEFAULT: $PATH]
--rapsearch <rapsearch>
directory containing the rapsearch executable
[DEFAULT: $PATH]
--diamond <diamond> directory containing the diamond executable
[DEFAULT: $PATH]
--taxonomic-profile <taxonomic_profile.tsv>
a taxonomic profile (the output file created by metaphlan)
[DEFAULT: file will be created]
--id-mapping <id_mapping.tsv>
id mapping file for alignments
[DEFAULT: alignment reference used]
--translated-alignment {usearch,rapsearch,diamond}
software to use for translated alignment
[DEFAULT: diamond]
--xipe {on,off} turn on/off the xipe computation
[DEFAULT: off]
--minpath {on,off} turn on/off the minpath computation
[DEFAULT: on]
--pick-frames {on,off}
turn on/off the pick_frames computation
[DEFAULT: off]
--gap-fill {on,off} turn on/off the gap fill computation
[DEFAULT: on]
--output-format {tsv,biom}
the format of the output files
[DEFAULT: tsv]
--output-max-decimals <10>
the number of decimals to output
[DEFAULT: 10]
--output-basename <sample_name>
the basename for the output files
[DEFAULT: input file basename]
--remove-stratified-output
remove stratification from output
[DEFAULT: output is stratified]
--remove-column-description-output
remove the description in the output column
[DEFAULT: output column includes description]
--input-format {fastq,fastq.gz,fasta,fasta.gz,sam,bam,blastm8,genetable,biom}
the format of the input file
[DEFAULT: format identified by software]
--pathways-database <pathways_database.tsv>
mapping file (or files, at most two in a comma-delimited list) to use for pathway computations
[DEFAULT: metacyc database ]
--pathways {metacyc,unipathway}
the database to use for pathway computations
[DEFAULT: metacyc]
--memory-use {minimum,maximum}
the amount of memory to use
[DEFAULT: minimum]
> humann2_databases -h
# humann2_databases -h
usage: humann2_databases [-h] [--available]
[--download <database> <build> <install_location>]
[--update-config {yes,no}]
[--database-location DATABASE_LOCATION]
HUMAnN2 Databases
optional arguments:
-h, --help show this help message and exit
--available print the available databases
--download <database> <build> <install_location>
download the selected database to the install location
--update-config {yes,no}
update the config file to set the new database as the default [DEFAULT: yes]
--database-location DATABASE_LOCATION
location (local or remote) to pull the database
> humann2_rename_table -h
# humann2_rename_table -h
usage: humann2_rename_table [-h] [-i INPUT]
[-n {pfam,eggnog,metacyc-pwy,ec,metacyc-rxn,go,kegg-pathway,kegg-module,kegg-orthology,infogo1000}]
[-c CUSTOM] [-s] [-o OUTPUT]
HUMAnN2 utility for renaming table features
===========================================
For additional name mapping files, run the following command:
$ humann2_databases --download utility_mapping full $DIR
Replacing, $DIR with the directory to download and install the databases.
optional arguments:
-h, --help show this help message and exit
-i INPUT, --input INPUT
Original output table (tsv or biom format); default=[TSV/STDIN]
-n {pfam,eggnog,metacyc-pwy,ec,metacyc-rxn,go,kegg-pathway,kegg-module,kegg-orthology,infogo1000}, --names {pfam,eggnog,metacyc-pwy,ec,metacyc-rxn,go,kegg-pathway,kegg-module,kegg-orthology,infogo1000}
Table features that can be renamed with included data files
-c CUSTOM, --custom CUSTOM
Custom mapping of feature IDs to full names (.tsv or .tsv.gz)
-s, --simplify Remove non-alphanumeric characters from names
-o OUTPUT, --output OUTPUT
Path for modified output table; default=[STDOUT]
> humann2_renorm_table -h
# humann2_renorm_table -h
usage: humann2_renorm_table [-h] [-i INPUT] [-u {cpm,relab}]
[-m {community,levelwise}] [-s {y,n}] [-p]
[-o OUTPUT]
HUMAnN2 utility for renormalizing TSV files
===========================================
Each level of a stratified table will be
normalized using the desired scheme.
optional arguments:
-h, --help show this help message and exit
-i INPUT, --input INPUT
Original output table (tsv or biom format); default=[TSV/STDIN]
-u {cpm,relab}, --units {cpm,relab}
Normalization scheme: copies per million [cpm], relative abundance [relab]; default=[cpm]
-m {community,levelwise}, --mode {community,levelwise}
Normalize all levels by [community] total or [levelwise] totals; default=[community]
-s {y,n}, --special {y,n}
Include the special features UNMAPPED, UNINTEGRATED, and UNGROUPED; default=[y]
-p, --update-snames Update '-RPK' in sample names to appropriate suffix; default=off
-o OUTPUT, --output OUTPUT
Path for modified output table; default=[STDOUT]
> humann2_join_tables -h
# humann2_join_tables -h
usage: humann2_join_tables [-h] [-v] -i INPUT -o OUTPUT
[--file_name FILE_NAME] [-s]
Join gene, pathway, or taxonomy tables
optional arguments:
-h, --help show this help message and exit
-v, --verbose additional output is printed
-i INPUT, --input INPUT
the directory of tables
-o OUTPUT, --output OUTPUT
the table to write
--file_name FILE_NAME
only join tables with this string included in the file name
-s, --search-subdirectories
search sub-directories of input folder for files
> humann2_barplot -h
humann2_barplot
usage: humann2_barplot [-h] -i <input table> [-f <feature id>] [-t <int>]
[-s <sorting methods> [<sorting methods> ...]]
[-l <feature>] [-m <feature>] [-c <colormap>]
[-k <colormap>] [-x] [-o <file.ext>] [-a <choice>] [-g]
[-r] [-z] [-w <int>] [-d <size> <size>]
[-y <limit> <limit>] [-e]
humann2_barplot: error: argument -i/--input is required
(metaphylan2) kamisakakazumanoMac-mini:human-unmap kazu$ humann2_barplot -h
usage: humann2_barplot [-h] -i <input table> [-f <feature id>] [-t <int>]
[-s <sorting methods> [<sorting methods> ...]]
[-l <feature>] [-m <feature>] [-c <colormap>]
[-k <colormap>] [-x] [-o <file.ext>] [-a <choice>] [-g]
[-r] [-z] [-w <int>] [-d <size> <size>]
[-y <limit> <limit>] [-e]
HUMAnN2 plotting tool
optional arguments:
-h, --help show this help message and exit
-i <input table>, --input <input table>
HUMAnN2 table with optional metadata
-f <feature id>, --focal-feature <feature id>
Feature ID of interest (give ID not full name)
-t <int>, --top-strata <int>
Number of top stratifications to highlight (top = highest grand means)
-s <sorting methods> [<sorting methods> ...], --sort <sorting methods> [<sorting methods> ...]
Sample sorting methods (can use more than one; will evaluate in order)
none : Default
sum : Sum of stratified values
dominant : Value of the most dominant stratification
similarity : Bray-Curtis agreement of relative stratifications
usimilarity : Bray-Curtis agreement of raw stratifications
metadata : Given metadata label
-l <feature>, --last-metadatum <feature>
Indicate end of metadata rows
-m <feature>, --focal-metadatum <feature>
Indicate metadatum to highlight / group by
-c <colormap>, --colormap <colormap>
Color space for stratifications
-k <colormap>, --meta-colormap <colormap>
Color space for metadata levels
-x, --exclude-unclassified
Do not include the 'unclassified' stratum
-o <file.ext>, --output <file.ext>
Where to save the figure
-a <choice>, --scaling <choice>
Scaling options for total bar heights (strata are always proportional to height)
none : Default
pseudolog : Total bar heights log-scaled (strata are NOT log scaled)
normalize : Bars all have height=1 (highlighting relative attribution)
-g, --as-genera Collapse species to genera
-r, --grid Add y-axis grid
-z, --remove-zeroes Do not plot samples with zero sum for this feature
-w <int>, --width <int>
Relative width of the plot vs. legend (default: 5)
-d <size> <size>, --dimensions <size> <size>
Image height and width in inches (default: 8 4)
-y <limit> <limit>, --ylims <limit> <limit>
Fix limits for y-axis
-e , --legend-stretch
Stretch/compress legend elements
conda install -c bioconda -y humann2==0.9.9
> humann2_rename_table -h
# humann2_rename_table -h
usage: humann2_rename_table [-h] [-i INPUT]
[-n {infogo1000,metacyc-rxn,uniref90,kegg-module,ec,go,metacyc-pwy,pfam,eggnog,uniref50,kegg-pathway,kegg-orthology}]
[-c CUSTOM] [-s] [-o OUTPUT]
HUMAnN2 utility for renaming table features
===========================================
optional arguments:
-h, --help show this help message and exit
-i INPUT, --input INPUT
Original output table (tsv or biom format); default=[TSV/STDIN]
-n {infogo1000,metacyc-rxn,uniref90,kegg-module,ec,go,metacyc-pwy,pfam,eggnog,uniref50,kegg-pathway,kegg-orthology}, --names {infogo1000,metacyc-rxn,uniref90,kegg-module,ec,go,metacyc-pwy,pfam,eggnog,uniref50,kegg-pathway,kegg-orthology}
Table features that can be renamed with included data files
-c CUSTOM, --custom CUSTOM
Custom mapping of feature IDs to full names (.tsv or .tsv.gz)
-s, --simplify Remove non-alphanumeric characters from names
-o OUTPUT, --output OUTPUT
Path for modified output table; default=[STDOUT]
動作チェック
humann2_test
#run also tool tests
humann2_test --run-functional-tests-end-to-end
データベースの準備
#現在ダウンロード可能なデータベースの確認
humann2_databases --available
#ChocoPhlAn database(5.6GB)
humann2_databases --download chocophlan full <path>/<to>/database_dir
#UniRef database (only download one of 1-4)
#1,UniRef90 EC filtered (推奨,846 MB)
humann2_databases --download uniref uniref90_ec_filtered_diamond <path>/<to>/database_dir
#2,full UniRef90 (11 GB)
humann2_databases --download uniref uniref90_diamond <path>/<to>/database_dir
#3,UniRef50 EC filtered (239 MB)
humann2_databases --download uniref uniref50_ec_filtered_diamond <path>/<to>/database_dir
#4,full UniRef50 (4.6 GB)
humann2_databases --download uniref uniref50_diamond <path>/<to>/database_dir
HUMAnN2におけるヌクレオチドレベルの検索は、種のパンゲノムコレクションを用いて行われ、このコレクションを "ChocoPhlAn "と呼ぶ(ChocoPhlAnの以前のバージョンはMetaRef46として発表された。HUMAnN2に組み込まれたChocoPhlAnのバージョンは、MetaPhlAn2とそのマーカーデータベースの基礎となっているものと同一である)。種のパンゲノムは、種のタンパク質コードの潜在的な可能性の非冗長な表現である。ある種のパンゲノムを構築するために、NCBI GenBankおよび/またはRefSeqから、その種の利用可能な単離ゲノムをすべてダウンロードし、関連するコーディング配列(CDS)アノテーションと一緒にダウンロードされた。各単離ゲノムをPhyloPhlAnで解析し、分類学的に正しい配置を確認する。UCLUSTを用いて、与えられた種の高品質の単離株ゲノムから97%のヌクレオチド同一性ですべてのCDSをクラスター化する。各クラスタから1つの代表的な(セントロイド)配列を保存した。これらのセントロイド配列は、種のパンゲノムを構成する。これらのステップはMetaPhlAn2開発の過程で行われた。ChocoPhlAnを機能プロファイリングに使用するために、著者らは各パンゲノムのセントロイド配列をUniRef90とUniRef50を使ってアノテーション付けた。(論文より)
実行方法
1、fastq,(fastq.gz, fasta, or fasta.gz )、マッピングのsamかbam、遺伝子テーブルファイル(TSVかbiom) を指定する。
cat pair1.fq.gz pair2.fq.gz > paired.fq.gz
#またはseqtkでintaerlace.fqを作る
seqtk mergepe pair1.fq.gz pair2.fq.gz > interleaved.fq.gz
#run - tsv出力
humann2 -i paired.fq.gz –o out_dir --threads 12
#run - biom出力。h5pyがないと最後にエラーに吐くので注意。
humann2 -i paired.fq.gz –o out_dir --threads 12 --output-format biom
#databaseのパスを指定してランする
humann2 -i paired.fq.gz –o out_dir --threads 20 \
--nucleotide-database <path>/<to>/chocophlan \
--protein-database <path>/<to>/uniref90
- -i <input.fastq> input file of type {fastq,fastq.gz,fasta,fasta.gz, sam,bam,blastm8,genetable,biom} [REQUIRED]
- -o <output> directory to write output files [REQUIRED]
- --threads number of threads/processes [DEFAULT: 1]
- --output-format {tsv, biom} the format of the output files [DEFAULT: tsv]
- --remove-stratified-output remove stratification from output [DEFAULT: output is stratified]
主要な3つのファイルが出力される。コミュニティ内の各遺伝子ファミリーの豊富さを表したSAMPLE_genefamilies.tsv、コミュニティ内の各パスウェイの豊富さを表したpathabundance.tsv、コミュニティ内の各パスウェイのカバー率を表したpathcoverage.tsvになる。
option; ファイルサイズを減らすために、フィーチャ名はデフォルトでは出力に含まれない。フィーチャー名をアタッチするには、humann2_rename_table スクリプトを使う。
#version0.9.9
humann2_rename_table --input genefamilies.tsv --output genefamilies-names.tsv --names uniref90
#biom
#version0.9.9
humann2_rename_table --input genefamilies.biom --output genefamilies-names.biom --names uniref90
なぜかツールのバージョンによって対応するデータベースが異なってしまっている。例えばdockerイメージ;humann2:2.8.2_cloudではuniref90や50は利用できない。condaでインストールできる最新版はuniref50は利用できる。version0.9.9ならuniref90もuniref50も利用できる( インストールに導入方法は載せています)。 このリネームはステップ4の後に行ってもよい。
2、シーケンシングデプスの異なるサンプル間の比較を容易にするために、遺伝子ファミリーとパスウェイのアバンダンスRPK値を組成単位(100万あたりのコピーまたは相対abundance)に正規化する(パスウェイカバレッジは正規化する必要はない)。
#genefamilies abundanceファイルの場合
#tsv input
humann2_renorm_table --input ggenefamilies-names.tsv --output genefamilies-cpm.tsv --units cpm --update-snames
#biom input
humann2_renorm_table --input genefamilies-names.biom --output genefamilies-cpm.biom --units cpm --update-snames
#pathway abundanceファイルの場合
#tsv input
humann2_renorm_table --input pathabundance-name.tsv --output pathabundance-cpm.tsv --units cpm --update-snames
#biom input
humann2_renorm_table --input pathabundance-name..biom --output pathabundance-cpm.biom --units cpm --update-snames
- --units {cpm, relab} Normalization scheme: copies per million [cpm],
- --update-snames Update '-RPK' in sample names to appropriate suffix; default=off
- --output Path for modified output table; default=[STDOUT]
100万になるようにスケールされるため、元のファイルよりも大きくなる傾向がある。
3、複数の結果をマージする。2の出力の〜merged_genefamilies-cpm.tsvを1つのディレクトリに集め、そのディレクトリを指定する。以下はTSVだがbiomも同様。
#gene
humann2_join_tables -i input_dir -o genefamilies.tsv
4、RPKMに再び正規化
humann2_renorm_table -i genefamilies.tsv -o genefamilies_cpm.tsv --units cpm
5、1−4で同定した、豊富さが有意に異なるパスウェイのプロットを作成する。
#metadataがない場合
humann2_barplot --input genefamilies_cpm.tsv --output plot.png
追記
biom => TSV (biom-format)
biom convert -i genefamilies-cpm.biom -o genefamilies-cpm.tsv --to-tsv
最近Phylophlanが3になりましたが(ツイート)、 humannも3が開発中になっています。
=>しばらく経ってversion3が公開されました。
引用
Species-level functional profiling of metagenomes and metatranscriptomes
Eric A. Franzosa, Lauren J. McIver, Gholamali Rahnavard, Luke R. Thompson, Melanie Schirmer, George Weingart, Karen Schwarzberg Lipson, Rob Knight, J. Gregory Caporaso, Nicola Segata & Curtis Huttenhower
Nature Methods volume 15, pages962–968(2018)
参考
関連























