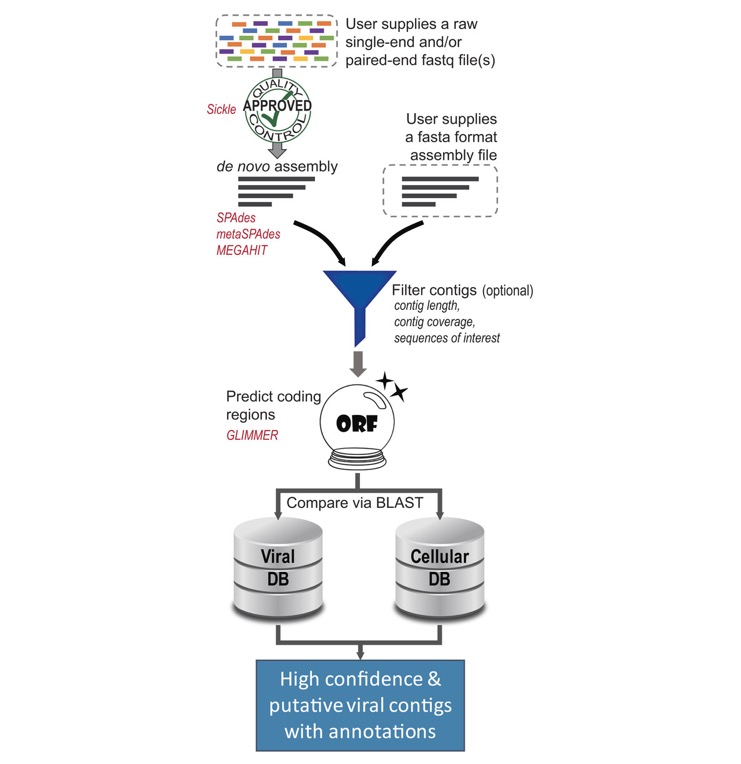
アセンブリはコンティグを生成するが、それ以上の分類学的なプロファイリングや機能解析のためには、OTUに分類することが重要である。このOTUクラスタリングはビニングとも呼ばれる。しかしコンティグの正確なビニングは、ゲノム中のリピート配列、シークエンシングエラー、同じ種内の株間レベル変動などによるキメラアセンブリなどの理由のために依然として困難である。現在利用可能なビニング手法は、分類手法とクラスタリング手法に大別される。分類アプローチ(Classification approaches )はtaxonomy依存であり、すなわち、コンティグまたはリードから有意なtaxonへの割り当てに参照するデータベースが必要である。分類は、配列同一性による相同性(最も一般的な分類学的祖先に割り当てるMEGANなど)、またはオリゴヌクレオチド組成パターンおよび分類学的cladeのようなゲノムシグネチャーに基づく分類(PhyloPythiaやkrakenなど)がある。クラスタリングアプローチは参照データベースまたは分類学情報は必要ない。クラスタリングアプローチは、GC含量、テトラマー組成(論文より Albertsen et al、2013; Chatterji et al、2008; Yang et al、2010)またはInterpolated Markov Models(Kelley and Salzberg、2010)からのコンティグカバレッジプロファイル(Baran and Halperin、2012; Wu and Ye、2011)を利用する。
最近ではコンティグのカバレッジプロファイルを用いてビニングする方法が開発されている。この考え方は、2つのコンティグが同じゲノムに由来する場合、複数のメタゲノムサンプルにわたるそれらのカバレッジプロファイルが高度に相関していることである。この方法は、コンティグプロファイルをテトラマー頻度と統合することによってさらに改善され得る。GroopM(Imelfort et al。、2014)は、視覚化されインタラクティブなパイプライン持ち、ユーザーは専門家の下でビンをマージして分割できる。他方、熟練者の介入がなければ、GroopMの自動ビニング結果はCONCOCTほど満足できるものではない(Alneberg et al、2014)。 CONCOCT(Alneberg et al、2014)は、コンティグをビンにクラスタリングするためにガウス混合モデル(GMM)を利用する。また、CONCOCTは、変分ベイズモデルの選択によって最適なOTU数を自動的に決定する。 MetaBAT(Kang et al、2015)は、対のコンティグについての積分距離を計算し、次いで、modifiled K-medoids algorithmによって反復的にコンティグをクラスター化する。 MaxBin(Wu et al。、2015)は同じゲノム間およびゲノム間の距離の分布を比較している。
COCACOLAはシーケンスの構成、カバレッジ、Co-alignment、および複数のサンプルにわたるペアエンドリンケージを組み込んでいる。デフォルトでは、COCACOLAは、複数のサンプルにわたって配列合成とカバレッジを使用してビニングを実行する。CONCOCT、GroopM、MaxBin、MetaBATと比較されており、Precision、recallでよりよい成績を出し、また、スケーラブルかつ高速であると示されている。
インストール
ubuntu16.0.4にて、conda createでpython2.7.15環境を作ってテストした。
本体 Github
ここではPythonバージョンをインストールする。
#conda createで仮想環境を作る
conda create -n cocacola_env -y python=2.7.14
source activate cocacola_env
#他の依存
conda install -y numpy scipy pandas scikit-learn cvxopt
python版COCACOLAはdropboxからダウンロードする。
link => here
docker imageもある。
docker pull bilalarxd/cocacola
> python COCACOLA-python/cocacola.py
# python COCACOLA-python/cocacola.py
usage: cocacola.py [-h] [--contig_file CONTIG_FILE]
[--abundance_profiles ABUNDANCE_PROFILES]
[--composition_profiles COMPOSITION_PROFILES]
[--edge_list EDGE_LIST] [--output OUTPUT]
[--clusters CLUSTERS]
cocacola.py: error: Data is missing, add file(s) using --contig_file <contig_file> and/or --abundance_profiles <abund_profiles> and/or --composition_profiles <comp_profiles>
(cocacola_env) root@c875fa54ab81:/data# python COCACOLA-python/cocacola.py -h
usage: cocacola.py [-h] [--contig_file CONTIG_FILE]
[--abundance_profiles ABUNDANCE_PROFILES]
[--composition_profiles COMPOSITION_PROFILES]
[--edge_list EDGE_LIST] [--output OUTPUT]
[--clusters CLUSTERS]
optional arguments:
-h, --help show this help message and exit
--contig_file CONTIG_FILE
The contigs file.
--abundance_profiles ABUNDANCE_PROFILES
The abundance profiles, containing a table where each
row correspond to a contig, and each column correspond
to a sample. All values are separated with tabs.
--composition_profiles COMPOSITION_PROFILES
The composition profiles, containing a table where
each row correspond to a contig, and each column
correspond to the kmer composition of particular kmer.
All values are separated with comma.
--edge_list EDGE_LIST
The edges encoding either the co-alignment or the
pair-end linkage information, one row for one edge in
the format: contig_name_A contig_name_B weight. The
edge is undirected.
--output OUTPUT The output file, storing the binning result. If not
specified, the result is displayed directly on the
console.
--clusters CLUSTERS specify the number of clusters. If not specified, the
cluster number is estimated by single-copy genes.
テストラン
contigファイル、abundanceファイルとk-merプロファイルファイルを指定する。
cd COCACOLA-python/
python cocacola.py \
--contig_file data/SpeciesMock/input/SpeciesMock_Contigs_cutup_10K_nodup_filter_1K.fasta \
--abundance_profiles data/SpeciesMock/input/cov_inputtableR.tsv \
--composition_profiles data/SpeciesMock/input/kmer_4.csv \
--output data/SpeciesMock/result.csv
-
--abundance_profiles ABUNDANCE_PROFILES: The abundance profiles, containing a table where each row correspond to a contig, and each column correspond to a sample. All values are separated with tabs.
-
--composition_profiles The composition profiles, containing a table where each row correspond to a contig, and each column correspond to the kmer composition of particular kmer. All values are separated with comma.
引用
COCACOLA: binning metagenomic contigs using sequence COmposition, read CoverAge, CO-alignment and paired-end read LinkAge
Yang Young Lu Ting Chen Jed A. Fuhrman Fengzhu Sun
Bioinformatics, Volume 33, Issue 6, 15 March 2017, Pages 791–798