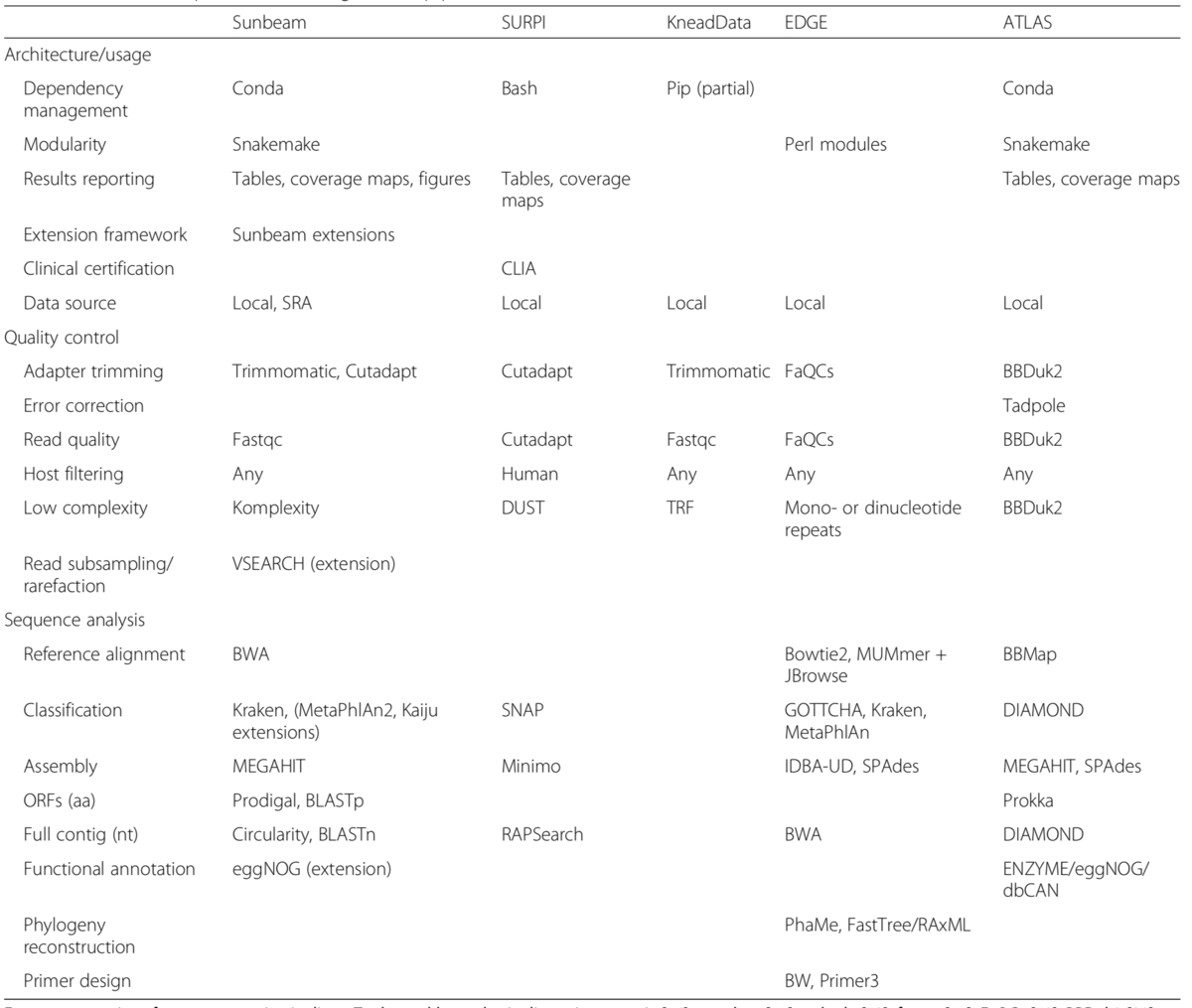
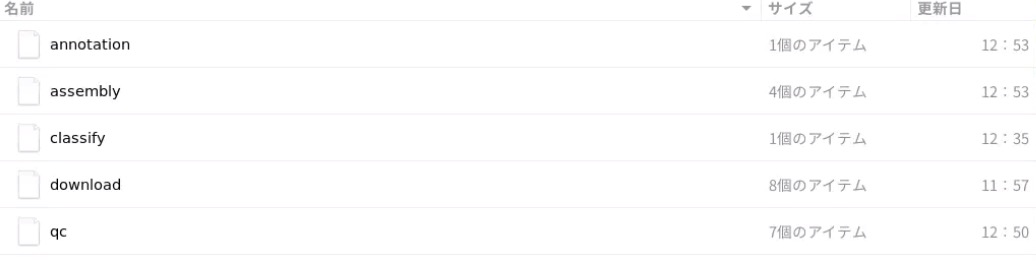
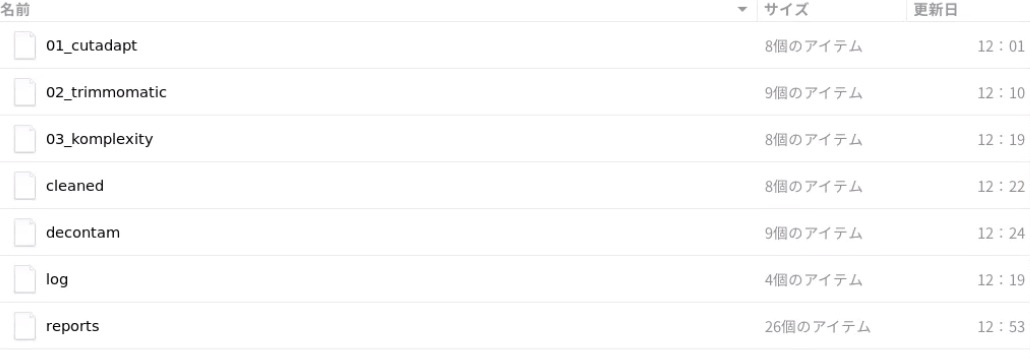
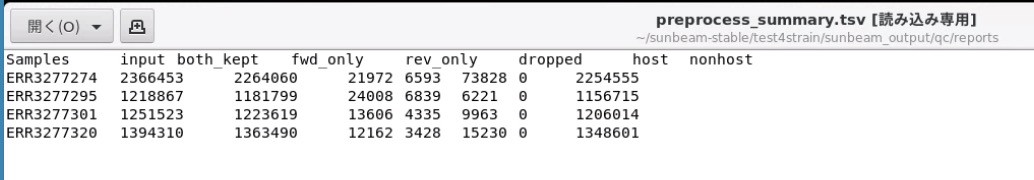
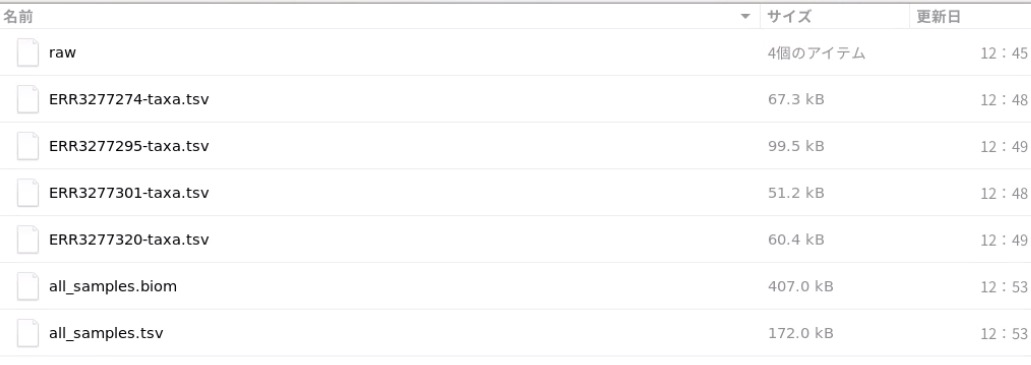
インストール
以前の記事を参照
> mmseqs
$ mmseqs
MMseqs2 (Many against Many sequence searching) is an open-source software suite for very fast,
parallelized protein sequence searches and clustering of huge protein sequence data sets.
Please cite: M. Steinegger and J. Soding. MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets. Nature Biotechnology, doi:10.1038/nbt.3988 (2017).
MMseqs2 Version: 905109c40ac720c407282d4d6517a164c3470fba
© Martin Steinegger (martin.steinegger@mpibpc.mpg.de)
Easy workflows (for non-experts)
easy-search Search with a query fasta against target fasta (or database) and return a BLAST-compatible result in a single step
easy-linsearch Linear time search with a query fasta against target fasta (or database) and return a BLAST-compatible result in a single step
easy-linclust Compute clustering of a fasta database in linear time. The workflow outputs the representative sequences, a cluster tsv and a fasta-like format containing all sequences.
easy-cluster Compute clustering of a fasta database. The workflow outputs the representative sequences, a cluster tsv and a fasta-like format containing all sequences.
easy-taxonomy Compute taxonomy and lowest common ancestor for each sequence. The workflow outputs a taxonomic classification for sequences and a hierarchical summery report.
Main tools (for non-experts)
createdb Convert protein sequence set in a FASTA file to MMseqs sequence DB format
search Search with query sequence or profile DB (iteratively) through target sequence DB
linsearch Search with query sequence DB through target sequence DB
map Fast ungapped mapping of query sequences to target sequences.
cluster Compute clustering of a sequence DB (quadratic time)
linclust Cluster sequences of >30% sequence identity *in linear time*
createindex Precompute index table of sequence DB for faster searches
createlinindex Precompute index for linsearch
enrich Enrich a query set by searching iteratively through a profile sequence set.
rbh Find reciprocal best hits between query and target
clusterupdate Update clustering of old sequence DB to clustering of new sequence DB
Utility tools for format conversions
createtsv Create tab-separated flat file from prefilter DB, alignment DB, cluster DB, or taxa DB
convertalis Convert alignment DB to BLAST-tab format or specified custom-column output format
convertprofiledb Convert ffindex DB of HMM files to profile DB
convert2fasta Convert sequence DB to FASTA format
result2flat Create a FASTA-like flat file from prefilter DB, alignment DB, or cluster DB
createseqfiledb Create DB of unaligned FASTA files (1 per cluster) from sequence DB and cluster DB
Taxonomy tools
taxonomy Compute taxonomy and lowest common ancestor for each sequence.
createtaxdb Annotates a sequence database with NCBI taxonomy information
addtaxonomy Add taxonomy information to result database.
lca Compute the lowest common ancestor from a set of taxa.
taxonomyreport Create Kraken-style taxonomy report.
filtertaxdb Filter taxonomy database.
An extended list of all tools can be obtained by calling 'mmseqs -h'.
Bash completion for tools and parameters can be installed by adding "source MMSEQS_HOME/util/bash-completion.sh" to your "$HOME/.bash_profile".
Include the location of the MMseqs2 binary is in your "$PATH" environment variable.
kazu@kazu:~/taniguchi_datadir/metagenome/sample2/test$
kamisakumanoMBP:~ kazuma$
> mmseqs cluster
$ mmseqs cluster
Usage: mmseqs cluster <i:sequenceDB> <o:clusterDB> <tmpDir> [options]
Compute clustering of a sequence DB (quadratic time)
Options:
Prefilter:
--max-seqs INT Maximum result sequences per query allowed to pass the prefilter (this parameter affects sensitivity) [20]
Align:
-c FLOAT list matches above this fraction of aligned (covered) residues (see --cov-mode) [0.800]
--cov-mode INT 0: coverage of query and target, 1: coverage of target, 2: coverage of query 3: target seq. length needs be at least x% of query length, 4: query seq. length needs be at least x% of target length [0]
-a add backtrace string (convert to alignments with mmseqs convertalis utility)
-e FLOAT list matches below this E-value (range 0.0-inf) [0.001]
--min-seq-id FLOAT list matches above this sequence identity (for clustering) (range 0.0-1.0) [0.000]
--min-aln-len INT minimum alignment length (range 0-INT_MAX) [0]
--seq-id-mode INT 0: alignment length 1: shorter, 2: longer sequence [0]
--alt-ali INT Show up to this many alternative alignments [0]
--force-reuse reuse tmp file in tmp/latest folder ignoring parameters and git version change
Clust:
--cluster-mode INT 0: Setcover, 1: connected component, 2: Greedy clustering by sequence length 3: Greedy clustering by sequence length (low mem) [0]
--single-step-clustering 0 switches from cascaded to simple clustering workflow [1, set to 0 to disable]
Common:
--threads INT number of cores used for the computation (uses all cores by default) [56]
--compressed INT write results in compressed format [0]
-v INT verbosity level: 0=nothing, 1: +errors, 2: +warnings, 3: +info [3]
An extended list of options can be obtained by calling 'mmseqs cluster -h'.
- Steinegger M, Soding J: Clustering huge protein sequence sets in linear time. Nature Communications, doi:10.1038/s41467-018-04964-5 (2018)
- Hauser M, Steinegger M, Soding J: MMseqs software suite for fast and deep clustering and searching of large protein sequence sets. Bioinformatics, 32(9), 1323-1330 (2016).
- Steinegger M, Soding J: MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets. Nature Biotechnology, doi:10.1038/nbt.3988 (2017)
3 Database paths are required
> mmseqs createdb
$ mmseqs createdb
Usage: mmseqs createdb <i:fastaFile1[.gz]> ... <i:fastaFileN[.gz]> <o:sequenceDB> [options]
Convert protein sequence set in a FASTA file to MMseqs sequence DB format
Options:
Misc:
--dont-split-seq-by-len 0 Dont split sequences by --max-seq-len [1, set to 0 to disable]
--dbtype INT Database type 0: auto, 1: amino acid 2: nucleotides [0]
--dont-shuffle 0 Do not shuffle input database [1, set to 0 to disable]
--id-offset INT numeric ids in index file are offset by this value [0]
Common:
--compressed INT write results in compressed format [0]
-v INT verbosity level: 0=nothing, 1: +errors, 2: +warnings, 3: +info [3]
An extended list of options can be obtained by calling 'mmseqs createdb -h'.
- Steinegger M, Soding J: MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets. Nature Biotechnology, doi:10.1038/nbt.3988 (2017)
2 Database paths are required
> mmseqs createtsv
$ mmseqs createtsv
Usage: mmseqs createtsv <i:queryDB> [<i:targetDB>] <i:resultDB> <o:tsvFile> [options]
Create tab-separated flat file from prefilter DB, alignment DB, cluster DB, or taxa DB
Options:
Misc:
--first-seq-as-repr Use the first sequence of the clustering result as representative sequence
--target-column INT Select a target column (default 1), 0 if no target id exists. [1]
--full-header Replace DB ID by its corresponding Full Header
--idx-seq-src INT 0: auto, 1: split/translated sequences, 2: input sequences [0]
--db-output Output a result db instead of a text file
Common:
--threads INT number of cores used for the computation (uses all cores by default) [56]
--compressed INT write results in compressed format [0]
-v INT verbosity level: 0=nothing, 1: +errors, 2: +warnings, 3: +info [3]
An extended list of options can be obtained by calling 'mmseqs createtsv -h'.
- Steinegger M, Soding J: MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets. Nature Biotechnology, doi:10.1038/nbt.3988 (2017)
> mmseqs result2flat -h
$ mmseqs result2flat -h
Usage: mmseqs result2flat <i:queryDB> <i:targetDB> <i:resultDB> <o:fastaDB> [options]
Create a FASTA-like flat file from prefilter DB, alignment DB, or cluster DB
By Martin Steinegger <martin.steinegger@mpibpc.mpg.de>
Options:
Misc:
--use-fasta-header use the id parsed from the fasta header as the index key instead of using incrementing numeric identifiers
Common:
-v INT verbosity level: 0=nothing, 1: +errors, 2: +warnings, 3: +info [3]
> mmseqs result2repseq
$ mmseqs result2repseq
Usage: mmseqs result2repseq <i:sequenceDB> <i:resultDB> <o:sequenceDb> [options]
Get representative sequences for a result database
Options:
Common:
--threads INT number of cores used for the computation (uses all cores by default) [56]
--compressed INT write results in compressed format [0]
-v INT verbosity level: 0=nothing, 1: +errors, 2: +warnings, 3: +info [3]
An extended list of options can be obtained by calling 'mmseqs result2repseq -h'.
- Steinegger M, Soding J: MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets. Nature Biotechnology, doi:10.1038/nbt.3988 (2017)
実行方法
mmseq2 cluster
1、まずproteome.fastaをデータベースに変換する。
mmseqs createdb proteome.fasta DB
2、 クラスタリング実行。作業ディレクトリが必要。ここでは/tmpとした。巨大なproteomeファイルの場合はかなりのディスクスペースを必要とするので、作業ディレクトリの空き容量に注意する。
mmseqs cluster DB DB_clu tmp
- --min-seq-id <FLOAT> list matches above this sequence identity (for clustering) (range 0.0-1.0) [0.000]
- --cov-mode <INT> 0: coverage of query and target, 1: coverage of target, 2: coverage of query 3: target seq. length needs be at least x% of query length, 4: query seq. length needs be at least x% of target length [0]
クラスタリング感度は"--min-seq-id"、"-c"、”--cov-mode”などを立てることで手動設定できる。
3、クラスタリング結果をTSV出力する。
mmseqs createtsv DB DB DB_clu clustered.tsv
> head clustered.tsv
$ head clustered.tsv
k161_4496204_2_367_+ k161_4496204_2_367_+
k161_2597819_391_528_- k161_2597819_391_528_-
k161_2397988_694_1326_- k161_2397988_694_1326_-
k161_2397988_694_1326_- k161_732702_1_726_+
k161_2397988_694_1326_- k161_4554834_402_1124_-
k161_1498756_826_1108_- k161_1498756_826_1108_-
k161_3197337_1_597_+ k161_3197337_1_597_+
k161_4696064_350_931_+ k161_4696064_350_931_+
k161_4696064_350_931_+ k161_4519536_22584_23195_-
k161_4696064_350_931_+ k161_3060204_1_699_+
4、クラスタリング後のfastaを出力する。
mmseqs result2flat DB DB DB_clu_rep DB_clu_rep.fasta --use-fasta-header
メタゲノムのアセンブリ配列をFragGeneScaにかけてprootein配列にしたもの(output.faa)を入力として、クラスタリングを実行した。

ラン後のファイルはDB_clu_rep.fastaだが、わずかに配列数が減少している。
mmseq2 linclust
Linclustは線形時間でのクラスタリングである。 実行時間は早くなるが、クラスタリングよりも少しだけ感度が劣る。
1、データベース作成
mmseqs createdb proteome.fasta DB
2、 クラスタリング実行。
mmseqs linclust DB DB_clu /tmp
4、クラスタリング後のfastaを出力する。
mmseqs result2repseq DB DB_clu DB_clu_rep
mmseqs result2flat DB DB DB_clu_rep DB_clu_rep.fasta --use-fasta-header
引用
MMseqs2 enables sensitive protein sequence searching for the analysis of massive data sets
Steinegger M, Söding J
Nat Biotechnol. 2017 Nov;35(11):1026-1028
MMseqs software suite for fast and deep clustering and searching of large protein sequence sets.
Hauser M, Steinegger M, Söding J
Bioinformatics. 2016 May 1;32(9):1323-30.
Clustering huge protein sequence sets in linear time
Martin Steinegger & Johannes Söding
Nat Commun. 2018; 9: 2542.
参考