基本的な流れは以下のリンクが参考になる。
github.com
今回はSRAに登録されているクラミドモナスのtime courseデータを使う。クラミドモナスはEnsemblにドラフトだがfastaもgtfも登録されているので非モデル生物より解析は楽になる。
クラミドモナスゲノムのダウンロードは
Chlamydomonas reinhardtii - Ensembl Genomes 35
のページの中ほどにある
Download DNA sequence (FASTA)
をクリックする。FTPで繋がり、任意のクロモソーム配列をダウンロードできる。
2、rRNAリードの除去
最初にrRNAにマッピングして、rRNAリードを除く。
rRNAのインデックス作成
bowtie2-build -f rRNA.fa rRNA
-f fastaファイルを指定
インデックス名をrRNA。この場合、rRNA~という6つのファイルが出力される。
rRNAへのマッピング
bowtie2 -x rRNA -1 R1.fastq -2 2.fastq -S rRNA_mapped.sam --un-conc rRNA_unmapped.fastq
-x 先ほどつけたインデックス名
-1 ペアードエンドデータのR1
-2 ペアードエンドデータのR2
-S マッピングされたデータ出力名(sam形式)
--un-conc マッピングされなかったfastqのペア。シングルなら--unを使う。上の場合はrRNA_unmapped 1.fastqとrRNA_unmapped 2.fastqが出力される。
ファイルサイズは2.03GBから2.02GBまでしか減らなかった。polyAから逆転写しているデータと思われる。ということで、このデータではrRNAの除去ステップは行わない。
3、クロモソームへのマッピング
faファイルとgtfファイルはEnsembl genomeの
http://plants.ensembl.org/info/website/ftp/index.html
のChlamydomonas reinhardtii からダウンロード。
インデックス作成
bowtie2-build -f Chlamydomonas_reinhardtii.v3.1.dna.toplevel.fa Chlamydomonas_reinhardtii.v3.1.dna.toplevel
ペアードエンドッピング
tophat -G Chlamydomonas_reinhardtii.v3.1.34.gtf -p 8 -o output --no-novel-juncs Chlamydomonas_reinhardtii.v3.1.dna.toplevel R1.fastq R2.fastq
-o 出力ディレクトリ名
-G gtfファイル。
-p スレッド数。多すぎるとエラーになるので8くらいに留める。
--no-novel-juncs アノテーションにない新規のジャンクションを破棄
最後にindex名、R1とR2のfastqを指定している。
4、FPKM算出
Cufflinksを使って計算する。
cufflinks -o output_FPKM -G genes.gtf -N --compatible-hits-norm mapping_0h/accepted_hits.bam
-G 既存のアイソフォームのみの計算の指示
N --compatible-hits-norm 既存のアイソフォームにマッピングされるものだけでFPKMを計算
-o FPKMの出力ディレクトリ名
最後にtophatで出力したbamファイルを指定する。複数結果がある時は順番に実行する。
出力されたらFPKMのカラムをcutで抜き出す
cut -f 10 mapping_0h/accepted_hits.bam > 0h_FPKM.txt
cut -f 10 mapping_2h/accepted_hits.bam > 2h_FPKM.txt
cut -f 10 mapping_0h/accepted_hits.bam > 4h_FPKM.txt
pasteコマンドでカラムを結合。
paste 0h_FPKM.txt 2h_FPKM.txt 4h_FPKM.txt > 0-4h_FPKM.txt
0-4h_FPKM.txtをエクセルかRに読み込んで経時変化をグラフ化する。
5、Cuffdiffによる発現量に差がある遺伝子の抽出
cuffdiff -o cuffdiff_out -p 20 Chlamydomonas_reinhardtii.v3.1.34.gtf -L 0h-1,0h-2,2h,4h,8h,12h,18h,24h,30h,45h,60h 0h-1_out/accepted_hits.bam 0h-2_out/accepted_hits.bam 2h_out/accepted_hits.bam 4h_out/accepted_hits.bam 8h_out/accepted_hits.bam 12h_out/accepted_hits.bam 18h_out/accepted_hits.bam 24h_out/accepted_hits.bam 30h_out/accepted_hits.bam 45h_out/accepted_hits.bam 60h_out/accepted_hits.bam
ファイルが多いとかなりの時間がかかる。
下のような大量のファイルが出力される。

おまけ、 Go termによる分類
今回使用したChlamydomonasのgtfファイルにはEntrezのgene_id情報がある。これがあればすぐにGo解析ができる。それはChlamydomonasの情報はEMBLの運営するEnsembl Genomesからダウンロードしており、すでにEnsembl のaccession IDが付いているからである。それに対して、De novoのトランスクリプトーム解析ならアノテーションがないため、アノテーション、ID変換等を駆使していかなかればGo termをつけてカテゴリ分類することはできない。
AgBase GORetrieverを貼ったプレーンテキストをアップロードして、Ensembl accession IDを選択。チェックは必要に応じて外すが、全選択してjobをなげても問題ない。しばらく待つとab-deliminatedファイルが
ダウンロードできるリンクが発生する。クリックしてダウンロードする。ただし、GOの分類はそもそもまだまだ主観性の強い分類であり、分類が間違っている可能性がある。どの階層を選ぶかによって結果も変わってくるため、確度の高い分析を行うのは厳しい。あくまで大雑把にどのような応答になっているのか掴むための手法と捉えた方がよさそうである。
7、統計解析
Rで行う。まずは準備。
CummuneRbandのインストール
参照したページ。こんなことができるようです。
CummeRbund - An R package for persistent storage, analysis, and visualization of RNA-Seq from cufflinks output
#ターミナルでcufflinks解析結果ディレクトリの上に移動して、(つまり上の解析時そのままの位置で) Rを起動(ターミナルでRと打ち、エンター)
R
library(cummeRbund) #ライブラリ読み込み
cuff <- readCufflinks("cuffdiff_out") #cufflinks解析結果のディレクトリ全体を読み込んでくれる
#これでCuffSet オブジェクトが生成された。
cuff
#正常に読み込まれていれば、cuffオブジェクトを分析しcuflinksの解析結果の概要が表示される。サンプルは0-60hのタイムコース実験の11サンプル。
CuffSet instance with:
11 samples
14487 genes
14553 isoforms 0
TSS 0
CDS 0
promoters 0
splicing 0
relCDS
上のgenesの分布
csBoxplot(genes(cuff))
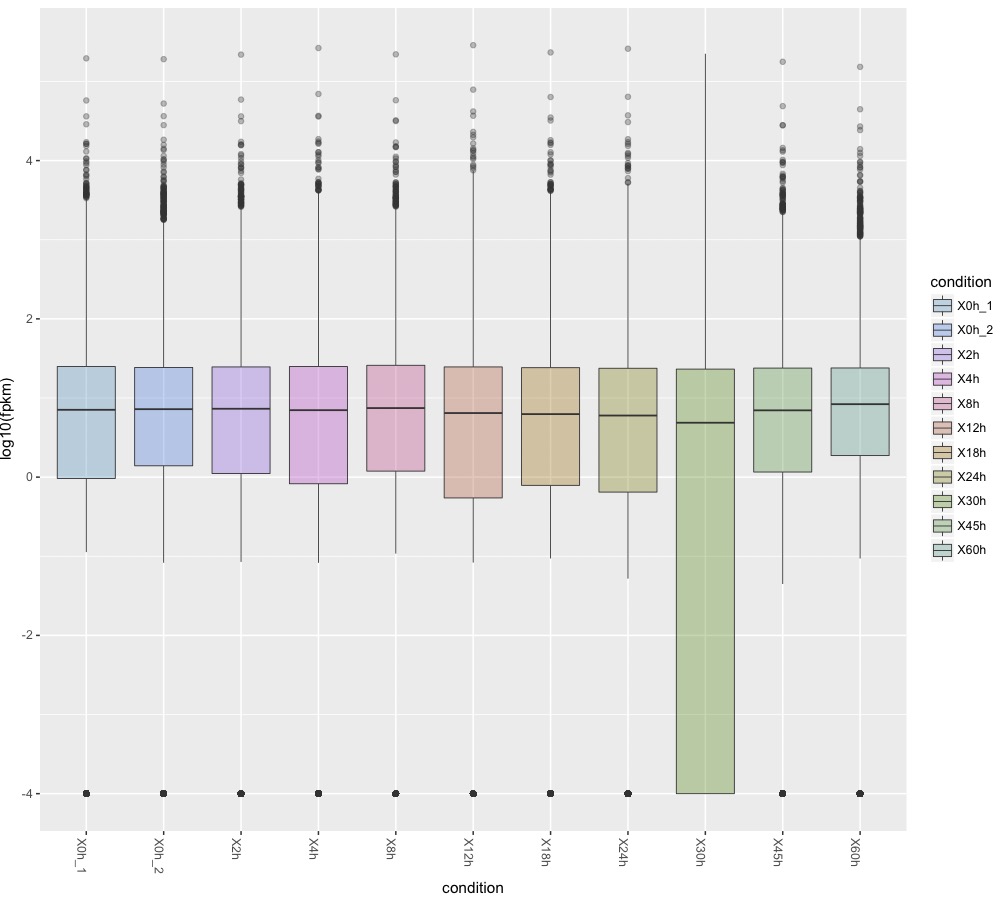
上の図作成は
box <- csBoxplot(genes(cuff))
print(box)
のようにやってもよい。この場合はまずcsBoxplot(genes(cuff))の結果をboxに代入し、それをpirntコマンドで画面に描画している。
#さらにpngで保存するなら
png("box.png")
plot(box)
dev.off()
これを同時にうつ。
分子生物学ではあまり使わないので、box plot(箱ひげ図)の見かたについて触れておく。(参考)
1、箱の中央付近のヨコ線 → データxの中央値。平均値ではない。また必ずしもboxの中央にあるわけでもない点に注意。
2、箱のヨコ線 → データxの第1四分位数(下側)と第3四分位数(上側)
分かりにくいので言い換える。箱の底辺の線の位置は、データの中央値と一番小さい値との間の中央値。つまり、中央値を最大値と考え、その最大値から下半分のデータについての中央値を求め、その位置を箱の低線の位置としている。この1/4番目の区切りの中央値を第1四分位数という。同様に箱の上辺の線の位置はデータの中央値と一番大きい値との間の中央値(=第3四分位数)。
3、箱の上下に向かって縦方向に伸びている線(ヒゲ) → データの最大値と最小値。
ここで言う最大値と最小値はデータの真の最大値と最小値ではなくて外れ値を除外したもの。ヒゲの下方向の長さは
箱の底辺 - (箱の高さ)x 1.5
で出たの範囲で最も小さな値となる。
上の式を難しく言い換えると
第1四分位数-1.5 x (第3四分位数-第1四分位数)
ヒゲの上方向の長さは
第3四分位数+1.5*(第3四分位数-第1四分位数)
となる。その範囲に入らなかった値が外れ値として黒いプロットで表現されている。
箱ひげ図は品質管理で用いられたりしている。RNA seqの品質を管理するものとして捉えればよい。箱の位置が被らないようなデータは有意な差がある可能性が高い。30hのデータを見ると、中央値は他のデータと大差ないが箱の底辺は遥か下まで伸びている。言い換えると第1四分位数が他のサンプルと大きく異なっている。
isoformのbox plotを書く。
csBoxplot(isoforms(cuff))
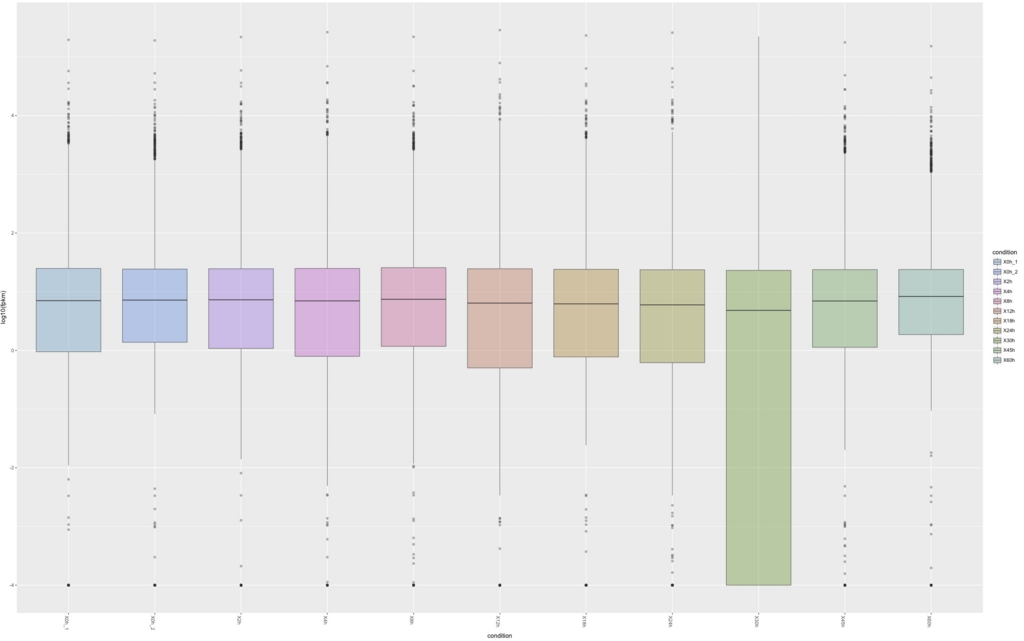
アイソフォームでも30hのデータはおかしい。これが生物的な応答なのかサンプルの品質なのかは後で追及する。
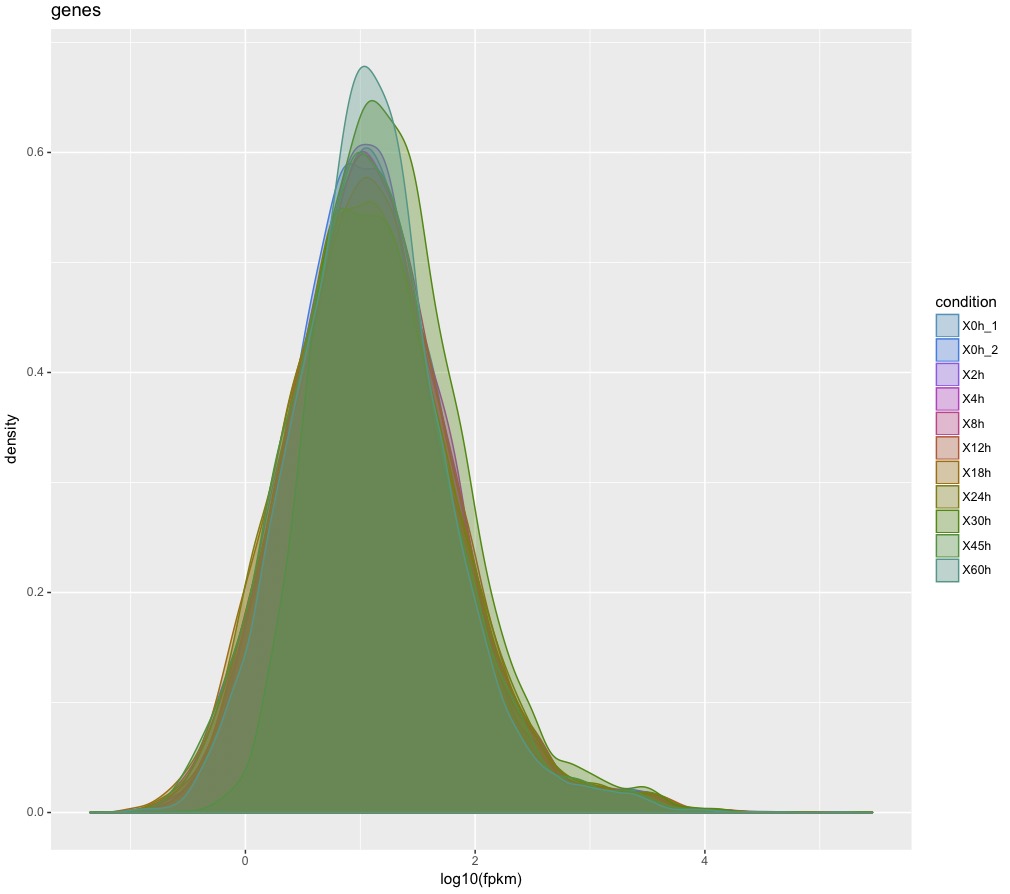
csDensity(genes(cuff)) #ヒストグラム
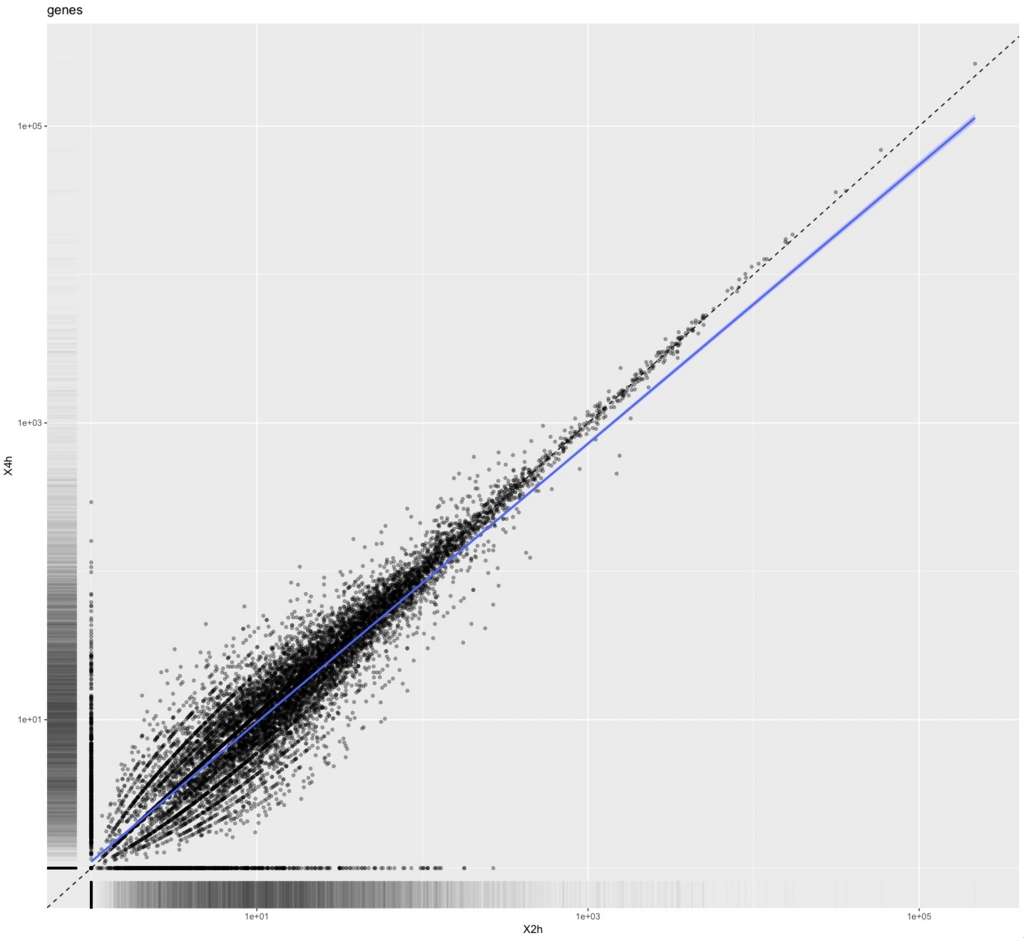
2サンプル間の散布図
(object)
を使う。
samples(cuff)
sample_index sample_name sample_name parameter value
1 1 X0h_1 <NA> <NA> <NA>
2 2 X0h_2 <NA> <NA> <NA>
3 3 X2h <NA> <NA> <NA>
4 4 X4h <NA> <NA> <NA>
5 5 X8h <NA> <NA> <NA>
6 6 X12h <NA> <NA> <NA>
7 7 X18h <NA> <NA> <NA>
8 8 X24h <NA> <NA> <NA>
9 9 X45h <NA> <NA> <NA>
10 10 X60h <NA> <NA> <NA>
なので、サンプルX2hとX4hを使って散布図を書くなら
csScatter(genes(cuff),"X2h","X4h",smooth=T)
csDendro(genes(cuff)) #樹形図
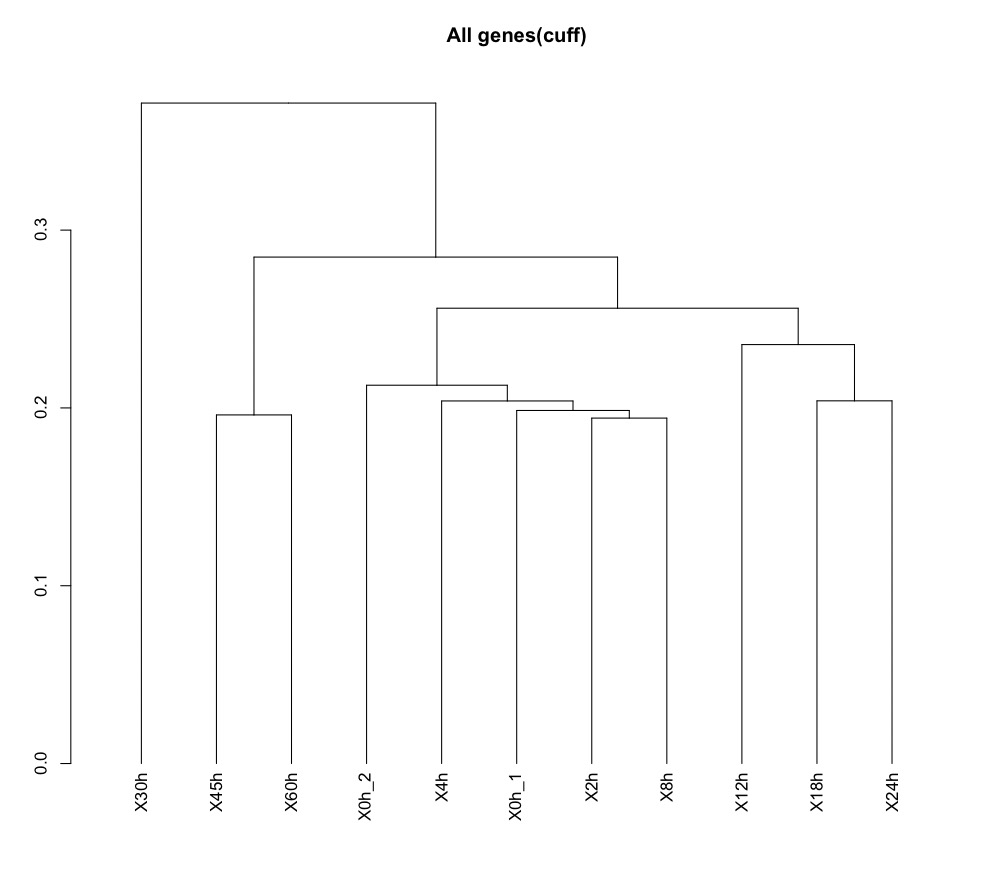
csScatterMatrix(genes(cuff))

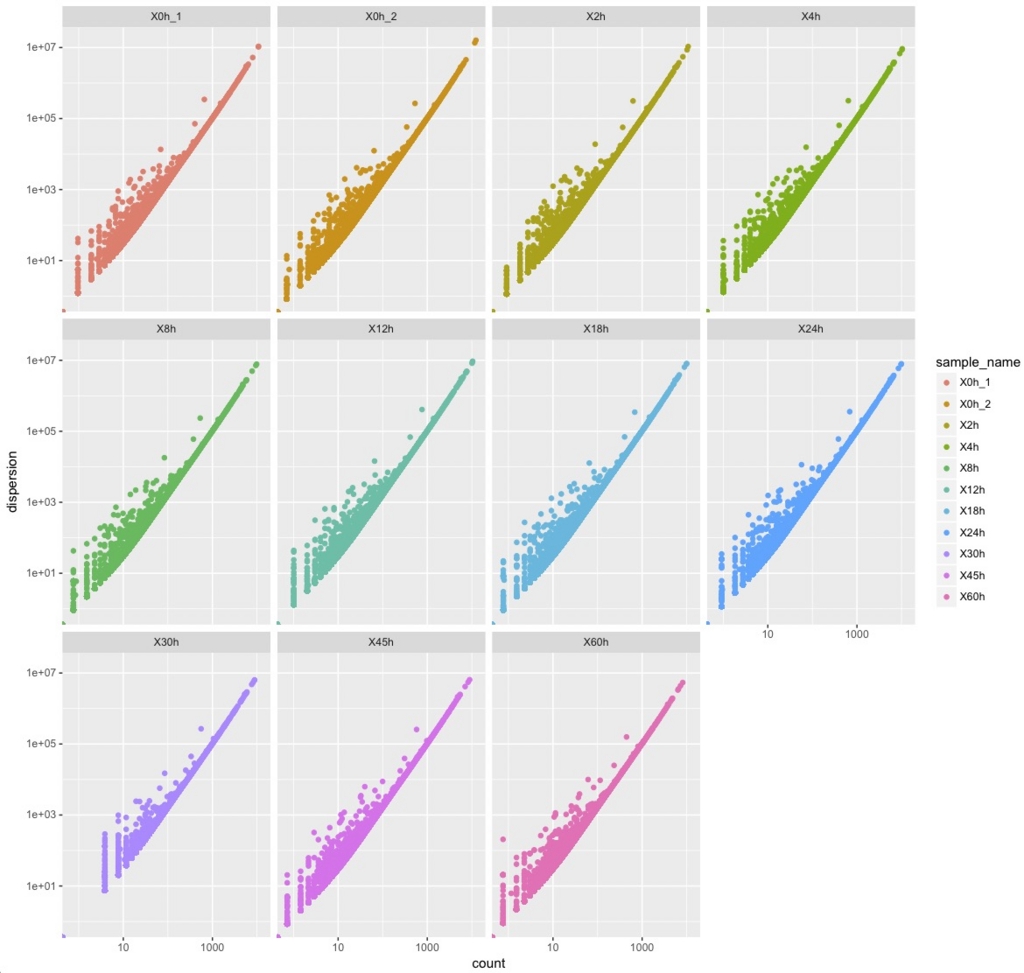
dispersionPlot(genes(cuff))
csVolcanoMatrix(genes(cuff))

一部分を拡大
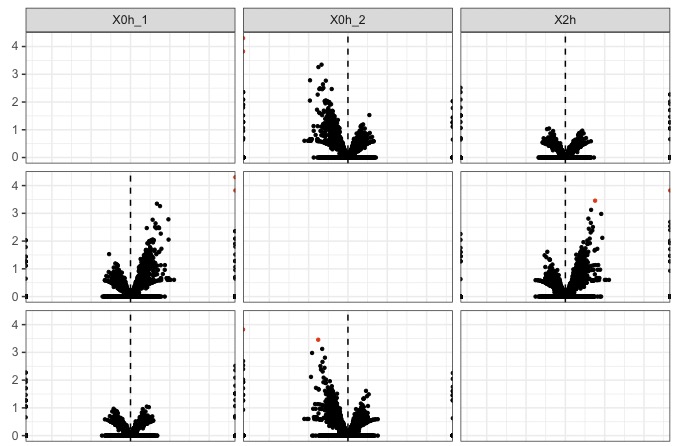
横軸はlog2(fold change)
縦軸はlog10(p value)
#genediffというオブジェクトに差のある遺伝子データ(genes)を読み込む。
genediff <- diffData(genes(cuff))
#最初の部分だけを表示
head(genediff,3)
gene_id sample_1 sample_2 status value_1 value_2 log2_fold_change test_stat p_value q_value significant
1 AAB93440 X0h_1 X0h_2 NOTEST 1.34796 4.71785 1.80735 0 1 1 1 no
2 AAB93441 X0h_1 X0h_2 NOTEST 0.00000 0.00000 0.00000 0 2 1 1 no
3 AAB93442 X0h_1 X0h_2 NOTEST 0.00000 0.00000 0.00000 0 3 1 1 no
ここでRの基本的なコマンドを学習しておく。
#一番前にgene_idがあることに注目して差のある遺伝子のIDのカラムだけ抽出、genediffdataIDに代入。
genediffdataID <- genediff[,1]
#中身をチェック
head(genediffdataID,10)
[1] "AAB93440" "AAB93441" "AAB93442"
[4] "AAB93443" "AAB93444" "AAB93445"
[7] "AAB93446" "AAB93447" "CHLREDRAFT_100005"
[10] "CHLREDRAFT_100008"
#headはカラムが少ないとこのように1つの行に3行分表示したりする。分かりにくい。
ファイルに保存してexcelで開いて見ると
write.table(genediffdataID, file="result.csv", sep=",")
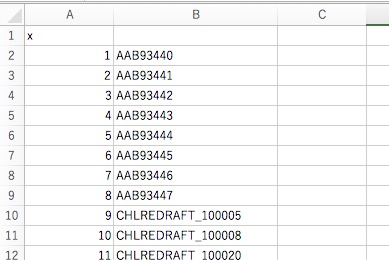
こんな感じ。
#データ解析には、縦のカラムではだめで行ベクトルに縦横変換する必要がある。縦横変換は
genediffdataID2 <- t(genediffdataID)
#ファイルを保存して
write.table(genediffdataID2, file="result.csv", sep=",")
#excelで開くと縦横変換されている。

#ちなみにこれをターミナルからカッコよく開くなら
open result.csv #csvのデフォルトアプリで開く
#uesakaGenesオブジェクトにcuffオブジェクトからこのgenediffdataID2を持つ遺伝子の情報だけ代入
uesakaGenes <-getGenes(cuff, genediffdataID2)
#uesakaGenesをもとにヒートマップを書かせる
csHeatmap(uesakaGenes)
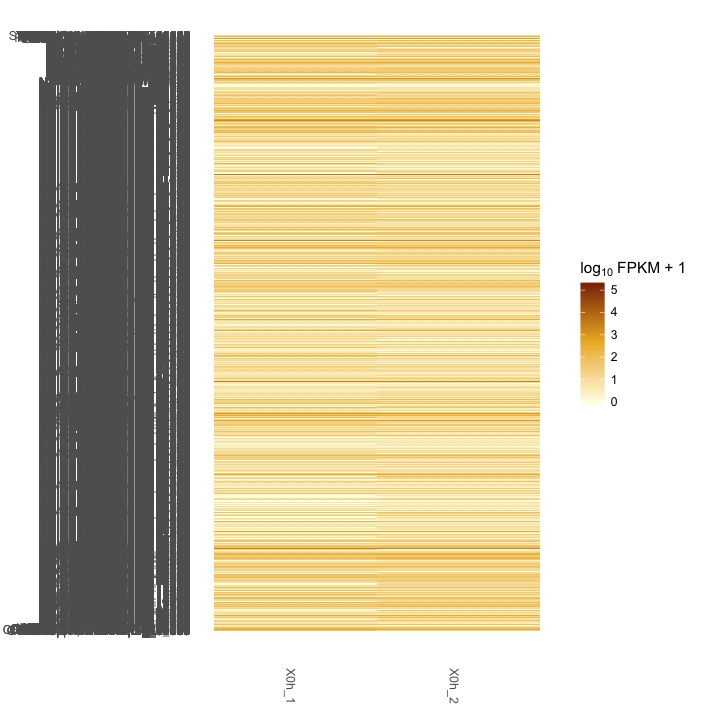
#数が多すぎてよくわからない。発現量が多い遺伝子だけに絞り込む。
#そのため差のある遺伝子のIDのカラムだけ抽出のところからやり直す。
genediff <- diffData(genes(cuff))
#統計的に優位なものだけ取ってくる。(significant列がyes)
genediff2 <- genediff[(genediff$significant == 'yes'),]
genediff2
#うてば何行残っているかわかる。2527行だけ残っていた。
#ファイルに保存してexcelで開くと
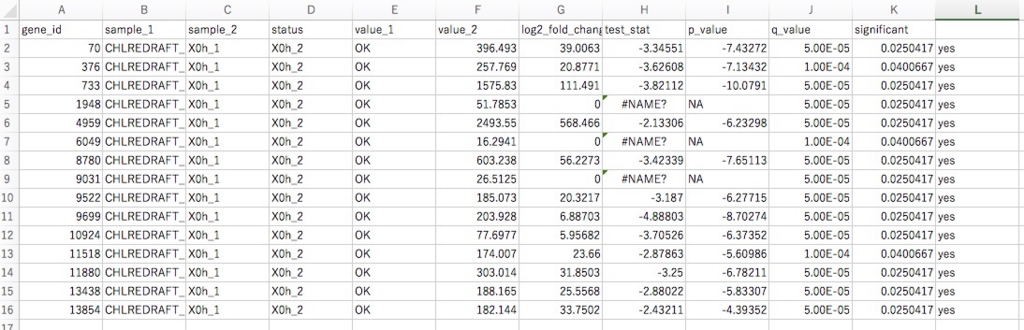
#右端の列がyesになっていることが確認できる。
あとは同じような流れ。
genediffdataID2 <- genediff2[,1]
genediffdataID2 <- t(genediffdataID2)
uesakaGenes2 <-getGenes(cuff, genediffdataID2)
#Rでは k-means によるクラスタリングを行う関数が標準で用意されている。
#はじめてk-means法を聞く人が概要を掴むならiOSアプリの
の中のk-meansの説明で素晴らしく分かりやすく説明されている。
k.means <-csCluster(uesakaGenes2, k=4)
k.means.plot <- csClusterPlot(k.means)
k.means.plot
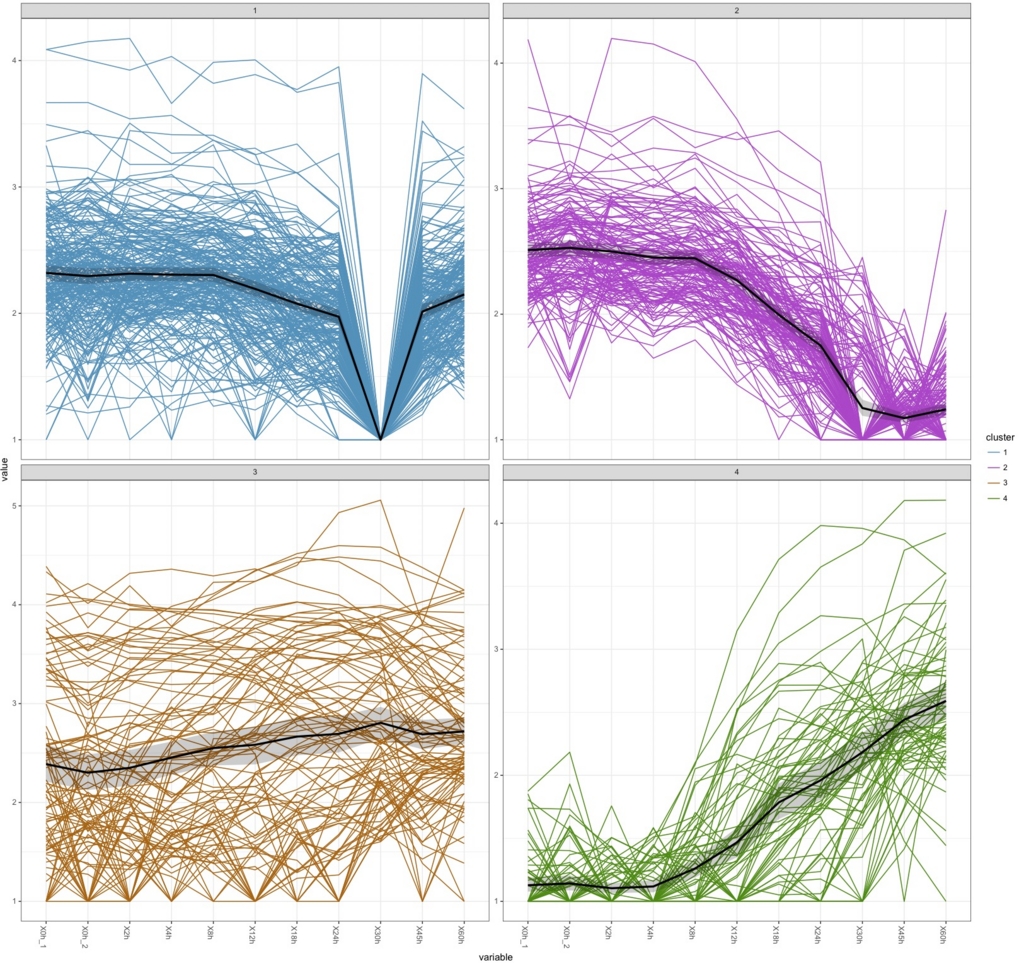
#k=6にすると
k.means <-csCluster(uesakaGenes2, k=6)
k.means.plot <- csClusterPlot(k.means)
k.means.plot
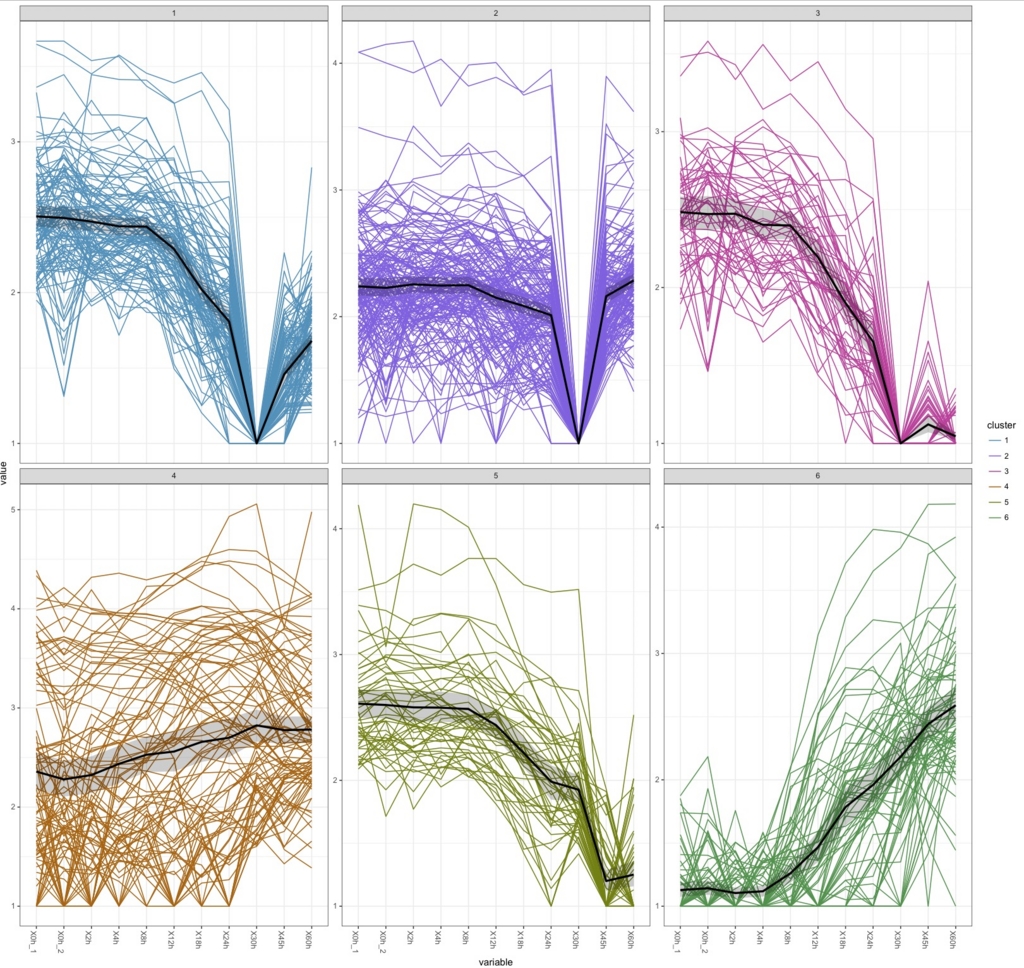
#k=8にすると
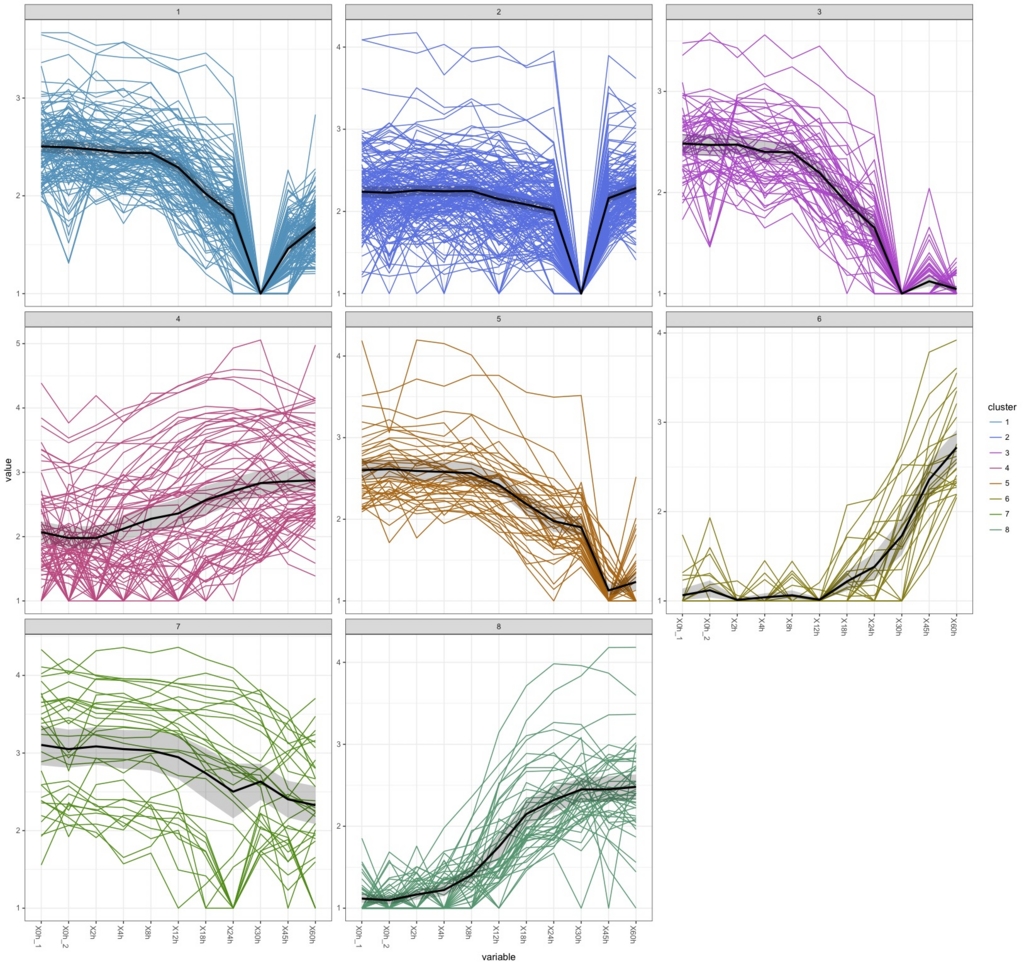
k.means <-csCluster(uesakaGenes2, k=8)
k.means.plot <- csClusterPlot(k.means)
k.means.plot
#k=9にすると
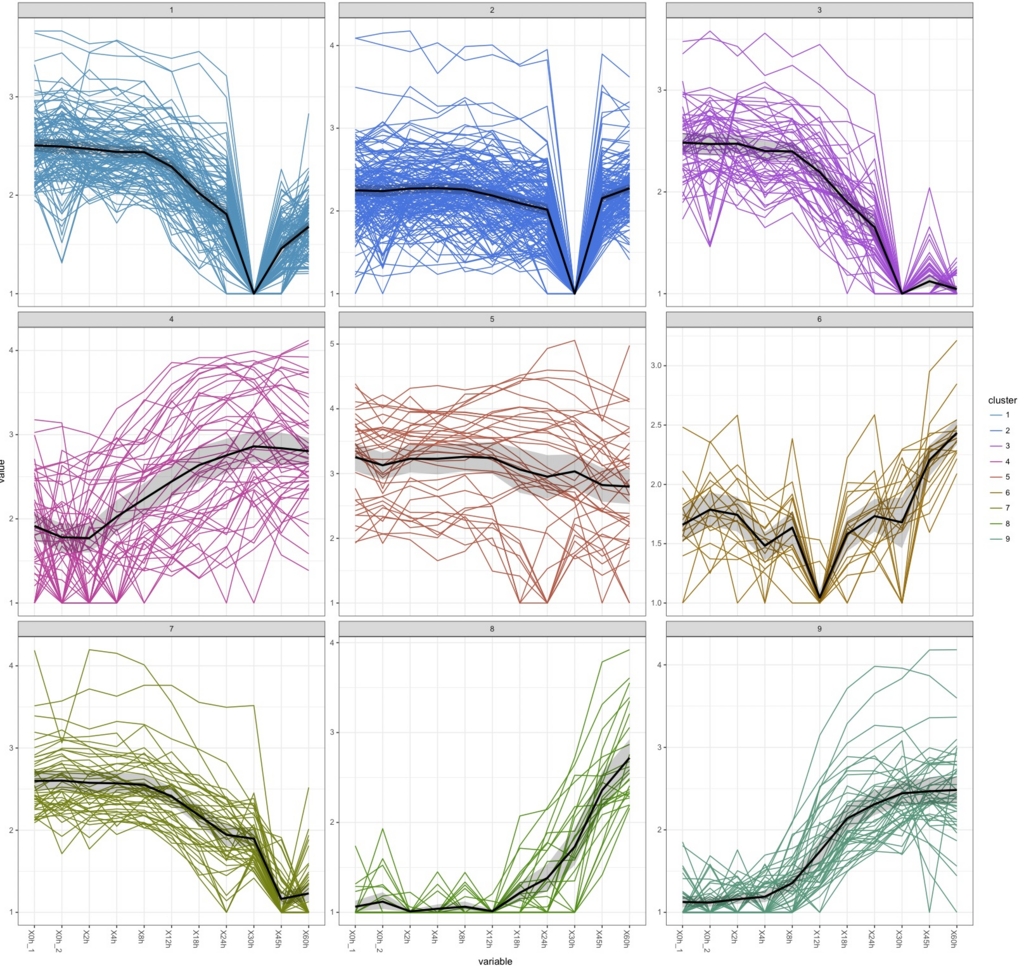
k.means <-csCluster(uesakaGenes2, k=9)
k.means.plot <- csClusterPlot(k.means)
k.means.plot
#k=12にすると
k.means <-csCluster(uesakaGenes2, k=12)
k.means.plot <- csClusterPlot(k.means)
k.means.plot
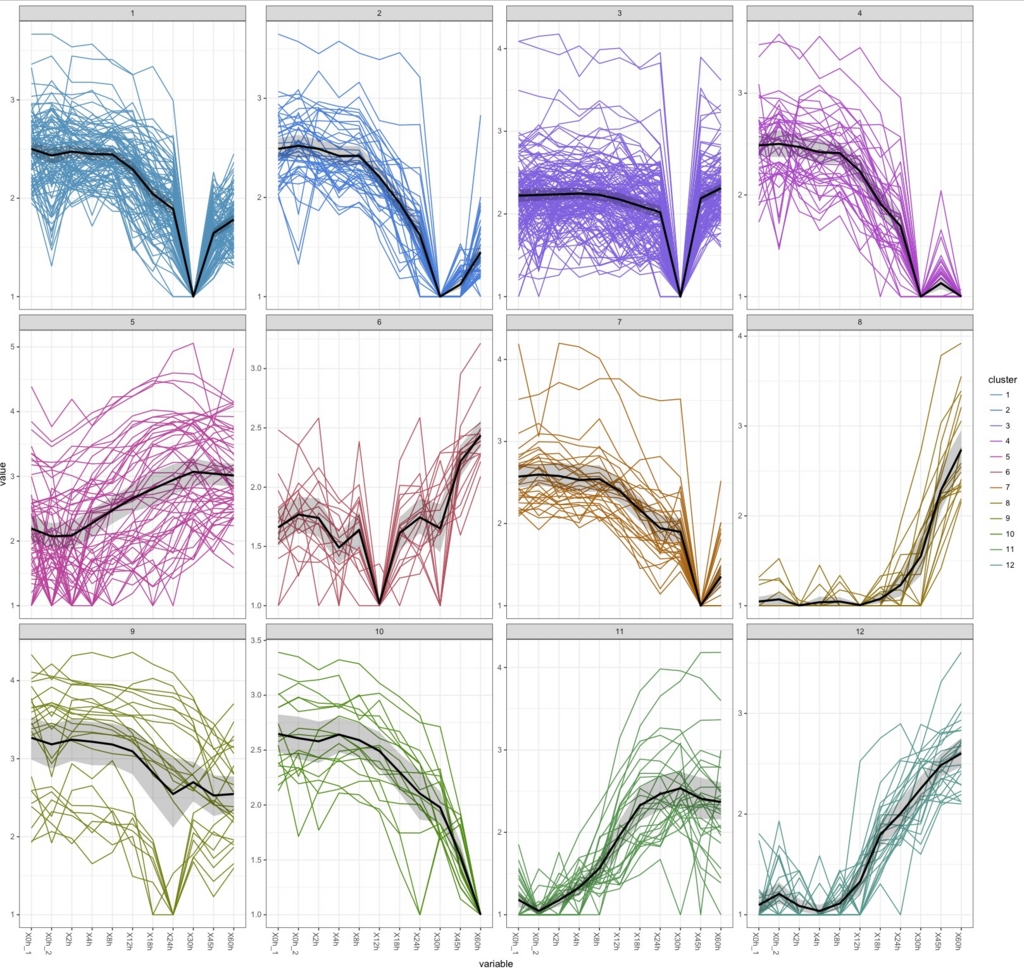
これはさすがにやりすぎ。直感的には9が過不足なく綺麗に分けられている気がするがどうだろう。
30hで沈むデータがかなりあるのがわかる。box plotで30hだけおかしかったのはこれを反映しているのだろう。ぶっちゃけて言うと、おそらく30hのデータを除かないと正しい解析はできないだろう。
次回に続く。




 disk9が該当するディスク。
disk9が該当するディスク。













































