2020 11/6 追記
2023/01/010. 01/11 インストール手順修正
Long terminal repeat retrotransposons (LTR-RT)は植物ゲノムに多く存在する。LTR-RTの同定は、高品質な遺伝子アノテーションを実現するために重要である。しかし、これらのプログラムは特異性が低く、偽発見率が高いという問題があった。ここでは、LTR-RTを同定し、ゲノム配列から高品質のLTRライブラリを生成するマルチスレッド対応のPerlプログラムであるLTR_retrieverについて報告する。LTR_retrieverは、イネ(Oryza sativa)において、感度(91%)、特異度(97%)、精度(96%)、精度(90%)の高いレベルを達成し、大幅な改善を示した。LTR_retrieverは、ロングシークエンシングリードにも対応している。シロイヌナズナ(Arabidopsis thaliana)の4.5×ゲノムカバレッジに相当する40kの自己補正PacBioリードで、構築されたLTRライブラリは優れた感度と特異性を示した。LTR_retrieverは、5'-TG...CA-3'末端を持つcanonical LTR-RTに加えて、これまでゲノムワイドな研究ではほとんど無視されてきたnoncanonical LTR-RT(non-TGCA)も同定した。そして植物ゲノム50個の中の42個から7種類のnoncanonical LTRを同定した。noncanonical LTRの大部分は Copia elementsであり、そのLTRは他の Copia elementsに比べて4倍も短く、これは標的特異性の結果であると考えられる。驚くべきことに、非TGCA Copia elementsは、多くの場合、遺伝子領域に位置し、近くまたは遺伝子内に優先的に挿入し、遺伝子の進化に影響を与え、突然変異誘発ツールとしての可能性を示している。
LTR_retrieverは、LTRharvest、LTR_FINDER、MGEScan 3.0.0、LTR_STRUC、LtrDetectorの出力からLTRレトロトランスポゾン(LTR-RT)を正確に同定し、ゲノムアノテーション用の非冗長LTR-RTライブラリを生成するためのコマンドラインプログラム(Perl)である。
インストール
ubuntu18.04でテストした。
Github
#以下3つはltr_retrieverで使用するファイルを作成するために必要
#genometools
sudo apt-get install -y genometools
mamba install -c bioconda ltr_finder -y
git clone https://github.com/oushujun/LTR_FINDER_parallel.git
#最後に本体
#bioconda (link)
mamba install -c bioconda ltr_retriever -y
> LTR_retriever -h
$ LTR_retriever -h
##########################
### LTR_retriever v2.9.0 ###
##########################
A program for accurate identification of LTR-RTs from outputs of LTRharvest and
LTR_FINDER, generates non-redundant LTR-RT library for genome annotations.
Shujun Ou (shujun.ou.1@gmail.com) 03/26/2019
Usage: LTR_retriever -genome genomefile -inharvest LTRharvest_input [options]
【Input Options】
-genome [File] Specify the genome sequence file (FASTA)
-inharvest [File] LTR-RT candidates from LTRharvest
-infinder [File] LTR-RT candidates from LTR_FINDER
-inmgescan [File] LTR-RT candidates from MGEScan_LTR
-nonTGCA [File] Non-canonical LTR-RT candidates from LTRharvest
【Output options】
-verbose/-v Retain intermediate outputs (developer mode)
-noanno Disable whole genome LTR-RT annotation (no GFF3 output)
【Filter options】
-misschar [CHR] Specify the ambiguous character (default N)
-Nscreen Disable filtering ambiguous sequence in candidates
-missmax [INT] Maximum number of ambiguous bp allowed in a candidate (default 10)
-missrate [0-1] Maximum percentage of ambiguous bp allowed in a candidate (default 0.8)
-minlen [INT] Minimum bp of the LTR region (default 100)
-max_ratio [FLOAT] Maximum length ratio of internal region/LTR region (default 50)
-minscore [INT] Minimum alignment length (INT/2) to identify tandem repeats (default 1000)
-flankmiss [1-60] Maximum ambiguous length (bp) allowed in 60bp-flanking sequences (default 25)
-flanksim [0-100] Minimum percentage of identity for flanking sequence alignment (default 60)
-flankaln [0-1] Maximum alignment portion allowed for 60bp-flanking sequences (default 0.6)
-motif STRING Specify non-canonical motifs to search for
(default -motif [TCCA TGCT TACA TACT TGGA TATA TGTA TGCA])
-notrunc Discard truncated LTR-RTs and nested LTR-RTs (will dampen sensitivity)
-procovTE [0-1] Maximum portion of allowed for cumulated DNA TE database and LINE database
lignments (default 0.7)
-procovPL [0-1] Maximum portion allowed for cumulated plant protein database alignments (default 0.7)
-prolensig [INT] Minimum alignment length (aa) for LINE/DNA transposase/plant protein alignment (default 30)
【Library options】
-blastclust STRING Trigger to use blastclust and customize parameters
(default -blastclust [-L .9 -b T -S 80])
-cdhit STRING Trigger to use cd-hit-est (default) and customize parameters
(default -cdhit [-c 0.8 -G 0.8 -s 0.9 -aL 0.9 -aS 0.9 -M 0])
-linelib [FASTA] Provide LINE transposase database for LINE TE exclusion
(default /database/Tpases020812LINE)
-dnalib [FASTA] Provide DNA TE transposase database for DNA TE exclusion
(default /database/Tpases020812DNA)
-plantprolib [FASTA] Provide plant protein database for coding sequence exclusion
(default /database/alluniRefprexp082813)
-TEhmm [Pfam] Provide Pfam database for TE identification
(default /database/TEfam.hmm)
【Dependencies】
-repeatmasker [path] Path to the RepeatMasker program. (default: find from ENV)
-blastplus [path] Path to the BLAST+ program. (default: find from ENV)
-blast [path] Path to the BLAST program. Required if -blastclust is used. (default: find from ENV)
-cdhit_path [path] Path to the CD-HIT program. Required if -cdhit is used. (default: find from ENV)
-hmmer [path] Path to the HMMER program. (default: find from ENV)
-trf_path [path] Path to the trf program. (default: find from ENV)
【Miscellaneous】
-u [FLOAT] Neutral mutation rate (per bp per ya) (default 1.3e-8 (from rice))
-step [STRING] Restart the program from a particular step. Existing outputs will be overwritten. Options:
Init (default, from the beginning);
Major (Tandem repeat cleanup finished, structrual analyses next)
Trunc (Structural analyses finished, truncated LTR recycle next)
Promask (Truncated LTR recycle finished, protein contamination cleanup next)
Library (Protein contamination cleanup finished, initial library construction next)
Next (Initial library construction finished, non-TGCA analyses next)
-threads [INT] Number of threads (≤ total available threads, default 4)
-help/-h Display this help information
###### For Questions and Issues Please See: https://github.com/oushujun/LTR_retriever ######
> perl ../LTR_FINDER_parallel/LTR_FINDER_parallel
$ perl ../LTR_FINDER_parallel/LTR_FINDER_parallel
Using this LTR_FINDER: /home/kazu/miniconda3/envs/ltr_retriever/bin/
At least 1 parameter mandatory:
1) Input fasta file: -seq
~ ~ ~ Run LTR_FINDER in parallel ~ ~ ~
Author: Shujun Ou (shujun.ou.1@gmail.com)
Date: 09/19/2018
Update: 01/28/2020
Version: v1.1
Usage: perl LTR_FINDER_parallel -seq [file] -size [int] -threads [int]
Options: -seq [file] Specify the sequence file.
-size [int] Specify the size you want to split the genome sequence.
Please make it large enough to avoid spliting too many LTR elements. Default 5000000 (bp)
-time [int] Specify the maximum time to run a subregion (a thread).
This helps to skip simple repeat regions that take a substantial of time to run. Default: 1500 (seconds).
Suggestion: 300 for -size 1000000. Increase -time when -size increased.
-try1 [0|1] If a region requires more time than the specified -time (timeout), decide:
0, discard the entire region.
1, further split to 50 Kb regions to salvage LTR candidates (default);
-harvest_out Output LTRharvest format if specified. Default: output LTR_FINDER table format.
-next Only summarize the results for previous jobs without rerunning LTR_FINDER (for -v).
-verbose|-v Retain LTR_FINDER outputs for each sequence piece.
-finder [file] The path to the program LTR_FINDER (default v1.0.7, included in this package).
-threads|-t [int] Indicate how many CPU/threads you want to run LTR_FINDER.
-check_dependencies Check if dependencies are fullfiled and quit
-help|-h Display this help information.
実行方法
1、create a suffix array index
#最初に必要なインデックス等を作成
gt suffixerator -db genome.fa -indexname genome_index -tis -suf -lcp -des -ssp -sds -dna
#ゲノム配列中のLTRレトロトランスポゾンをde novoで予測(manual)
gt ltrharvest -index genome_index -minlenltr 100 -maxlenltr 7000 -mintsd 4 -maxtsd 6 -motif TGCA -motifmis 1 -similar 85 -vic 10 -seed 20 -seqids yes > genome.fa.harvest.scn
#ゲノム配列中の完全長LTRレトロトランスポンソンを見つけるLTR_FINDER(開発終了)のparallel版をラン
perl LTR_FINDER_parallel/LTR_FINDER_parallel -seq genome.fa -threads 10 -harvest_out -size 1000000 -time 300
#2つのscnを結合
cat genome.fa.harvest.scn genome.fa.finder.combine.scn > genome.fa.rawLTR.scn
LTR_FINDER_parallelのランにより、genome.fa.rawLTR.scnが出力される。
2、genome.fa.rawLTR.scnとゲノムを指定する。
LTR_retriever -genome genome.fa -inharvest genome.fa.rawLTR.scn -threads 20
#時間のかかるアノテーションは行わないなら-noannoをつける
LTR_retriever -genome genome.fa -inharvest genome.fa.rawLTR.scn -threads 20 -noanno
- -noanno Disable whole genome LTR-RT annotation (no GFF3 output)
テストラン
Chlamydomonas_reinhardtiiのv5,5アセンブリを使ってテストする。パラメータはオーサーらの例に従う。ゲノムアセンブリのfastaのヘッダー名やファイル名は、特殊文字や空白が残らないようにあらかじめ置き換えている。
gt suffixerator -db C.reinhardtii.fa -indexname genome_index -tis -suf -lcp -des -ssp -sds -dna
gt ltrharvest -index genome_index -minlenltr 100 -maxlenltr 7000 -mintsd 4 -maxtsd 6 -motif TGCA -motifmis 1 -similar 85 -vic 10 -seed 20 -seqids yes > C.reinhardtii.harvest.scn
perl ../LTR_FINDER_parallel/LTR_FINDER_parallel -seq C.reinhardtii.fa -threads 20 -harvest_out -size 1000000 -time 300
cat C.reinhardtii.harvest.scn C.reinhardtii.fa.finder.combine.scn > genome.fa.rawLTR.scn
LTR_retriever -genome C.reinhardtii.fa -inharvest genome.fa.rawLTR.scn -threads 20
出力

LTR_retriever の出力には、以下のものが含まれる。
- 座標および構造情報を持つインタクトな LTR-RT
- サマリーテーブル (.pass.list)
- GFF3 形式の出力 (.pass.list.gff3)
- LTR-RTライブラリ
- すべての冗長でない LTR-RT (.LTRlib.fa)
- すべての非TGCA LTR-RT (.nmtf.LTRlib.fa)
- 冗長性のあるすべての LTR-RT (.LTRlib.redundant.fa)
- 非冗長ライブラリによる全ゲノムLTR-RTアノテーション
- GFF フォーマット出力 (.out.gff)
- LTR ファミリー要約 (.out.fam.size.list)
- LTR スーパーファミリー要約 (.out.superfam.size.list)
- 各染色体におけるLTRの分布 (.out.LTR.distribution.txt)
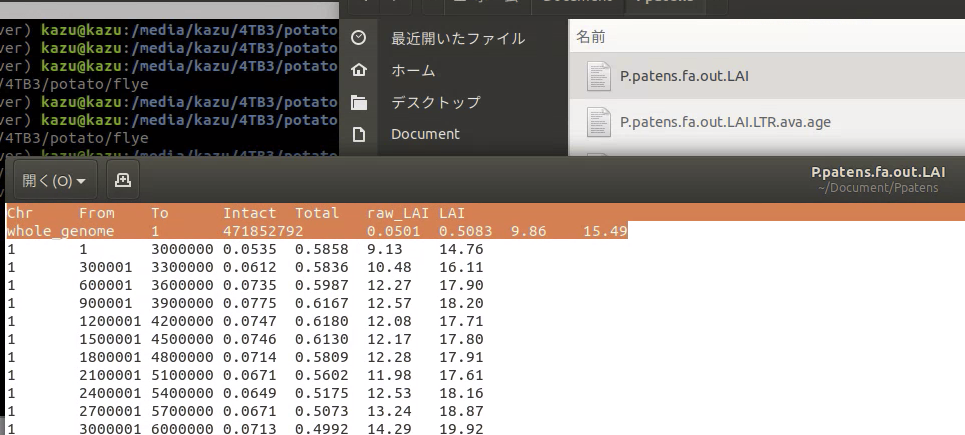
- LTR アセンブリインデックス (.out.LAI)
引用
LTR_retriever: A Highly Accurate and Sensitive Program for Identification of Long Terminal Repeat Retrotransposons
Shujun Ou, Ning Jiang
Plant Physiol. 2018 Feb;176(2):1410-1422
関連
genometoolsのgtサブコマンドのヘルプ(link)
> gt suffixerator -help
$ gt suffixerator -help
Usage: gt suffixerator [option ...] (-db file [...] | -ii index)
Compute enhanced suffix array.
-ssp output sequence separator positions to file
default: yes
-des output sequence descriptions to file
default: yes
-sds output sequence description separator positions to file
default: yes
-md5 output MD5 sums to file
default: yes
-clipdesc clip descriptions after first whitespace
default: no
-sat specify kind of sequence representation
by one of the keywords direct, bytecompress, eqlen, bit, uchar,
ushort, uint32
default: undefined
-dna input is DNA sequence
default: no
-protein input is protein sequence
default: no
-indexname specify name for index to be generated
default: undefined
-db specify database files
-smap specify file containing a symbol mapping
default: undefined
-lossless allow lossless original sequence retrieval
default: no
-mirrored virtually append the reverse complement of each sequence
default: no
-pl specify prefix length for bucket sort
recommendation: use without argument;
then a reasonable prefix length is automatically determined.
default: 0
-dc specify difference cover value
default: 0
-spmopt optimize esa-construction for suffix-prefix matching
default: 0
-memlimit specify maximal amount of memory to be used during index
construction (in bytes, the keywords 'MB' and 'GB' are allowed)
default: undefined
-kys output/sort according to keys of the form |key| in fasta header
default: nosort
-dir specify reading direction (fwd, cpl, rev, rcl)
default: fwd
-suf output suffix array (suftab) to file
default: no
-lcp output lcp table (lcptab) to file
default: no
-bwt output Burrows-Wheeler Transformation (bwttab) to file
default: no
-bck output bucket table to file
default: no
-v be verbose
default: no
-showprogress show a progress bar
default: no
-ii specify existing encoded sequence
default: undefined
-help display help for basic options and exit
-help+ display help for all options and exit
-version display version information and exit