2020 3/37 タイトル修正
Antimicrobial resistance(AMR)は、公衆衛生に対する脅威の増加である。 AMRを決定する現在の方法は、非効率的な表現型アプローチに依存しており、多くの病原体と抗菌薬の組み合わせのAMRメカニズムの理解が不完全なままとなっている。多様な細菌の高密度ゲノムデータの可用性が急速に継続的に増加していることを考えると、表現型を予測するためにゲノム情報を利用できるアルゴリズムの開発は臨床的に有用であり、これまで認識されていなかったAMRパスウェイの発見を支援する可能性がある。 DNA変異と表現型AMRの関係を理解しやすくするために、新しいバイオインフォマティクスツールvariant mapping and prediction of antibiotic resistance (VAMPr) を開発した。これは(1)タンパク質バリアントの遺伝子オルソログに基づく配列の特徴を導き出し、(2)AMRとの既知または新規の関連についてこれらの説明可能な遺伝子レベルのバリアントを調査し、 (3)正確なモデルを構築して、全ゲノムシーケンスデータに基づいてAMRを予測する。 29種類の抗生物質のAMR表現型を含む9 speciesから、3,393の単離された細菌分離株の公開シーケンスデータをキュレーションした。 14,615のバリアント遺伝子型を検出し、93の関連モデルと予測モデルを構築した。関連モデルにより、blaKPCやカルバペネム耐性などの既知の遺伝的抗生物質耐性メカニズムが、アプローチの性質と一致することが確認された。予測モデルは、ネストされた交差検証を通じて内部的に高い精度(すべての抗生物質と病原体の組み合わせで91.1%の平均精度)を達成し、外部臨床データセットを使用して検証された。 VAMPrバリアントの検出方法、関連付け、および予測モデルは、臨床応用の可能性を秘めた基礎科学者にとってのAMR研究の貴重なツールとなる。
HP
https://qbrc.swmed.edu/softwares.php
Documentation
https://cdc.biohpc.swmed.edu/VAMPr/VAMPr.cgi

HPより転載
ローカル環境へのインストール
依存
- Perl - https://www.perl.org
- R - http://www.r-project.org
- Perl module Bio::DB::Fasta - https://metacpan.org/pod/Bio::DB::Fasta
- Perl module Statistics::R - https://metacpan.org/pod/Statistics::R
- R library caret - https://cran.r-project.org/web/packages/caret/index.html
- R library xgboost - https://cran.r-project.org/web/packages/xgboost/index.html
- DIAMOND - https://github.com/bbuchfink/diamond
- Linux commands: sort, wget - https://www.gnu.org/software/wget/
#perl library
cpanm Bio::DB::Fasta
cpanm Statistics::R
#R library. Rにて
> install.packages("caret", dependencies = c("Depends", "Suggests"))
> install.packages("xgboost")
#Diamond
conda install -c bioconda -y diamond
本体 Gihub
git clone https://github.com/jiwoongbio/VAMPr.git
cd VAMPr/
> perl VAMP.pl -h
$ perl VAMP.pl -h
Usage: perl VAMP.pl [options] genome.fasta [variant.vcf [sample [...]]] > VAMP.txt
Options: -h display this help message
-t DIR directory for temporary files [$TMPDIR or /tmp]
-C STR codon and translation e.g. ATG=M [NCBI genetic code 11 (Bacterial, Archaeal and Plant Plastid)]
-S STR comma-separated start codons [GTG,ATG,CTG,TTG,ATA,ATC,ATT]
-T STR comma-separated termination codons [TAG,TAA,TGA]
-L INT minimum translation length [10]
-p INT number of threads [1]
-e FLOAT maximum e-value to report alignments [10]
-c FLOAT minimum coverage [0.8]
-s FILE output SAM file
-a FILE output alignment file
-A all variants
データベースの準備
perl VAMP_database.pl

https://cdc.biohpc.swmed.edu/VAMPr/VAMPr.cgi にアクセスする。
アセンブルされたゲノム配列(FASTAファイル)、またはproteome配列をアップロードする。

種を指定してサブミットする。
出力
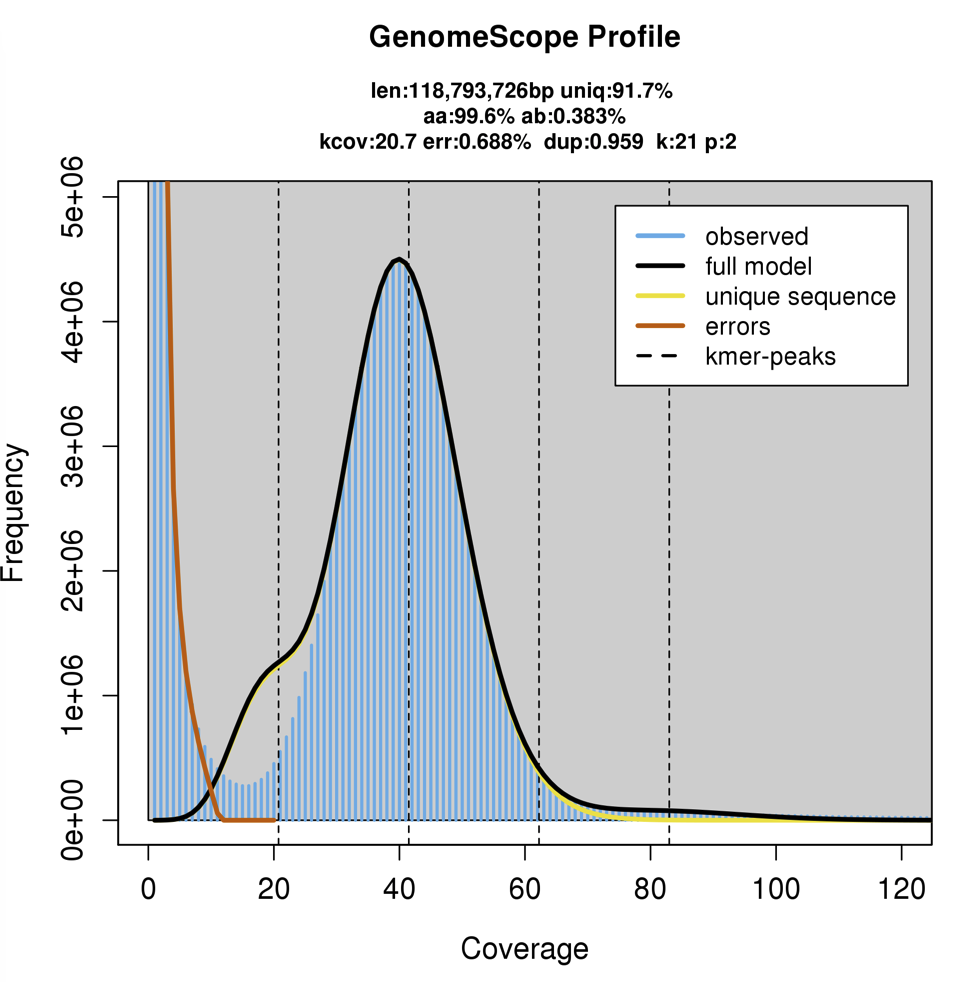
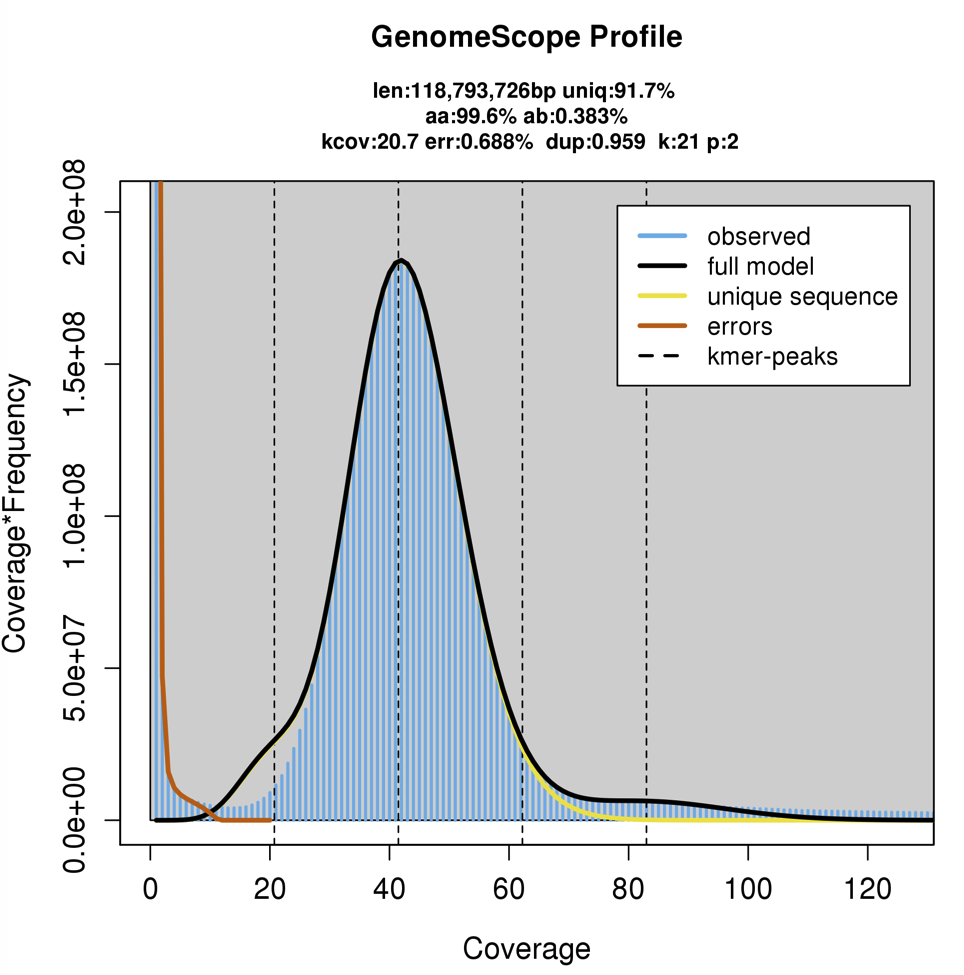
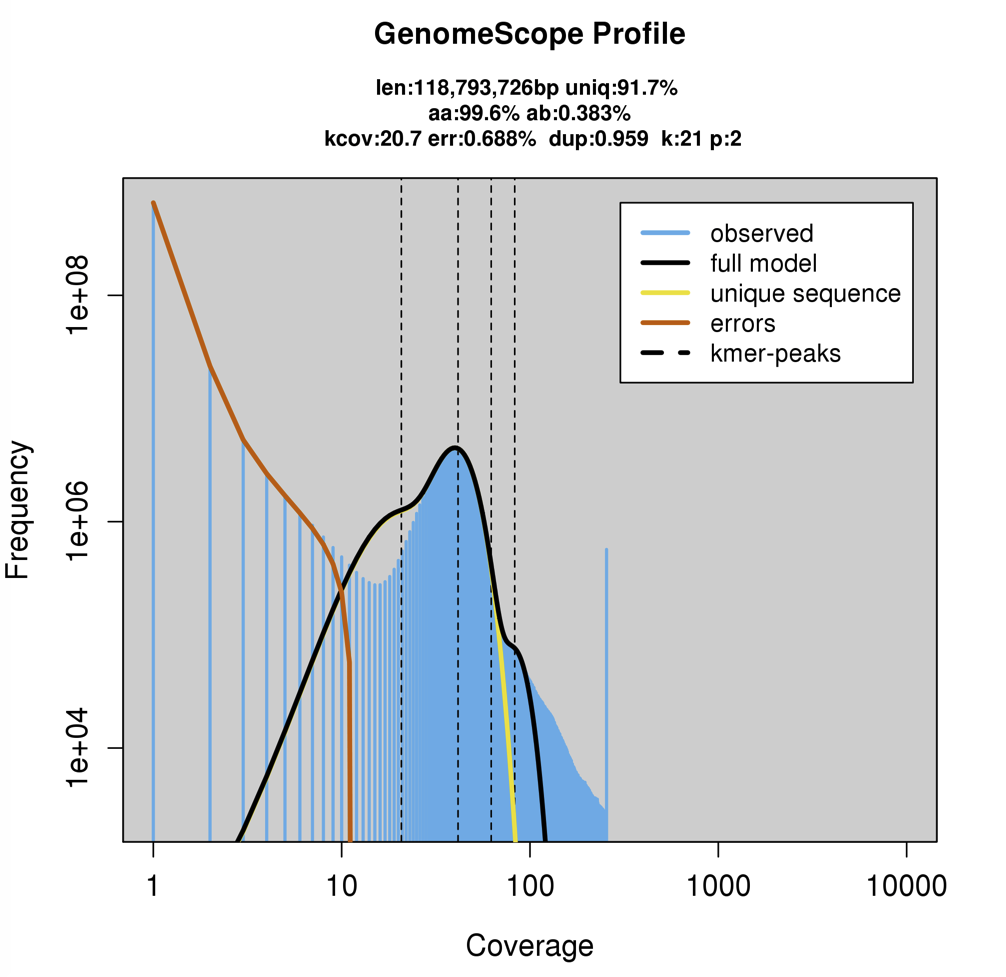
AMR遺伝子が含まれているかどうか調べられる。含まれている場合、AMR遺伝子のバリアントが調べられる。それから予測モデルに配列のバリアントが読み込まれ、抵抗性の確率が出力される。

Antibiotics耐性予測結果がまとめられている。エビデンスは計算されたオッズ比とFisherの正確検定によるP値に基づく。
データベース構築手順と耐性モデルの構築については論文に記載されています。確認して下さい。
引用
VAMPr: VAriant Mapping and Prediction of antibiotic resistance via explainable features and machine learning
Jiwoong Kim , David E. Greenberg , Reed Pifer, Shuang Jiang, Guanghua Xiao, Samuel A. Shelburne, Andrew Koh, Yang Xie, Xiaowei Zhan
PLoS Comput Biol. 2020 Jan 13;16(1):e1007511