2019 6/26 誤字修正
メタゲノミックショットガンシークエンシングは、関心のある微生物混合群からDNAを抽出し、無作為に抽出されたDNAをディープシーケンシングする。これは、特定の標的遺伝子領域が増幅およびシーケンシングされるマーカー遺伝子シーケンシング(例えば、細菌の16S rRNA遺伝子)とは対照的である。メタゲノムシーケンシングは、特にウイルスおよびバクテリオファージコミュニティの研究において、微生物学において重要な洞察を可能にしました[ref.1–9]。ただし、継続的な課題は、結果として得られる大規模データセットを標準的で信頼性の高い方法で分析および解釈することである[ref.16–23]。
微生物のメタゲノムを調べる一般的な方法は、イルミナのシークエンシング技術を使用して、目的のサンプルから単離した断片化DNAから多数の短い(100〜250塩基対)リードを取得することである。シーケンスの取得後、シーケンスを使用して基礎となる生物学的洞察を得る前に、いくつかの後処理ステップを実行する必要がある[ref.21、24]。
研究者はそれぞれの後処理ステップを実行するための多くのツールを自由に使えるようにしているが、その結果として、得られるアウトプットや下流の分析を変える可能性がある各ツールそれぞれに様々なパラメータが存在する。分析間でパラメータ、ツール、またはリファエンスデータベースのバージョンを変えると、異なるメタゲノムシーケンシング実験の結果を比較するのが難しくなる[ref.25]。逆に、研究間で一貫したワークフローを採用することで、実験は比較可能であり、下流分析は再現可能であることが保証される[ref.21]。使用されるソフトウェア、データベース、およびパラメータの文書化は、このプラクティスの重要な要素である。そうでなければ、一貫した再現可能なワークフローの利点が失われる。
メタゲノムのポストプロセッシングワークフローは、その有用性と柔軟性を最大にするために以下の特性を持つべきである。それは共有コンピューティングシステムとクラウドにデプロイ可能であるべきであり、ソフトウェアパラメータとリファレンスデータベースの簡単な設定を許容する。中断の後に再開する能力、それは不必要なステップがスキップされるか無視されることができるようにモジュール式であるべきである、そしてそれは新しい手順がユーザによって加えられることを可能にするべきである。機関プラットフォームとクラウドプラットフォームの両方にワークフローを展開する機能により、さまざまなラボでさまざまなコンピューティング設定でワークフローを繰り返すことができ、研究者が機関とクラウドのコンピューティングリソースを柔軟に選択できるようになる。同様に、設定ファイルを使用して実行中のパラメータを記録する機能により、実験固有のソフトウェアパラメータを使用することができ、今後の参考資料として使用できる。
いくつかの機能が効率的なデータ分析に貢献する。ワークフローのエラーや中断が最初からやり直す必要がない場合は、有益である。シーケンス実験では大量のデータが生成されるため、データ処理のステップを繰り返す必要があり、時間と費用がかかる可能性がある。さらに、ワークフローのすべてのステップがすべての実験に必要なわけではなく、実験によってはカスタム処理が必要な場合もある。実験を適切に処理するために、ワークフローは不要なステップをスキップし、必要に応じて後で実行する簡単な方法を提供するべきである。フレームワークを広く有用にするために、ユーザーは必要に応じてワークフローに新しいステップを簡単に追加して他のユーザーと共有できなければならない。これらの目標の多くを達成するパイプラインがいくつか開発されているが[ref.26-29]、メタゲノムデータセットの処理における柔軟性の向上と分析の長期的な再現性に対する我々(本著者ら)のニーズを満たしていなかった。
ここでは、メタゲノムシーケンス実験から一貫した一連の後処理済みファイルを生成する、簡単に配置および構成可能なパイプラインであるSunbeamを紹介する。 Sunbeamは自己完結型で、管理者権限なしでGNU / Linuxシステムにインストールできる。 Snakemakeワークフロー言語での実装により、エラー処理、タスク再開、および並列コンピューティング機能を備えている[ref.30]。ほぼすべての手順は事前に指定された妥当なデフォルト値を使用して構成可能であるため、広範なパラメータ調整を行わなくても迅速に展開できる。 Sunbeamは、ローカルデータを直接使用することも、NCBIのシーケンスリードアーカイブ(SRA)からの外部データを使用することもできる[ref.31]。 Sunbeamは、新しい手順をユーザーが追加できるようにする単純なメカニズムを使用して拡張可能である。
さらに、Sunbeamは、低品質またはホスト由来の配列を多く含む困難なサンプルのデータを処理できるカスタムソフトウェアを備えている。これらには、任意の数の宿主ゲノムまたは汚染ゲノム配列に対するカスタム調整された宿主由来のリード除去ステップ、およびKomplexity、問題のある低複雑性リードを下流の分析の前に迅速かつ正確に除去する新しい配列複雑性分析プログラムが含まれる。マイクロサテライトDNA配列は、ヒトゲノムのかなりの部分を占めており、個体間で非常に多様であり[ref.32-34]、単一のリファレンスゲノムに対するアラインメントによってそれらを除去することの難しさを増している。 Komplexityを開発したのは、ヌクレオチド配列の複雑さを分析するための既存のツール[ref.35-37]が、スピード、怪しいヒットの除去、およびfastqファイルのネイティブ処理の点で、本著者らのニーズを満たしていなかったためである。ここでは宿主関連の low-microbial biomass body sites [ref.19、38、39]、virome[ref.40]、およびマイクロバイオームの縦断サンプリング[ref.41、42]の公表され進行中の研究においてSunbeamを使用した。
SunbeamはPython、Bash、およびRustで実装されている。 GPLv3でライセンスされている。https://github.com/sunbeam-labs/sunbeamで自由に利用可能であり、ドキュメントはhttp://sunbeam.readthedocs.ioで利用可能である。
Sunbeamは、「可用性と要件」に記載されているハードウェアとソフトウェアの初期要件を満たすGNU / Linuxディストリビューションにインストールできる。インストールは、リポジトリからソフトウェアをダウンロードして「install.sh」を実行することによって実行される。Sunbeamは、インストールまたは実行に管理者権限を必要としない。 Sunbeamの基本的な分析ワークフローをDebian 9、 CentOS 6と7、Ubuntu 14.04、16.04、18.04、および18.10、 Red Hat Enterprise 6および7、そしてSUSE Enterprise 12にインストールして実行できることを確認した。
SunbeamはCondaパッケージ管理システム[ref.43]を利用して、「可用性と要件」のセクションに記載されているもの以外にも追加のソフトウェア依存関係をインストールする。 Condaは、依存関係の自動解決を提供し、管理者以外のユーザーにソフトウェアのインストールを容易にし、追加のソフトウェアをパッケージ化してサードパーティのソフトウェアチャネルを管理するための標準化されたシステムを使用する。 Condaは、パイプラインの外側にある既存のソフトウェアとの競合を避けるために、追加のソフトウェア依存関係解決のための分離された環境も提供する。必要に応じて、CondaはSunbeamインストールスクリプトによってインストールされる。 Sunbeamが使用する分離ソフトウェア環境もインストールスクリプトによって作成される。
Sunbeamパイプラインへの入力は、ローカルのファイルまたはSRAを通じて利用可能なサンプルのいずれかで、raw Illuminaシーケンスリードからなる。デフォルトでは、Sunbeamはリードに対して次の順序で予備操作を実行する。
クオリティ管理:オプションで、grabseq [ref.44]とsra-tools [ref.31]を使ってSRAからリードをダウンロードする。アダプター配列を除去し、そしてCutadapt [ref.45]およびTrimmomatic [ref.46]ソフトウェアを用いて塩基をクオリティフィルタリングする。クオリティフィルタリングを通過したリードペアは保持される。クオリティはFastQCを使用して評価され[ref.47]、別々のレポートにまとめられる。
低複雑度マスキング:各リードにおける配列複雑度は、後述する新規複雑度スコアリングアルゴリズムであるKomplexityを使用して評価される。ユーザーがカスタマイズ可能なシーケンス複雑度のしきい値を下回るリードは削除される。削除されたリード数のログは後で検査するために書き込まれる。
ホストリード除去:リードはbwaを使用してユーザー指定のホストまたは汚染物質シーケンスのセットに対してマッピングされる。特定の同一性および長さのしきい値内でこれらのシーケンスのいずれかにマップされるリードは削除される。削除されたリード数は後で検査するために記録される。
初期クオリティ管理プロセスの後、複数のオプションの下流工程を並行して実施することができる。分類ステップでは、汚染配列が除去されクオリティ管理されたリードがKraken [ref.49]を使用して分類的に分類され、タブ区切り形式とBIOM [ref.50]形式の両方で要約される。アセンブリステップでは、MEGAHIT [ref.51]を使用して、各サンプルからのリードを連続アセンブリする。事前に指定された長さを超えるコンティグには、あのテーションが付けられる。オープンリーディングフレーム(ORF)は、Prodigal [ref.52]を用いて予測される。次に、コンティグ全体(および関連するORF)を、コンティグ全体および推定ORFの両方を使用して、任意の数のユーザー指定のヌクレオチドまたはタンパク質BLAST [ref.53]データベースに対して検索する。結果は各サンプルのレポートにまとめられる。最後に、マッピングステップでは、bwa [ref.48]を使用してクオリティ管理されたリードを任意の数のユーザー指定のリファレンスゲノムまたは遺伝子セットにマッピングし、結果のBAMファイルをSAMtools [ref.54]を使用してソートおよびインデックス付けする。
Sunbeamは、出力ファイルを概念的に異なるフォルダにグループ化し、論理的な出力フォルダ構造を提供するよう設計されている。 Sunbeamからの標準出力には、クオリティ管理プロセスの各ステップからのfastqファイル、各リードの分類系統アサイン、各サンプルからアセンブリされたコンティグ、遺伝子予測、およびすべてのクオリティ管理されたリードのアラインメントファイルが含まれる。ほとんどのルールは、後の検査と要約のためにその操作のログを作成する。ユーザーは特定の出力を個別にまたはグループとして要求でき、パイプラインは必要なファイルを作成するのに必要なステップのみを実行する。これにより、ユーザーはモジュール式にパイプラインの任意の部分をスキップまたは再実行できる。
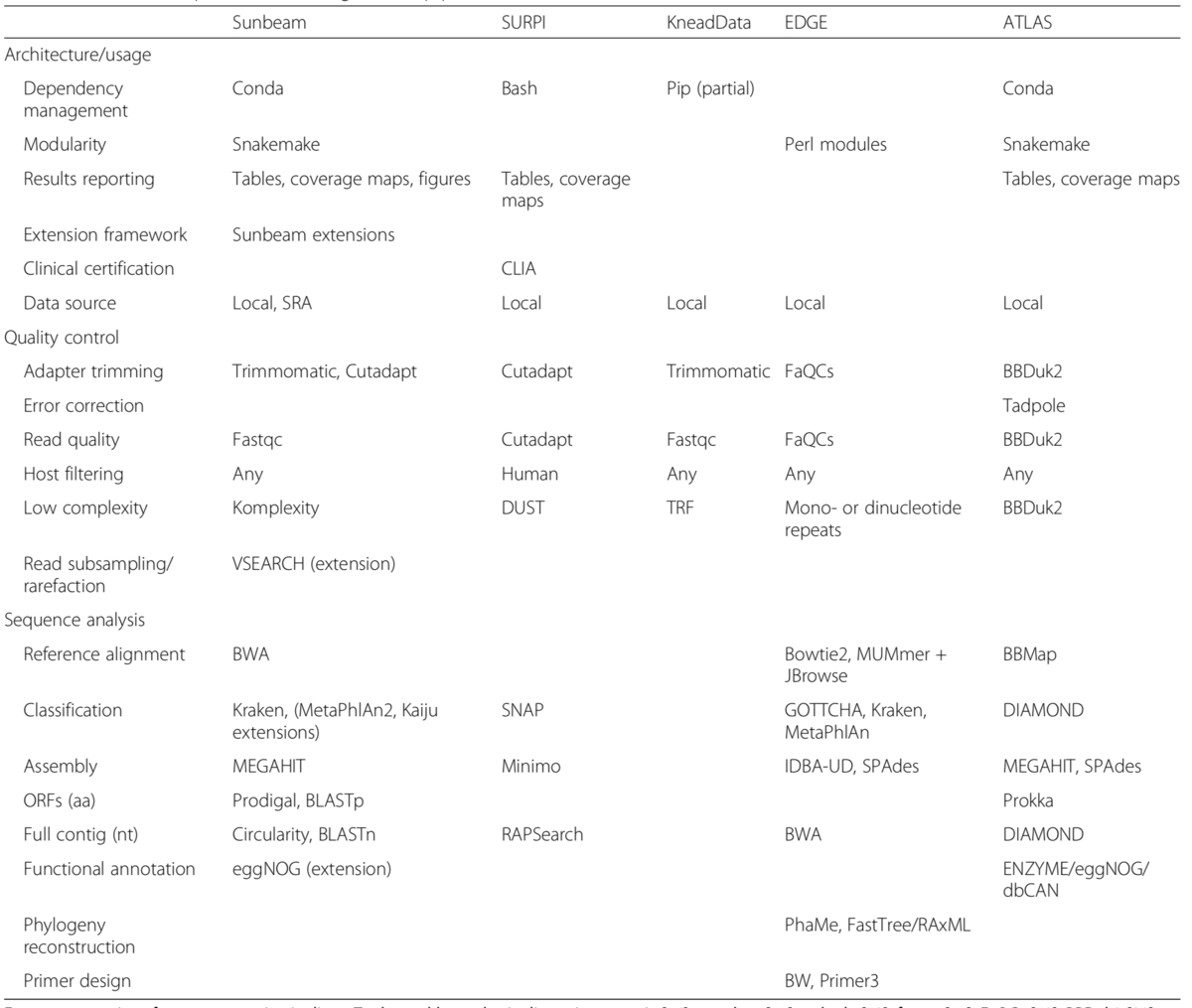
表1、他のツールとの機能比較
論文より転載
以下の作業が自動化されている (Githubより)
- Quality control, including adaptor trimming, host read removal, and quality filtering;
- Taxonomic assignment of reads to databases using Kraken;
- Assembly of reads into contigs using Megahit;
- Contig annotation using BLAST[n/p/x];
- Mapping of reads to target genomes; and
- ORF prediction using Prodigal.
Documentation
https://sunbeam.readthedocs.io/en/latest/
インストール
ubuntu16.0.4のminiconda3-4.3.30環境でテストした。
依存
- python3.6 ~ 3.7
Gihutb
git clone -b stable https://github.com/sunbeam-labs/sunbeam sunbeam-stable
cd sunbeam-stable
#condaがない環境で実行するとcondaも含めて導入される
./install.sh
#condaをここで入れたなら
echo "export PATH=$PATH:/root/miniconda3/bin" >> ~/.bashrc && source ~/.bashrc
source activate sunbeam
テスト
#テストラン時に出た文字化けエラーはC.UTF-8をexportして応急処置
export LC_ALL=C.UTF-8
export LANG=C.UTF-8
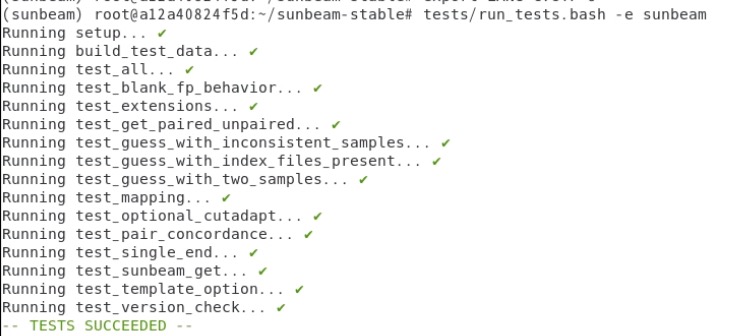
> tests/run_tests.bash -e sunbeam
> sunbeam
# sunbeam
usage: sunbeam [-h/--help,-v/--version] <subcommand>
subcommands:
init Create a new config file for a project using local or SRA data.
run Execute the pipeline.
config Modify or update config files.
list_samples Make a list of samples from a directory.
optional arguments:
-v, --version show program's version number and exit
For more help, see the docs at http://sunbeam.readthedocs.io.
Unrecognized command.
> sunbeam init -h
# sunbeam init -h
usage: init [-h] [-f] [--output FILE] [--defaults FILE] [--template FILE]
[--data_fp PATH] [--format STR] [--single_end]
[--data_acc ACC [ACC ...]]
project_fp
Initialize a new Sunbeam project in a given directory, creating a new config
file and (optionally) a sample list. The sample list source can be either a
folder of input files or a list of SRA accession numbers.
positional arguments:
project_fp project directory (will be created if it does not
exist)
optional arguments:
-h, --help show this help message and exit
-f, --force overwrite files if they already exist
--output FILE name of config file (sunbeam_config.yml)
config file options:
--defaults FILE set of default values to use to populate config file
--template FILE custom config file template, in YAML format
sample list options:
Options to automatically generate a sample list. --data_fp (for reading
filenames from a specified folder) and --data_acc (for fetching SRA
accession numbers to be downloaded during a run) are mutually exclusive.
If --data_acc is used, all SRA Run (sample) entries corresponding to the
given accession numbers (e.g. PRJNA###, SAMN###, SRR###) will be used to
create the sample list. Note in this case project_fp should either be
given before --data_acc or separated by a '--' to distinguish it from an
accession number.
--data_fp PATH path to folder containing fastq.gz files
--format STR filename format for --data_fp (default: guessed)
--single_end fastq files are in single-end, not paired-end, format
for --data_fp
--data_acc ACC [ACC ...]
list of SRA-compatible accession numbers
> sunbeam run -h
# sunbeam run -h
usage: sunbeam run [options] -- <snakemake options>
Executes the Sunbeam pipeline by calling Snakemake.
optional arguments:
-h, --help show this help message and exit
-s SUNBEAM_DIR, --sunbeam_dir SUNBEAM_DIR
Path to Sunbeam installation
You can pass further arguments to Snakemake after --, e.g:
$ sunbeam run -- --cores 12
See http://snakemake.readthedocs.io for more information.
> sunbeam config -h
# sunbeam config -h
usage: sunbeam config [-h] {update,modify} ...
positional arguments:
{update,modify}
optional arguments:
-h, --help show this help message and exit
> sunbeam list_samples -h
# sunbeam list_samples -h
usage: sunbeam list_samples [-h] [-s] [-f STR] data_fp
positional arguments:
data_fp Path to folder containing reads
optional arguments:
-h, --help show this help message and exit
-s, --single_end Samples are single-end (not paired-end)
-f STR, --format STR Filename format (e.g. {sample}_R{rp}.fastq.gz).
(default: guessed)
最小限の手間でパイプラインにツールを追加することも可能になっている(未テスト)。
#sunbeam内にて
git clone https://github.com/sunbeam-labs/sbx_kaiju extensions/sbx_kaiju
cd extensions/sbx_kaiju/
conda install -c bioconda --file requirements.txt
#databaseをダウンロード後、ymlファイルにパスを追加。(kaiju github)
mkdir kaijudb
cd kaijudb
kaiju-makedb -s (database name)
#それからrun前にymlファイルに追加する
cat extensions/sbx_kaiju/config.yml >> my_config.yml
データベースの準備
1、kraken database(version.1.0)
ここではpre-build (2017)のMiniKraken DB_8GBデータベースを使う。
mkdir /data/kraken
cd /data/kraken/
wget https://ccb.jhu.edu/software/kraken/dl/minikraken_20171019_8GB.tgz
tar -zxvf minikraken_20171019_8GB.tgz && rm minikraken_20171019_8GB.tgz
ymlファイルのclassify:kraken_db_fp: にパスを記載する。上の通りなら
kraken_db_fp: '/data/kraken/minikraken_20171019_8GB'
2、filtering of contaminated sequences
コンタミネーション(ホストゲノムやテクニカルな汚染配列) の除去。例えばヒトゲノムの汚染を除く。リファレンスの拡張子はfastaにする必要がある。
mkdir /data/contamination_genome
cd /data/contamination_genome/
wget http://hgdownload.soe.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz
unpigz -c hg38.fa.gz -p 8 > hg38.fasta && rm hg38.fa.gz
ymlファイルのhost_fp: にパスを記載する。上の通りなら
host_fp: '/data/contamination_genome'
3、blast(n、p、x)
blastしてアノテーションをつけることができる。ここではNCBI NTを使う。抗生物質耐性遺伝子、virulance factor、NCBI nt、など目的に応じてデータベースを構築しておく。
mkdir /data/annotation_database/
cd /data/annotation_database/
#NCBI ntを入れる (容量に注意)
mkdir nt
cd nt
wget "ftp://ftp.ncbi.nlm.nih.gov/blast/db/nt.*.tar.gz"
for a in nt.*.tar.gz; do tar xzf $a; done
#CARDデータベース
mkdir /data/annotation_database/card
#調べてから追記します
#VFDBータベース
ymlファイルの blastdbs以下に記載する。ここではproteinは載せていないのでblastpは実行されない。
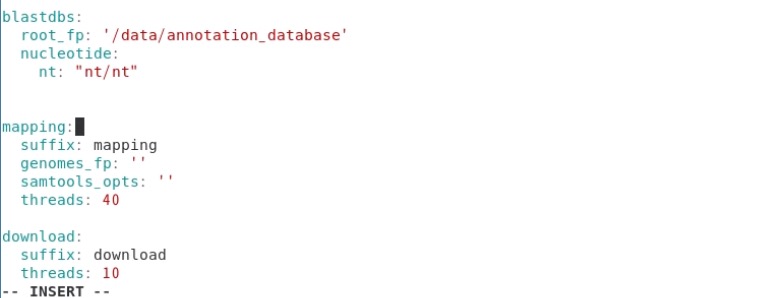
ダウンロードしたntは70以上に分かれているが、sunbeamはnt/ntで認識する。ここではblastデータベースは全て/data/annotation_database/にあり、その下位ディレクトリのnt/にNCBI ntデータベースファイルがあるという設定で進めている。
下の画像の通り、 nt/にはダウンロードし解凍したntのblastデータベースが保存されている。
実行方法
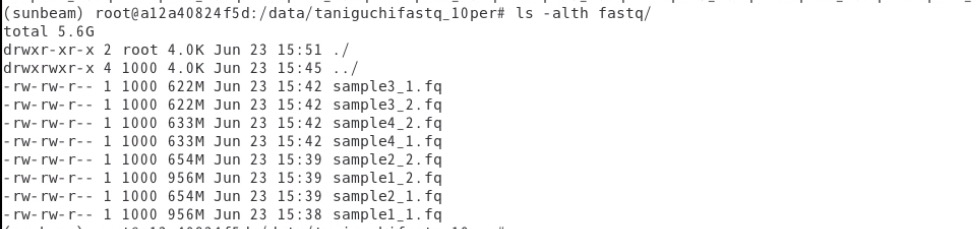
1、fastqの準備。
特定のディレクトリに解析対象のfastqをまとめる。 例えばこのようなサンプルを分析する。ファイル名は推測できるような名前になっていないといけない。下のファイル名だと問題なく認識した(sample1, sample2, ...)。
2、ランのconfigファイル作成。
ここではfastqを保存しているディレクトリを/sequencing/fastq、プロジェクトファイル名はmy_project1としている。
sunbeam init my_project1 --data_fp /sequencing/fastq
my_project1ディレクトリができ、その中にsamples.csvとsunbeam_config.ymlができる。
3、.ymlの編集。他のツールのconfiguration fileに相当するymlファイルができるので、ラン前に編集する。
vim my_project1/sunbeam_config.yml
sunbeam_config.yml
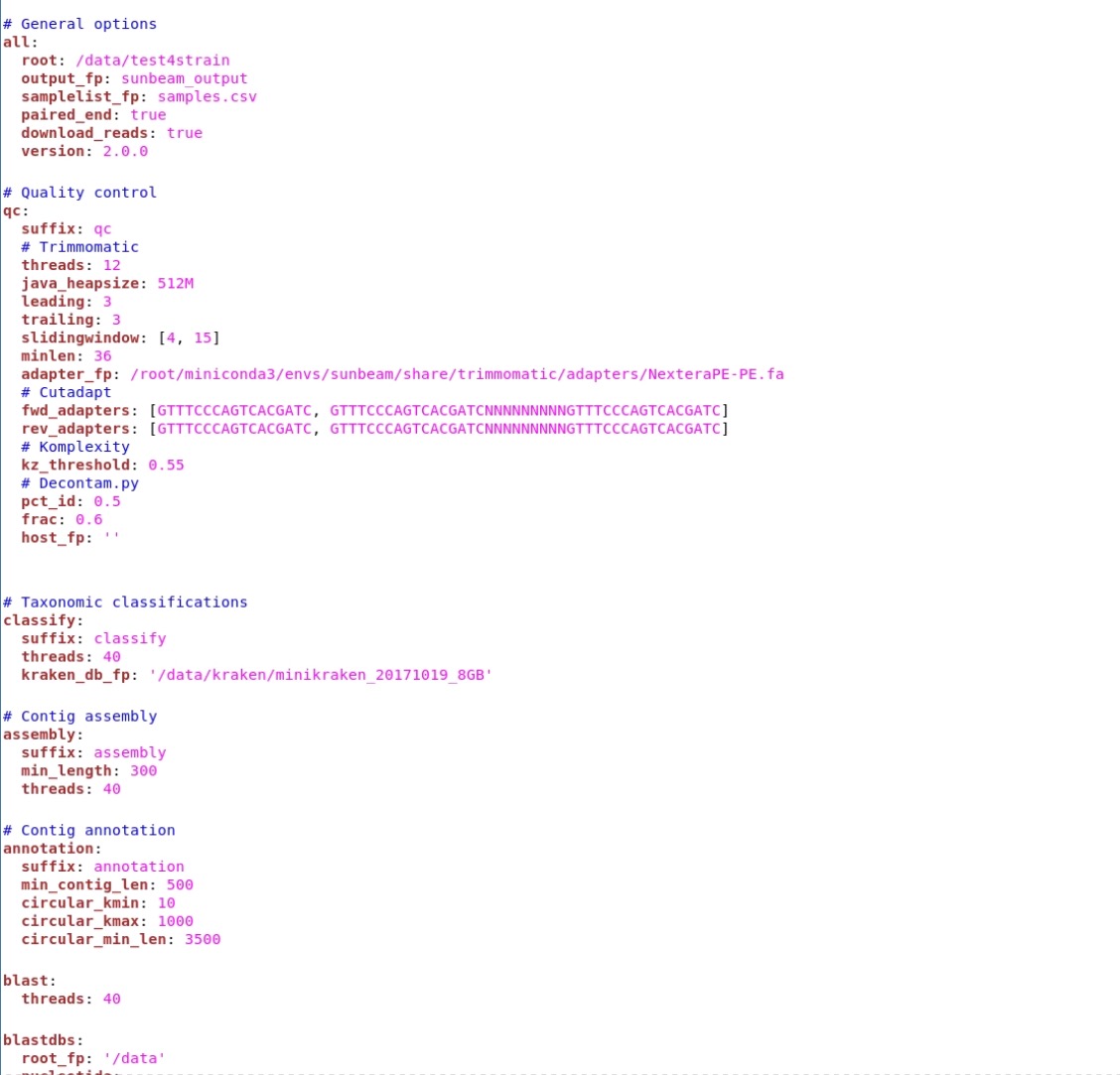
上から順番に見ていって、パラメータを変更していく。モジュラー設計のパイプラインなのでステップを丸ごと追加・削除することもできる。
4、実行する。-nでdry run
sunbeam run --configfile my_project1/sunbeam_config.yml
出力
SRAデータを使ったテストラン
例えばmetagenome 4サンプルを分析する。
mkdir ~/test4strain
sunbeam init /data/test4strain/ --data_acc ERR3277301 ERR3277295 ERR3277320 ERR3277274
#.ymlとsample.csvができる。
#~/testにできた.ymlを指定してラン
sunbeam run --configfile /data/test4strain/sunbeam_config.yml
ymlファイルで出力defaultは"subbeam_output"になっている。
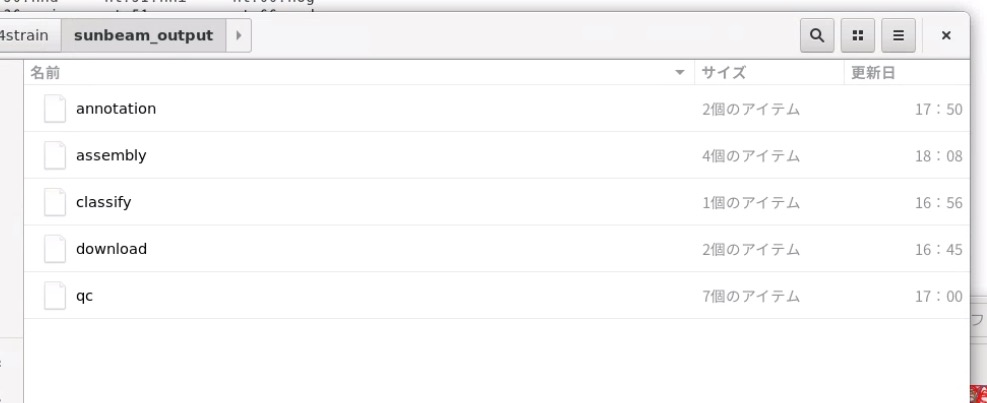
ラン後、subbeam_outputを開く。
dowload、qc、assembly、classify、annotationに分かれている。
download/にはダウンロードされたfastqが収められている。

qc/にはcutadapt、trimmomaticによるアダプター除去/QT、low comlexility reads除去、ホストコンタミネーションリード除去後のそれぞれのfastqが収められている。したがってかなりサイズが大きくなる。
qc/reportsにはQC結果のレポート
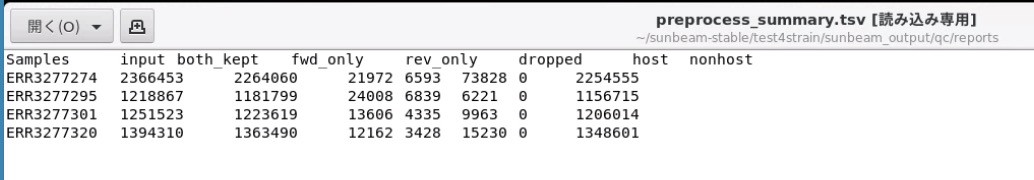
classify/にはdefaultではkrakenによる分析結果が収められている。
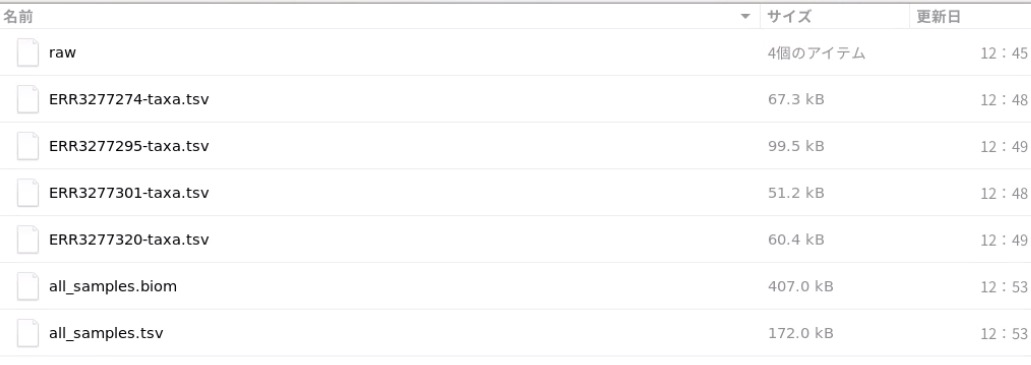
全サンプル結果はBIOM形式とTSV形式でまとめられる。
all_samples.tsv

biomはqiimeやRのphyloseqなどで可視化できる。
assembly/にはmegahitのcontigsやカバレッジファイルが収められている。
全サンプルの全contigのカバレッジやサイズレポート
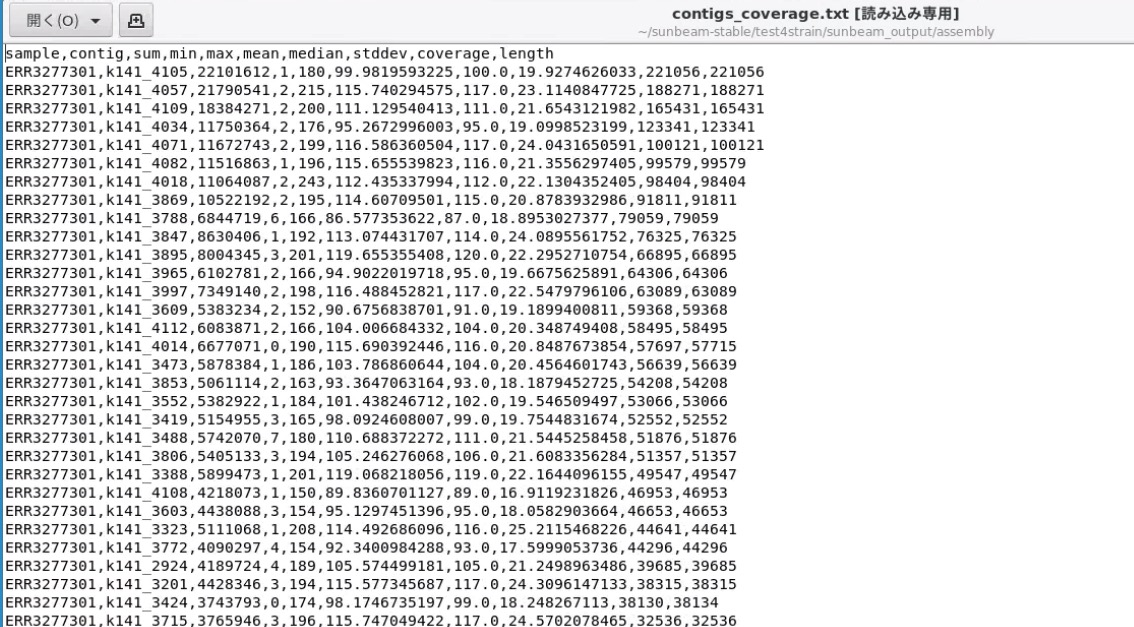
アノテーションにはユーザーが指定したblastデータベースによるアノテーション結果が収められる。
snakemakeのスケーラビリティにより、シングルノードサーバーから、クラスターの分散メモリ環境でも特別なチューニングなしにランできるようになっています。パラメータは
https://sunbeam.readthedocs.io/en/latest/usage.html#configuration
のCluster optionsで確認して下さい。
引用
Sunbeam: an extensible pipeline for analyzing metagenomic sequencing experiments
Clarke EL, Taylor LJ, Zhao C, Connell A, Lee JJ, Fett B, Bushman FD, Bittinger K
Microbiome. 2019 Mar 22;7(1):46.