2019 3/11 タイトル修正
2019 3/12 コマンド追記、誤ったコメント削除
Albacore software overview(2019 3/10確認)
Albacoreは、Oxford Nanoporeベースコールアルゴリズム、およびいくつかの後処理ステップを提供するデータ処理パイプラインである。 Windows、Mac OS X、および複数のLinuxプラットフォームでコマンドラインから実行できる。Albacoreパイプラインは以下を含む。
1、Basecalling: MinKNOW basecallingに見られるのと同様のアルゴリズムの実装。ただし、MinKNOWでは現在処理されていないベースコールケミストリ用の設定ファイルも含まれている( e.g. 1D2 reads)。
2、 Calibration Strand Detection: 読み取り値は、内蔵されたminimap2アライナを介してキャリブレーションストランドの基準に対して調整される。キャリブレーションストランドは、ナノポアと実験の品質管理として機能する。現在の読み取り値がキャリブレーションストランドであると識別された場合、バーコードステップやアライメントステップは実行されない。
3、Barcoding/Demultiplexing: 各ストランドの始めと終わりは、オックスフォードナノポアテクノロジーズによって現在提供されているバーコードに対してアライメントされ、バーコードの結果によってdemultiplexingされる。
4、Alignment: ユーザーは、FASTA、lastdb、またはminimap2インデックス形式のリファレンスファイルを提供できる。この場合、リードは統合されたminimap2アライナを介してこの参照に対してアライメントされる。
現在の仮定と制限
このソフトウェアは現在、linear(1D)または1D2ケミストリのベースコールを提供している。このソフトウェアへの入力として使用されるリードの.fast5ファイルには、生データが含まれている必要がある。 MinKNOWソフトウェアによって生成されたリードファイルには、生データがデフォルトで含まれているので、ユーザーが古いファイルを処理しようとしない限り、これは問題にはならない。
ベースコール速度
ベースコールにかかる時間は、コンピューターの仕様、割り当てられたスレッド数、および分析されたリード数によって異なる。ガイドラインに従うと、1Dベースコールは、リード長の中央値7 kbで、毎分約90リード実行できる。
manual(サード?)
https://denbi-nanopore-training-course.readthedocs.io/en/latest/basecalling/basecalling.html
Release notes (2019 3/9)
インストール
docker(公式イメージ使用)と、mac os 10.12 python3.6.1環境でfast5のサブディレクトリ1つだけ使ってテストした。
version2.3.4のハードウエア要件
Albacoreを実行するための一般的なシステム要件
4 GBのRAMと1 Dベースコール用のワーカースレッドあたり1 GB
4 GBのRAMと1 D2ベースコール用のワーカースレッドあたり2 GB
.debまたは.msiインストーラーの管理者アクセス権(オプションで.whlの場合)
インストール用に最大100 MBのドライブスペース、ベースコールされたファイル用に最低512 GBのストレージスペース(1 TB推奨)
1Dベースコール:生データのみを含む.fast5ファイルから開始すると、ファイルサイズは元のサイズの約4.5倍に増加する。
version2.3.4のインストールソフトウエア依存
- 64-bit OSX 10.11 (El Capitan) or higher
- Python 3.6 (we recommend installing Python via brew)
- pip for Python 3 (this should come with your Python 3 installation)
- Python 3.4, 3.5 or 3.6
- pip for Python 3 version 8.1 or higher (depending on how this has been installed it may be referred to as "pip" or "pip3")
AlbacoreのダウンロードはOxford nanopore HPのsoftware downloadsから行う。
↑このページにジャンプするには購入用アカウントでlog inしておく必要あり。
インストール方法は上のページの各ツールのInstallation guideに書いてある。ここではdockerを使ってテストする。
docker pull genomicpariscentre/albacore
> read_fast5_basecaller.py -h
# read_fast5_basecaller.py -h
usage: read_fast5_basecaller.py [-h] [-l] [-v] [-i INPUT] -t WORKER_THREADS -s
SAVE_PATH [--resume [X]] [-f FLOWCELL]
[-k KIT] [--barcoding] [-c CONFIG]
[-d DATA_PATH] [-b] [-r]
[-n FILES_PER_BATCH_FOLDER] [-o OUTPUT_FORMAT]
[-q READS_PER_FASTQ_BATCH]
[--disable_filtering] [--disable_pings]
ONT Albacore Sequencing Pipeline Software
optional arguments:
-h, --help show this help message and exit
-l, --list_workflows List standard flowcell / kit combinations.
-v, --version Print the software version.
-i INPUT, --input INPUT
Folder containing read fast5 files (if not present,
will expect file names on stdin).
-t WORKER_THREADS, --worker_threads WORKER_THREADS
Number of worker threads to use.
-s SAVE_PATH, --save_path SAVE_PATH
Path to save output.
--resume [X] Resume previous run for the given save path. Optional
parameter X is for debugging purposes only.
-f FLOWCELL, --flowcell FLOWCELL
Flowcell used during the sequencing run.
-k KIT, --kit KIT Kit used during the sequencing run.
--barcoding Search for barcodes to demultiplex sequencing data.
-c CONFIG, --config CONFIG
Optional configuration file to use.
-d DATA_PATH, --data_path DATA_PATH
Optional path to model files.
-b, --debug Output additional debug information to the log.
-r, --recursive Recurse through subfolders for input data files.
-n FILES_PER_BATCH_FOLDER, --files_per_batch_folder FILES_PER_BATCH_FOLDER
Maximum number of files in each batch subfolder. Set
to 0 to disable batch subfolders.
-o OUTPUT_FORMAT, --output_format OUTPUT_FORMAT
desired output format, can be fastq,fast5 or only one
of these.
-q READS_PER_FASTQ_BATCH, --reads_per_fastq_batch READS_PER_FASTQ_BATCH
number of reads per FastQ batch file. Set to 1 to
receive one reads per file and file names which
include the read ID. Set to 0 to have all reads per
run ID written to one file.
--disable_filtering Disable filtering into pass/fail folders
--disable_pings Do not send summary information about the run
>read_fast5_basecaller.py -v
# read_fast5_basecaller.py -v
ONT Albacore Sequencing Pipeline Software (version 2.3.4)
テスト時のバージョンは2.3.4
#1D² chemistry
> full_1dsq_basecaller.py -h
# full_1dsq_basecaller.py -h
usage: full_1dsq_basecaller.py [-h] [-l] [-v] -i INPUT -t WORKER_THREADS -s
SAVE_PATH [--resume [REASON]] [--barcoding] -f
FLOWCELL -k KIT [-d DATA_PATH] [-b] [-r]
[-n FILES_PER_BATCH_FOLDER] [-o OUTPUT_FORMAT]
[-q READS_PER_FASTQ_BATCH]
[--disable_filtering] [--disable_pings]
ONT Albacore Sequencing Pipeline Software
optional arguments:
-h, --help show this help message and exit
-l, --list_workflows List standard flowcell / kit combinations.
-v, --version Print the software version.
-i INPUT, --input INPUT
Folder containing read fast5 files.
-t WORKER_THREADS, --worker_threads WORKER_THREADS
Number of worker threads to use.
-s SAVE_PATH, --save_path SAVE_PATH
Path to save output.
--resume [REASON] Resume previous run for the given save path. Optional
parameter X is for debugging purposes only.
--barcoding Search for barcodes to demultiplex sequencing data.
-f FLOWCELL, --flowcell FLOWCELL
Flowcell used during the sequencing run.
-k KIT, --kit KIT Kit used during the sequencing run.
-d DATA_PATH, --data_path DATA_PATH
Optional path to model files.
-b, --debug Output additional debug information to the log.
-r, --recursive Recurse through subfolders for input data files.
-n FILES_PER_BATCH_FOLDER, --files_per_batch_folder FILES_PER_BATCH_FOLDER
Maximum number of files in each batch subfolder. Set
to 0 to disable batch subfolders.
-o OUTPUT_FORMAT, --output_format OUTPUT_FORMAT
desired output format, can be fastq,fast5 or fast5.
Applies only to 1d part of process
-q READS_PER_FASTQ_BATCH, --reads_per_fastq_batch READS_PER_FASTQ_BATCH
number of reads per FastQ batch file. Set to 0 to
receive one reads per file and file names which
include the read ID.
--disable_filtering Disable filtering into pass/fail folders
--disable_pings Do not send summary information about the run
> paired_read_basecaller.py -h
# paired_read_basecaller.py -h
usage: paired_read_basecaller.py [-h] [-l] [-v] [-i INPUT] -f FILE_INDEX -t
WORKER_THREADS -s SAVE_PATH
[--resume [REASON]] [--barcoding] -c CONFIG
[-d DATA_PATH] [-b] [-r]
[-q READS_PER_FASTQ_BATCH]
[--disable_filtering] [--disable_pings]
ONT Albacore 1D-Squared Sequencing Pipeline Software
optional arguments:
-h, --help show this help message and exit
-l, --list_workflows List standard flowcell / kit combinations.
-v, --version Print the software version.
-i INPUT, --input INPUT
Folder containing read fast5 files (if not present,
will expect file names on stdin).
-f FILE_INDEX, --file_index FILE_INDEX
Summary file for indexing potential paired reads.
-t WORKER_THREADS, --worker_threads WORKER_THREADS
Number of worker threads to use.
-s SAVE_PATH, --save_path SAVE_PATH
Path to save output.
--resume [REASON] Resume previous run for the given save path. Optional
parameter X is for debugging purposes only.
--barcoding Search for barcodes to demultiplex sequencing data.
-c CONFIG, --config CONFIG
Configuration file to use.
-d DATA_PATH, --data_path DATA_PATH
Optional path to model files.
-b, --debug Output additional debug information to the log.
-r, --recursive Recurse through subfolders for input data files.
-q READS_PER_FASTQ_BATCH, --reads_per_fastq_batch READS_PER_FASTQ_BATCH
number of reads per FastQ batch file. Set to 0 to
receive one reads per file and file names which
include the read ID.
--disable_filtering Disable filtering into pass/fail folders
--disable_pings Do not send summary information about the run
サポートされているフローセルとkitの組み合わせの確認
> read_fast5_basecaller.py -l
# read_fast5_basecaller.py -l
Parsing config files in /opt/albacore.
Available flowcell + kit combinations are:
flowcell kit barcoding config file
FLO-MIN106 SQK-DCS108 r94_450bps_linear.cfg
FLO-MIN106 SQK-LRK001 r94_450bps_linear.cfg
FLO-MIN106 SQK-LSK108 r94_450bps_linear.cfg
FLO-MIN106 SQK-LSK109 r94_450bps_linear.cfg
FLO-MIN106 SQK-LWB001 included r94_450bps_linear.cfg
FLO-MIN106 SQK-LWP001 r94_450bps_linear.cfg
FLO-MIN106 SQK-PBK004 included r94_450bps_linear.cfg
FLO-MIN106 SQK-PCS108 r94_450bps_linear.cfg
FLO-MIN106 SQK-PSK004 r94_450bps_linear.cfg
FLO-MIN106 SQK-RAB201 included r94_450bps_linear.cfg
FLO-MIN106 SQK-RAB204 included r94_450bps_linear.cfg
FLO-MIN106 SQK-RAD002 r94_450bps_linear.cfg
FLO-MIN106 SQK-RAD003 r94_450bps_linear.cfg
FLO-MIN106 SQK-RAD004 r94_450bps_linear.cfg
FLO-MIN106 SQK-RAS201 r94_450bps_linear.cfg
FLO-MIN106 SQK-RBK001 included r94_450bps_linear.cfg
FLO-MIN106 SQK-RBK004 included r94_450bps_linear.cfg
FLO-MIN106 SQK-RLB001 included r94_450bps_linear.cfg
FLO-MIN106 SQK-RLI001 r94_450bps_linear.cfg
FLO-MIN106 SQK-RNA001 r941_70bps_rna_linear.cfg
FLO-MIN106 SQK-RPB004 included r94_450bps_linear.cfg
FLO-MIN106 VSK-VBK001 r94_450bps_linear.cfg
FLO-MIN106 VSK-VMK001 included r94_450bps_linear.cfg
FLO-MIN106 VSK-VSK001 r94_450bps_linear.cfg
FLO-MIN107 SQK-DCS108 r95_450bps_linear.cfg
FLO-MIN107 SQK-LRK001 r95_450bps_linear.cfg
FLO-MIN107 SQK-LSK108 r95_450bps_linear.cfg
FLO-MIN107 SQK-LSK109 r95_450bps_linear.cfg
FLO-MIN107 SQK-LWB001 included r95_450bps_linear.cfg
FLO-MIN107 SQK-LWP001 r95_450bps_linear.cfg
FLO-MIN107 SQK-PBK004 included r95_450bps_linear.cfg
FLO-MIN107 SQK-PCS108 r95_450bps_linear.cfg
FLO-MIN107 SQK-PSK004 r95_450bps_linear.cfg
FLO-MIN107 SQK-RAB201 included r95_450bps_linear.cfg
FLO-MIN107 SQK-RAB204 included r95_450bps_linear.cfg
FLO-MIN107 SQK-RAD002 r95_450bps_linear.cfg
FLO-MIN107 SQK-RAD003 r95_450bps_linear.cfg
FLO-MIN107 SQK-RAD004 r95_450bps_linear.cfg
FLO-MIN107 SQK-RAS201 r95_450bps_linear.cfg
FLO-MIN107 SQK-RBK001 included r95_450bps_linear.cfg
FLO-MIN107 SQK-RBK004 included r95_450bps_linear.cfg
FLO-MIN107 SQK-RLB001 included r95_450bps_linear.cfg
FLO-MIN107 SQK-RLI001 r95_450bps_linear.cfg
FLO-MIN107 SQK-RNA001 r941_70bps_rna_linear.cfg
FLO-MIN107 SQK-RPB004 included r95_450bps_linear.cfg
FLO-MIN107 VSK-VBK001 r95_450bps_linear.cfg
FLO-MIN107 VSK-VMK001 included r95_450bps_linear.cfg
FLO-MIN107 VSK-VSK001 r95_450bps_linear.cfg
FLO-PRO001 SQK-LSK109 r941_450bps_linear_prom.cfg
実行方法
フローセルとkitの種類、raw callのディレクトリパス等を指定する(*1)。例えばMinION/GridIONのR.9.4.1FLO-MIN106D(リンク)、Rapid barcoding lit(リンク)を使っているなら現行はSQK-RBK004なので、以下のようになる(2019 3/10現在)。
read_fast5_basecaller.py -f FLO-MIN106 -k SQK-LSK308 -t 24 \
-s /data/albacore_basecalled/ -o fastq -q 100000 \
-i /data/Ecoli_R7_ONI_flowcell_18/
-
-f Flowcell used during the sequencing run
-
-i Folder containing read fast5 files (if not present, will expect file names on stdin)
-
-s Path to save output
-
-t Number of worker threads to use
- -k Kit used during the sequencing run
-
-o desired output format, can be fastq,fast5 or only one of these
-
-q number of reads per FastQ batch file. Set to 0 to receive one reads per file and file names which include the read ID
重いので利用できるだけスレッドを指定した方が良いです(-t <INT>)。また、-o fast5でexportし、さらに他のbase callerに使うこともできる。
出力
failtとpassにそれぞれfastqができる(-o fastq)。-qで指定した数でfastqは分割される。
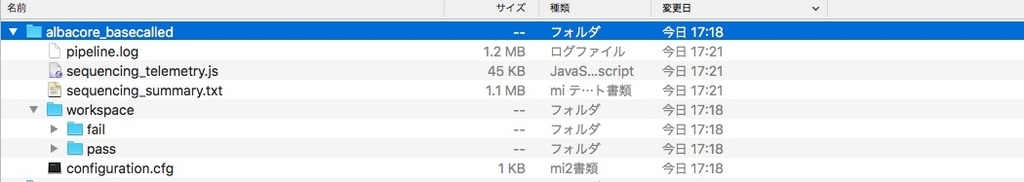
追記
@kazumachack いつもツールの紹介記事をありがたく拝見しております。
— あかまる (@aka_maru_k) March 12, 2019
ご存知だったら申し訳ないですが、
AlbacoreとGuppyについて、「--recursive」をつけることでサブフォルダ内のfast5をすべて検索してベースコールできます。全部のfast5を一つのフォルダにまとめなくても大丈夫です。
サブディレクトリも含めてbasecallingする。
read_fast5_basecaller.py -f FLO-MIN106 -k SQK-LSK308 -t 24 \
-s /data/albacore_basecalled/ -o fastq -q 100000 \
-i /data/Ecoli_R7_ONI_flowcell_18/
--recursive
- -r, --recursive Recurse through subfolders for input data files
あかまるさん、教えていただきありがとうございます。
引用
nanoporetech Albacore 2.3.3 Release
https://community.nanoporetech.com/posts/albacore-2-3-3
参考
https://github.com/rrwick/Basecalling-comparison/tree/95bf07476f61cda79e6971f20f48c6ac83e634b3
基本的なSQK-RAD004などが入ってませんね。MinKNOW(リンク)を使ってということでしょうか。












