2018 10/9 誤字修正
2018 10/22 CyVerseチュートリアル追記
2018 12/09 mapping追記
2018 12/12 前処理リンク追加
2019 10/21 リンク追加
植物のRNA seqを初めてされる方向けに作成した資料です。
真似すれば流れを再現できるように記載しています。興味があれば同じように実行してみてください。
論文
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3566969/
シーケンスデータ
https://www.ncbi.nlm.nih.gov/Traces/study/?acc=SRP010481
シロイヌナズナのfastaファイルとgtfファイル
http://plants.ensembl.org/info/website/ftp/index.html
追記;コマンドにエラーがあったので修正。Rの流れも一部修正。(8/9)
追記
0、前処理には、こちらのツールが使えます。
1、index
bowtie2-build -f A_thaliana_TAIR10.fa chr
2、mapping
tophat2 -p 4 -I 50000 -i 20 -o tophat_WT1-1 -G Arabidopsis_thaliana.TAIR10.36.gtf chr WT_15flower_receptacles1-1/input.fastq
- -p num-threads
- -I --max-intron-length (default: 500000)
- -i --min-intron-length (default: 50)
- -G --GTF
- -o --output-dir (default: ./tophat_out)
以下はアライメントのsensitivityに影響するパラメータ。
- --read-gap-length default: 2
- --read-edit-dist default: 2
- --read-mismatches default: 2
- --read-realign-edit-dist default: "read-edit-dist" + 1
- --min-anchor default: 8
- --read-mismatches default: 2
ここではtophatでなくtophat2を使ってますが、HISAT2(紹介)への切り替えも考えて下さい。
2021 追記
マッピングからリードカウントまでの流れはRSEMに切り替えることも検討してください。bowtie/bowtie2/star/hisat2のいずれかをアライナーとして使い、その後確率的リードカウントを行うパッケージです。出力は遺伝子レベルの発現行列ファイル、または転写産物レベルの発現行列ファイルになります。マッピングからリードカウントまでRSEMだけで一本化できます(アライナーのバイナリは必要)。マルチマッピングがない植物ゲノムはないと思うので、まず間違いなくカウント精度も上がるはずです(ランタイムは長くなります)。
3、rename
mv tophat_WT1-1/accepted_hits.bam tophat_WT1-1/WT1-1.bam
4、index
samtools index tophat_WT1-1/WT1-1.bam
step1-4を全てのサンプルに対して実行(またはスクリプトで処理)。
5、IGVでアライメントを確認
igv -g A_thaliana_TAIR10.fa
Arabidopsis_thaliana.TAIR10.36.gtf,WT1-1/WT1-1.bam,WT1-2/WT1-2.bam,WT1-3/WT1-3.bam,MT1-1/MT1-1.bam,MT1-2/MT1-2.bam,MT1-3/MT1-3.bam
6、featurecountsでカウント。
featureCounts -T 8 -t exon -g gene_id -a Arabidopsis_thaliana.TAIR10.36.gtf \
-o counts.txt \
WT1-1/WT1-1.bam WT1-2/WT1-2.bam WT1-3/WT1-3.bam \
WT2-1/WT2-1.bam WT2-2/WT2-2.bam WT2-3/WT2-3.bam \
WT3-1/WT3-1.bam WT3-2/WT3-2.bam WT3-3/WT3-3.bam \
MT1-1/MT1-1.bam MT1-2/MT1-2.bam MT1-3/MT1-3.bam \
MT2-1/MT2-1.bam MT2-2/MT2-2.bam MT2-3/MT2-3.bam \
MT3-1/MT3-1.bam MT3-2/MT3-2.bam MT3-3/MT3-3.bam
cut -f 1,7-12 counts.txt > count_selected.txt #いらない情報を削る。
#先頭行も不要なら削る
sed -e '1d' count_selected.txt > count_filtered.txt
#今回は問題ないが、gene idに不要な文字があれば削る。"transcript:"を削るなら
sed -e "s/transcript://" count_selected.txt > count_filtered.txt
7、データの分析(R)
library("ggplot2")
library("reshape2")
library("reshape")
rawCount = read.table("count_selected.txt", header = TRUE, row.names = 1)
head(rawCount) #必ず毎回確認
colnames(rawCount) <- c("WT1_1","WT1_2","WT1_3","WT2_1","WT2_2","WT2_3","WT3_1","WT3_2","WT3_3","MT1_1","MT1_2","MT1_3","MT2_1","MT2_2","MT2_3","MT3_1","MT3_2","MT3_3") #rename
head(rawCount) #必ず毎回確認
artificialCount = log2(rawCount + 1)
head(artificialCount) #必ず毎回確認
ggplot(artificialCount, aes(x = WT1_1)) +
ylab(expression(log[2](count + 1))) +
geom_histogram(colour = "white", fill = "#525252", binwidth = 0.4)
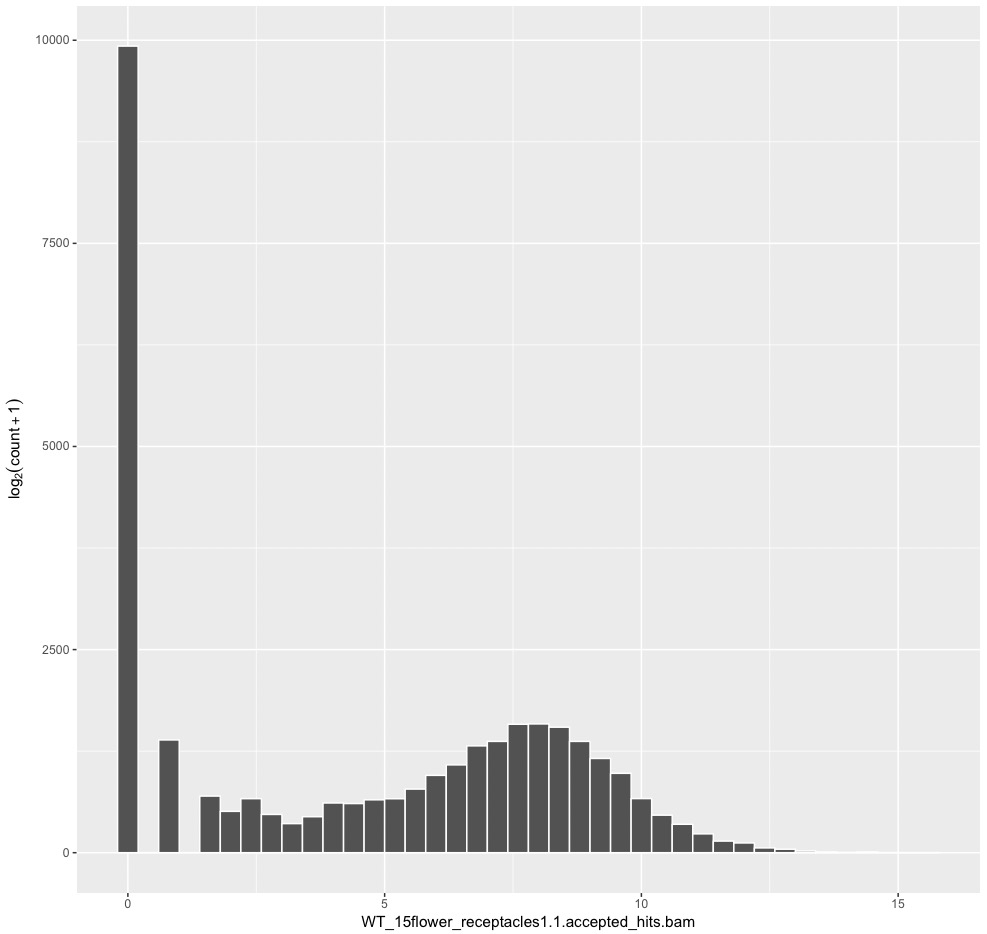
#WT1_1のraw count dataのヒストグラム。
df <- melt(artificialCount) #data.frame形式に変換。
head(df) #必ず毎回確認
g <- ggplot(df, aes (x = variable, y = value )) + geom_boxplot()
plot(g)
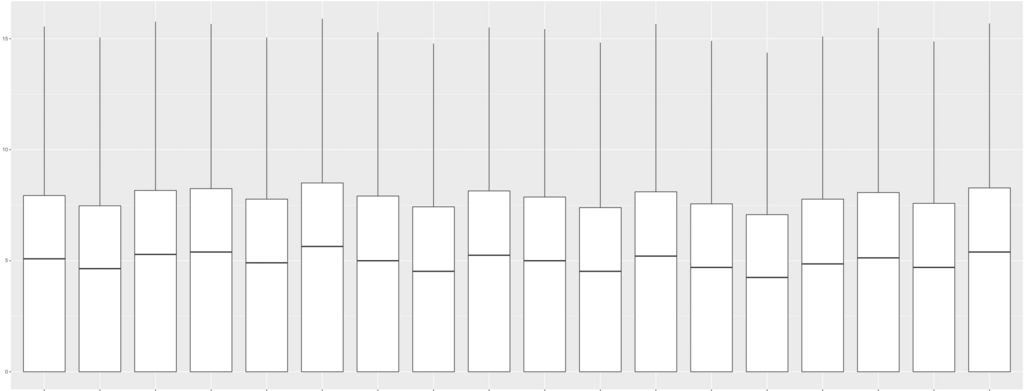
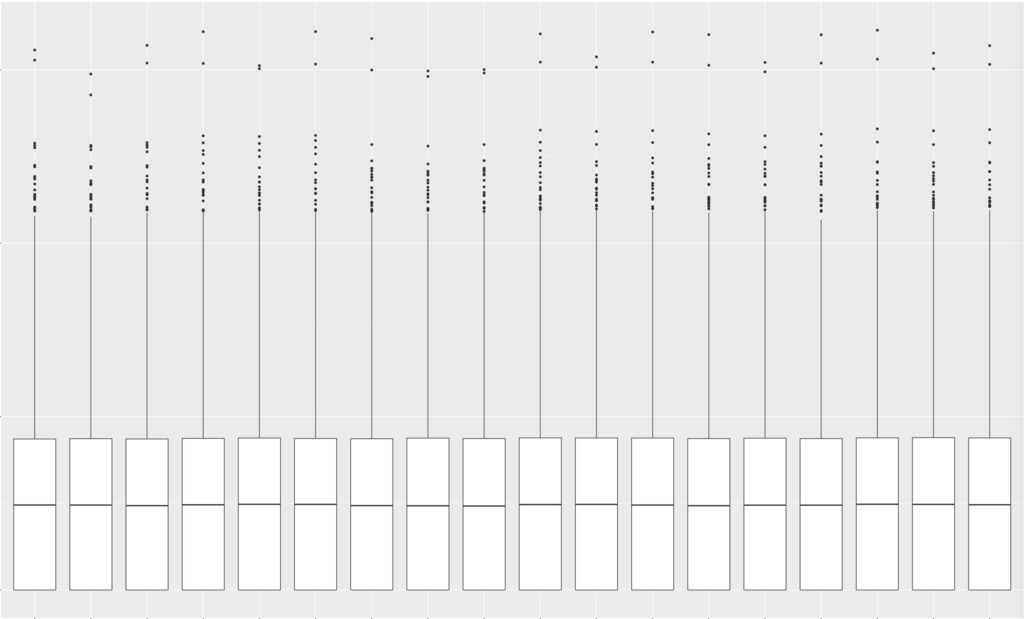
18サンプルのraw count dataのbox plot。 縦軸はlog2。
8、edgeRで正規化、DEG検出(R)
次にedgeRでTMM正規化してbox plotを書く。
library("edgeR")
count <- as.matrix(rawCount)
dim(count)
group <- factor(c("WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT", "WT","MT", "MT", "MT", "MT", "MT", "MT", "MT", "MT", "MT"))
d <- DGEList(counts = count, group = group)
d <- calcNormFactors(d) #全遺伝子commonの分散を計算
d <- estimateCommonDisp(d) #moderated tagwise dispersionの計算)
d <- estimateTagwiseDisp(d) #exact test
result <- exactTest(d)
topTags(result)
table <- as.data.frame(topTags(result, n = nrow(count))) #toptagsで全行指定してデータフレームに変換
is.DEG <- as.logical(table$FDR < 0.05) #logical クラス(true、false)に判定付きで変換。
DEG.names <- rownames(table)[is.DEG] #trueだけDEG.namesに収納
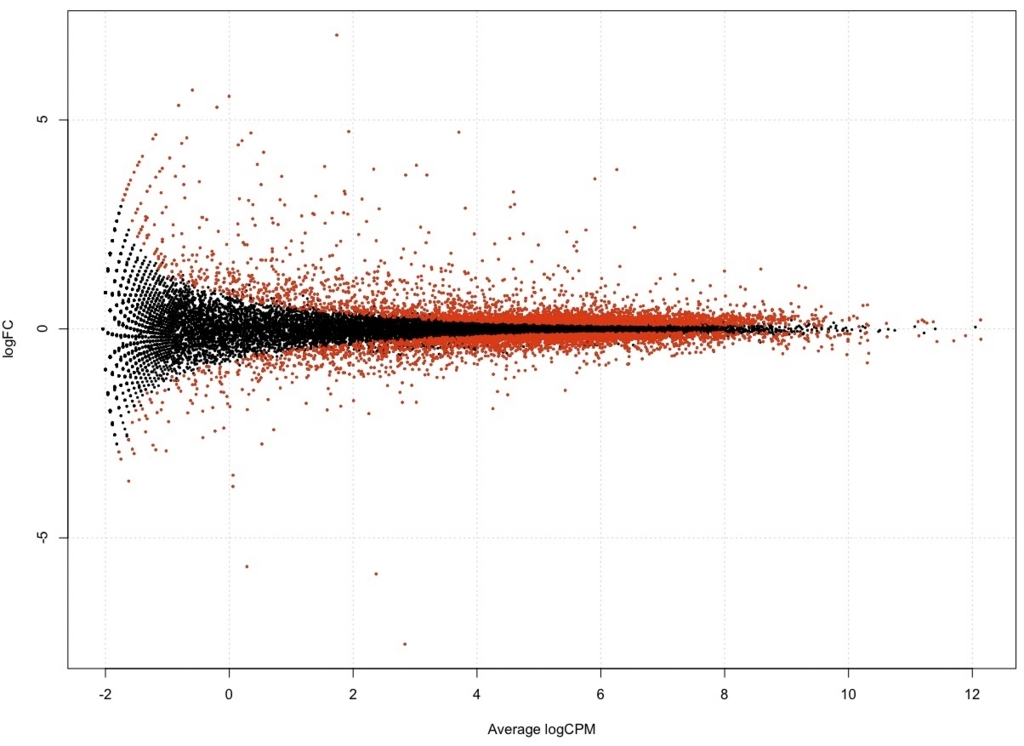
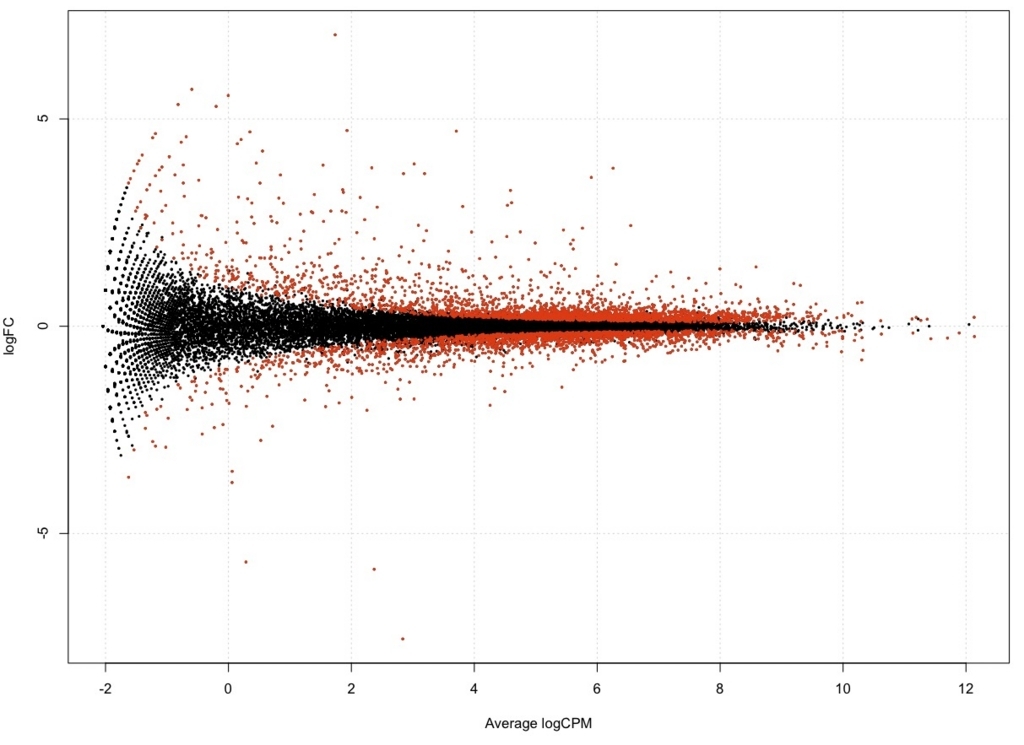
plotSmear(result, de.tags = DEG.names, cex=0.3)
FDR < 0.05
FDR < 0.01
正規化係数とライブラリサイズの確認
正規化係数
> d$samples$norm.factors
[1] 0.9888439 0.9993429 0.9877061 0.9953806 1.0011457 0.9931222 0.9891827
[8] 0.9974448 0.9891228 0.9994781 1.0106604 1.0006687 1.0045275 1.0151970
[15] 0.9991256 1.0073002 1.0186367 1.0037970
ライブラリサイズ
> d$samples$lib.size
[1] 8621707 6139876 10119340 10551274 7571024 12623646 8430112 5948333
[9] 9897606 8022571 5732382 9460971 6524094 4626588 7563974 9157905
[17] 6514360 10694322
TMM正規化。
calidata <- rawCount
head(calidata) #確認
calidata <- transform(calidata, WT1_1=(rawCount$WT1_1 / (d$samples$norm.factors[1] * d$samples$lib.size[1]) * 10^6)) #WT1_1 TMM calibration
calidata <- transform(calidata, WT1_2=(rawCount$WT1_2 / (d$samples$norm.factors[2] * d$samples$lib.size[2]) * 10^6)) #WT1_2 TMM calibration
calidata <- transform(calidata, WT1_3=(rawCount$WT1_3 / (d$samples$norm.factors[3] * d$samples$lib.size[3]) * 10^6)) #WT1_3 TMM calibration
.
. #continue to MT3-3 省略しますが18サンプル全部に実行。
.
calidata <- transform(calidata, MT1_3=(rawCount$MT1_3 / (d$samples$norm.factors[6] * d$samples$lib.size[6]) * 10^6)) #MT3_3 TMM calibration
#rename
colnames(calidata) <- c("WT1_1cali","WT1_2cali","WT1_3cali","WT2_1cali","WT2_2cali","WT2_3cali","WT3_1cali","WT3_2cali","WT3_3cali","MT1_1cali","MT1_2cali","MT1-3cali","MT2_1cali","MT2_2cali","MT2-3cali","MT3_1cali","MT3_2cali","MT3-3cali")
head(calidata) #確認
改めてbox plotを描画。
artificialCount = log2(calidata + 1)
head(artificialCount) #毎回確認
df <- melt(artificialCount) #data.frame形式に変換。
head(df) #必ず毎回確認
g <- ggplot(df, aes (x = variable, y = value )) + geom_boxplot()
plot(g)
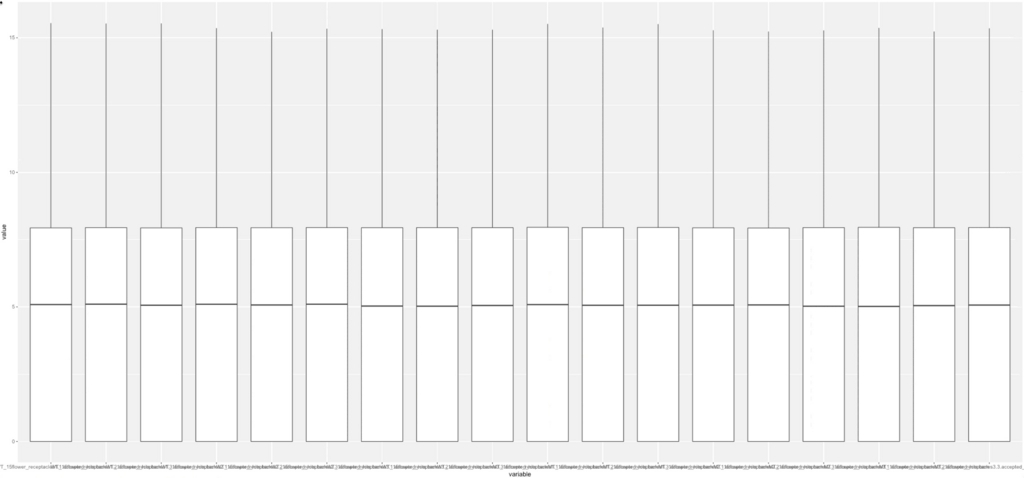
TMM補正するとboxの高さがほぼ揃った。 縦軸はlog2。
参考
FPKM補正
参考。 左からraw count、FPKM、TMM。

散布図 #参考サイト
plot(artificialCount[]) #全散布図 log2データ

小さすぎるので1/3だけキャプチャして載せています。
plot(artificialCount[,1],artificialCount[,9]) #WT1-1とMT1-1の散布図 log2データ

WT1_1とMT1_1の比較。
9、DEGの保存。(R)
edgeRのtableをresult.txtとして保存する(全遺伝子)。またFDRが0.01以下の遺伝子をsignificant.txtとして保存する。さらにsignificantのうち log Fold changeが0以上のtranscripts(fold change >1:誘導)と、 log Fold changeが0以下のtranscripts(fold change <1: 抑制)を取り出し保存する。
write.table(table, file = "result.txt", col.names = T, row.names = T, sep = " ", quote = F) #データを出力する。
#FDR<=0.01だけ取り出す
FDRThreshold <- table$FDR <= 0.01
significant <- table[FDRThreshold,]
head(significant) #確認
write.table(significant, file = "significant.txt", col.names = T, row.names = T, sep = ",", quote = F) #FDR<=0.01を保存
#誘導遺伝子を抽出
induction <- significant[(significant$logFC >= 0),] #誘導
head(induction) #確認
write.table(induction, file = "induction.txt", col.names = T, row.names = T, sep=",", quote = F) #保存
#抑制遺伝子を抽出
repression <- significant[(significant$logFC < 0),] #抑制
head(repression) #確認
write.table(repression, file = "repression.txt", col.names = T, row.names = T, sep = ",", quote = F) #保存
#Note
Modify the first row of induction.txt and repression.txt as shown below.
"logFC","logCPM","PValue","FDR"
↓
"name","logFC","logCPM","PValue","FDR"
#注意
induction.txtとrepression.txtを開いて、1行目にnameをつけ保存。
"logFC","logCPM","PValue","FDR"
↓
"name","logFC","logCPM","PValue","FDR"
10、クラスタリング(R)
クラスタリング
count <- as.matrix(calidata)
rho <- cor(count, method = "spearman")
d <- dist(1 - rho)
h <- hclust(d, method = "ward.D2")
plot(h)

WTとMTで仕分けられた。また容器ごとに仕分けられている。
11、ヒートマップ(R)
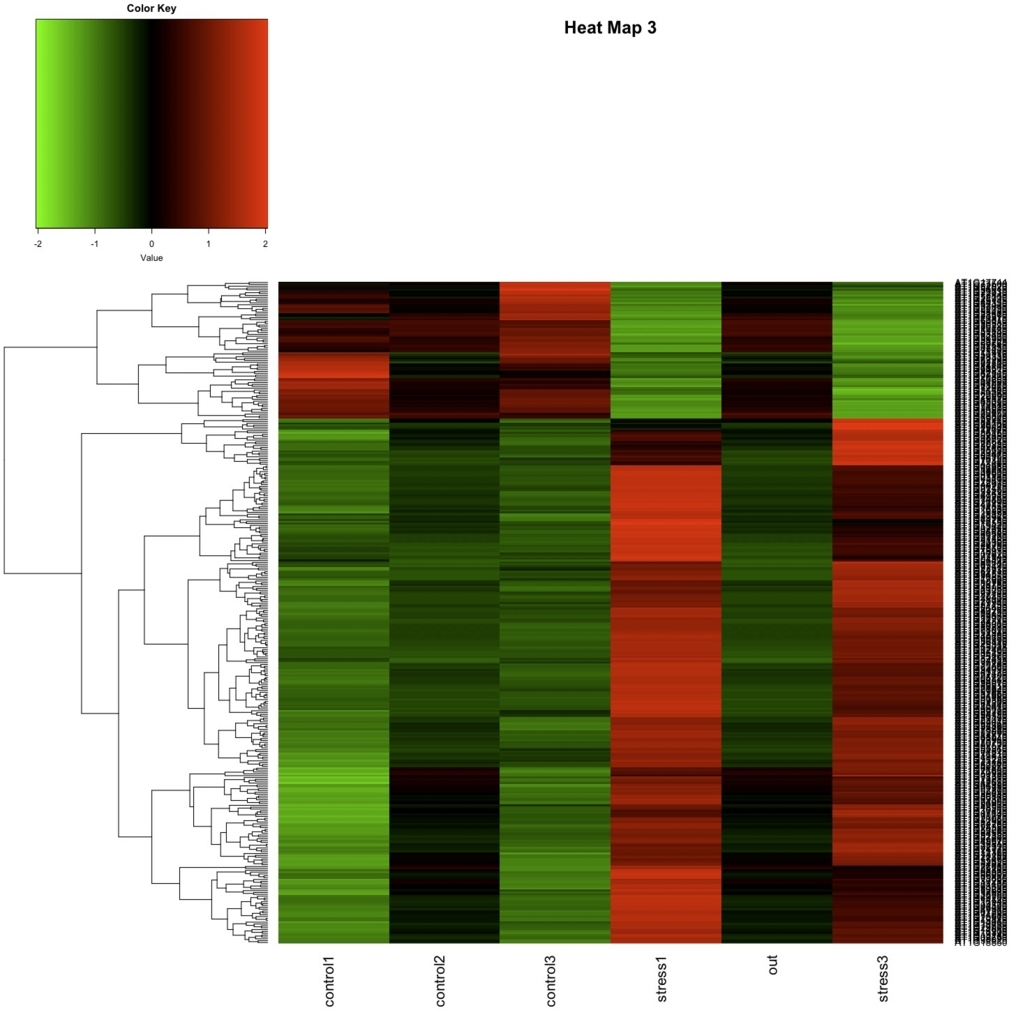
FDR < 0.01のtrranscripts (significant) を選抜し、heat-mapを描く。
data = read.table("result_FDR0.01.txt", header = TRUE, row.names = 1)
colnames(data) <- c("WT1_1","WT1_2","WT1_3","WT2_1","WT2_2","WT2_3","WT3_1","WT3_2","WT3_3","MT1_1","MT1_2","MT1_3","MT2_1","MT2_2","MT2_3","MT3_1","MT3_2","MT3_3")
#確認
head(data, n = 3)
> head(data, n = 3) #出力を載せておく。
WT1_1 WT1_2 WT1_3 WT2_1 WT2_2 WT2_3 WT3_1
AT1G01070 238 176.4618 257.6013 224.8571 212.5842 238.6935 233.1020
AT1G01080 421 426.5650 431.6101 362.8561 411.6709 399.8626 397.7047
AT1G01100 2438 2324.5710 2383.2383 2406.8644 2439.6563 2410.7365 2587.6366
WT3_2 WT3_3 MT1_1 MT1_2 MT1_3 MT2_1 MT2_2
AT1G01070 219.8509 243.8365 271.128 266.3540 299.8743 265.3806 246.8589
AT1G01080 379.3506 419.7471 340.239 338.4609 353.0051 357.7434 323.0947
AT1G01100 2362.3197 2598.6001 2205.174 2051.3672 2270.2193 2258.3366 2116.4519
MT2_3 MT3_1 MT3_2 MT3_3
AT1G01070 258.3369 241.2162 268.5197 242.2265
AT1G01080 337.3045 395.5576 396.9981 373.2671
AT1G01100 2229.1425 2294.7885 2128.8861 2354.7594
heatmap(as.matrix(data), margin=c(20,4), main="Heat Map 1 (Raw Data)")

library(genefilter) #リンク
data.z <- genescale(data, axis=1, method="Z")
heatmap(as.matrix(data.z), margin=c(20,4), main="Heat Map 2")

library(gplots) #リンク
heatmap.2(as.matrix(data.z), col=greenred(75), scale="none", key=TRUE, symkey=FALSE, density.info="none", trace="none", cexRow=1, margin=c(6,8), main="Heat Map 3", Colv=NA)

12、 主成分分析(R)
count <- as.matrix(count)
count.log <- log10(count + 1)
count.log.var <- apply(count.log, 1, var)
count.log.pca <- count.log[order(count.log.var, decreasing = TRUE), ][1:200, ]
res <- prcomp(count.log.pca)
col <- c("red", "red", "red", "red", "red", "red", "red", "red", "red", "blue", "blue", "blue", "blue", "blue", "blue", "blue", "blue", "blue")
time <- c("1", "1", "1", "2", "2", "2", "3", "3", "3", "1", "1", "1", "2", "2", "2", "3", "3", "3")
pc1 <- res$rotation[, 1]
pc2 <- res$rotation[, 2]
pc3 <- res$rotation[, 3]
plot(pc1, pc2, type = "n", asp = TRUE)
text(pc1, pc2, labels = time, col = col)
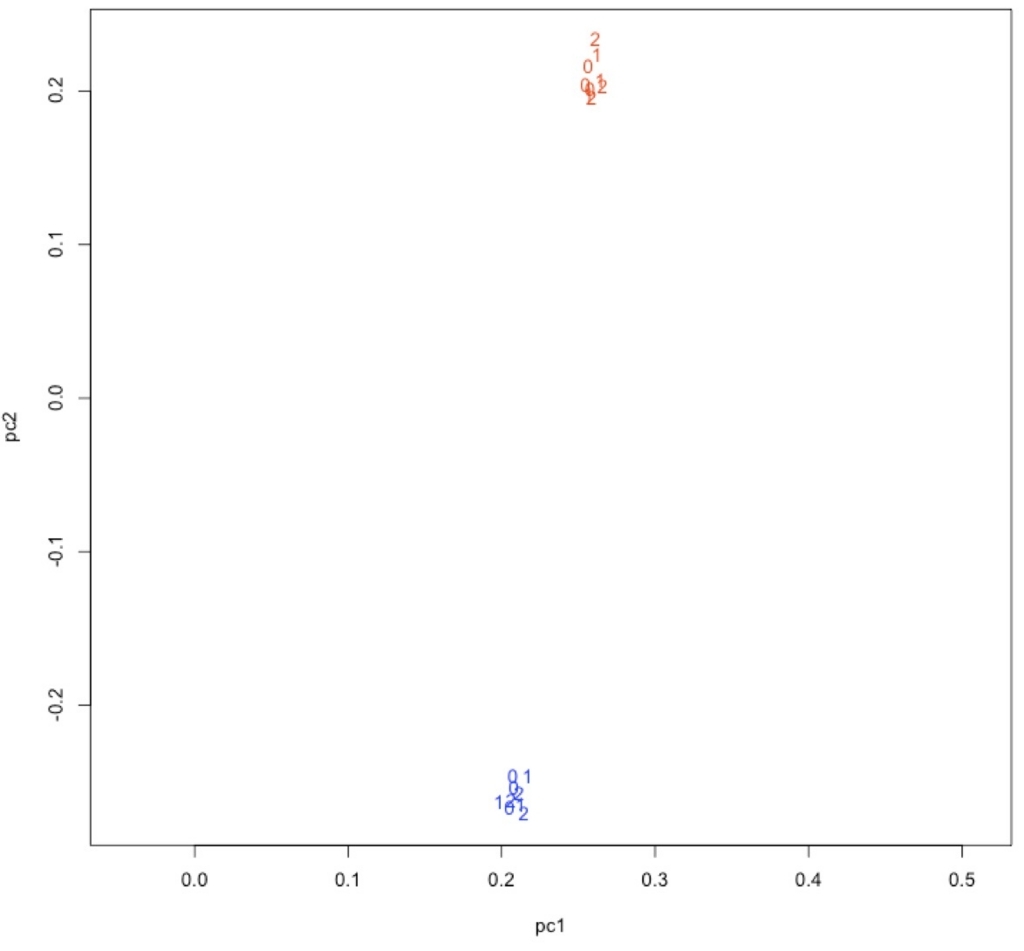
青がWT、赤がMT。
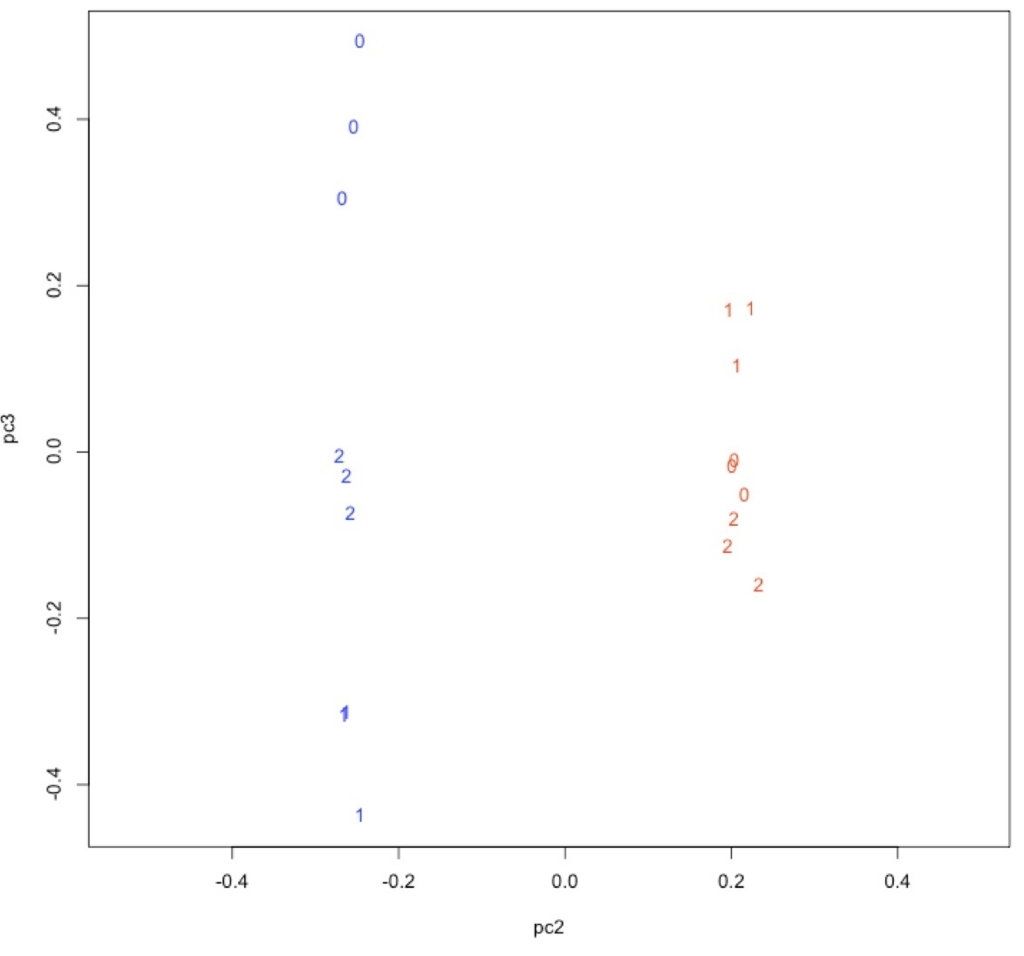
plot(pc2, pc3, type = "n", asp = TRUE)
text(pc2, pc3, labels = time, col = col)
induction.txt、repression.txt、significant.txtの1行目を"logFC","logCPM","PValue","FDR" から "name","logFC","logCPM","PValue","FDR"に変更。
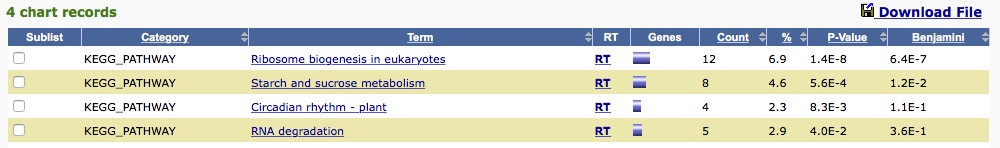
13、GOエンリッチメント解析
library(org.At.tair.db) #リンク
library(goProfiles) #リンク
x <- read.csv("induction.txt", header=TRUE, sep=",")#上の方でlogFC>=0条件で抽出したデータを読み込む。
head(x, n=3)
# name logFC logCPM PValue FDR
# 1 AT5G03820 3.811659 6.257744 0.00e+00 0.00e+00
# 2 AT2G02010 3.590547 5.904101 0.00e+00 0.00e+00
# 3 AT1G71380 2.365841 5.759736 0.00e+00 0.00e+00
induction <- basicProfile(genelist=x$name, onto='ANY', level=2, orgPackage="org.At.tair.db") #x$nameが遺伝子リスト
basicProfile(リンク)。
- genelist List of genes on which the Profile has to be based
- onto Ontology on which the profile has to be built( ’ANY’, ’MF’,’BP’,’CC’).
- Level Level of the ontology at which the profile has to be built
- orgPackage Name of a Bioconductor's organism annotations package ('org.Xx-eg-db').
plotProfiles(induction) #plotProfilesリンク
出力が以下になる。

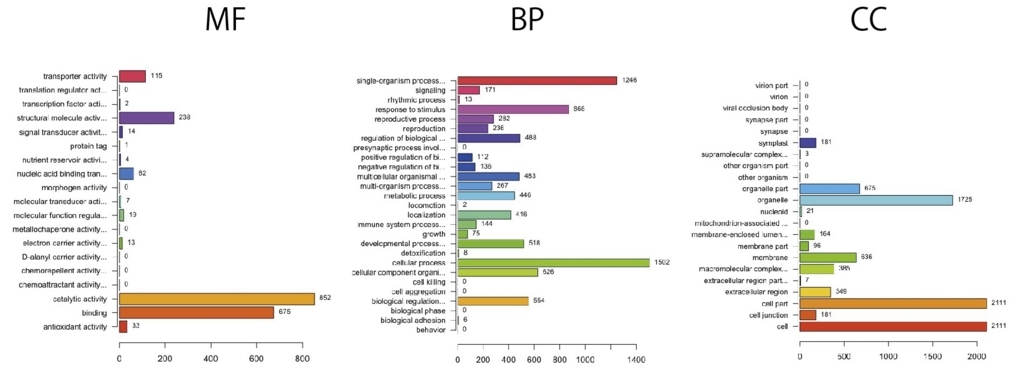
ANYだと一部のカテゴリが削られている。 ’MF’,’BP’,’CC’に分けた方が良い。下はANYと同じパラメータで ’MF’,’BP’,’CC’を指定してグラフを書いている。
抑制
y <- read.csv("repression.txt", header=TRUE)#上の方でlogFC<0条件で抽出したデータを読み込む。
head(y, n=3)
# name logFC logCPM PValue FDR
# 1 AT1G75945 -7.543735 2.836073 1.53e-272 8.35e-269
# 2 AT3G23460 -1.906906 4.256721 4.08e-145 8.37e-142
# 3 AT4G05715 -5.862613 2.371176 2.38e-97 2.37e-94
repression <- basicProfile(genelist=y$name, onto='MF', level=2, orgPackage="org.At.tair.db")
plotProfiles(repression)
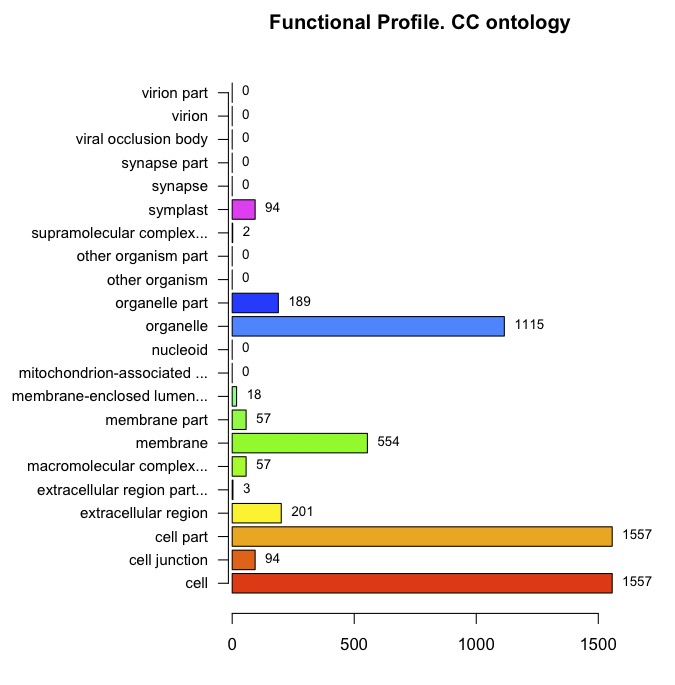
誘導、抑制を同時に書く。MFを書いてみる。
induction <- basicProfile(genelist=x$name, onto='MF', level=2, orgPackage="org.At.tair.db") #MF、level2
repression <- basicProfile(genelist=y$name, onto='MF', level=2, orgPackage="org.At.tair.db") #MF、level2
result <- mergeProfilesLists(induction, repression, profNames = c("induction", "repression"))
plotProfiles(result, legendText = c("induction", "repression"), percentage = TRUE)

保存
write.table(result, file = "GO_profile.txt")
テキストファイルが保存される。
| MF.Description "MF.GOID" "MF.induction" "MF.repression" |
| 13 "antioxidant activity" "GO:0016209" 33 12 |
| 9 "binding" "GO:0005488" 674 492 |
| 4 "catalytic activity" "GO:0003824" 852 609 |
| 1 "electron carrier activity" "GO:0009055" 13 4 |
| 14 "metallochaperone activity" "GO:0016530" 0 1 |
| 20 "molecular function regulator" "GO:0098772" 18 18 |
| 19 "molecular transducer activity" "GO:0060089" 7 18 |
| 3 "nucleic acid binding transcription factor activity" "GO:0001071" 62 96 |
| 6 "nutrient reservoir activity" "GO:0045735" 4 5 |
| 10 "protein tag" "GO:0031386" 1 0 |
| 5 "signal transducer activity" "GO:0004871" 14 25 |
| 7 "structural molecule activity" "GO:0005198" 238 10 |
| 2 "transcription factor activity, protein binding" "GO:0000988" 2 0 |
| 8 "transporter activity" "GO:0005215" 115 171 |
数が多い少ないでは意味が薄い。割合を出し検定を行い、エンリッチメントされた可能性がある系を絞り込む必要がある。
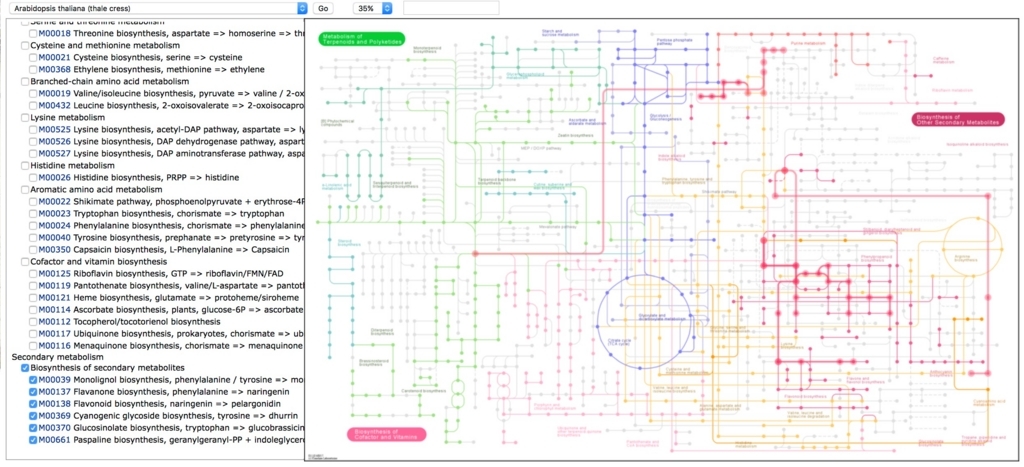
pathway
#先ほど読み込んだx(誘導された遺伝子を示したdata.frame)を使う。
library(pathview) #(pathviewインストール情報リンク)
path.id <- "00010" #解糖系
path <- pathview(gene.data = as.matrix(x$name), pathway.id = path.id, species = "ath", gene.idtype = "KEGG")
- gene.data either vector (single sample) or a matrix-like data (multiple sample).
- pathway.id character vector, the KEGG pathway ID(s), usually 5 digit, may also include the 3 letter KEGG species code.
- species character, either the kegg code, scientific name or the common name of the tar- get species.
- gene.idtype character, ID type used for the gene.data, case insensitive. Default gene.idtype="entrez", i.e. Entrez Gene, which are the primary KEGG gene ID for many common model organisms. For other species, gene.idtype should be set to "KEGG" as KEGG use other types of gene IDs.
pathwayのpngがワーキングディレクトリに保存される。
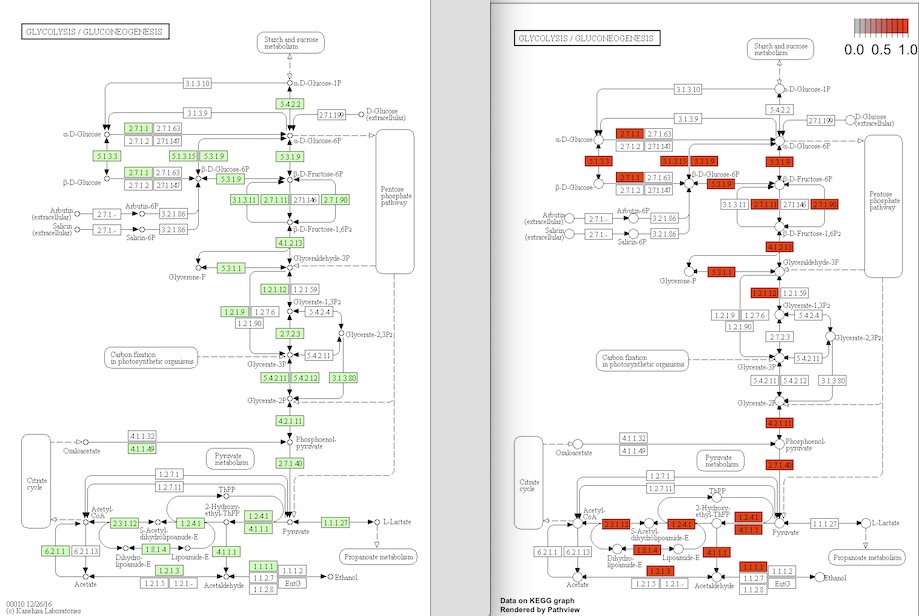
解糖系のpathway。左はアラビでアノテーションされている遺伝子(緑)の図。右が誘導されていた遺伝子(赤)の図。
最初は全てのpathwayを見た方が良いかもしれない。しかし全部調べるのは面倒臭い。それならpathwayのIDを一括取得して、ループで回した方が便利なので、pathwayのIDを取得して全pathwayを描画する流れを書いておく。
1、Rを中断(crt + z)、または新しいターミナルのタブを開く。
2、まずkeggに登録された生物名をこのリンクから確認する。Arabidopsis属だと2つ登録されている。
T00041 ath Arabidopsis thaliana (thale cress) Eukaryotes;Plants;Eudicots;Mustard family
T01578 aly Arabidopsis lyrata (lyrate rockcress) Eukaryotes;Plants;Eudicots;Mustard family
2、A.thaliana とA.lyrataが見つかるが、ここではA.thalianaを使う。左端にathと略号が書かれていることに注目。これをURL(http://rest.kegg.jp/list/pathway/)につけて、http://rest.kegg.jp/list/pathway/ath と検索する。
3、A.thalianaのpathway一覧が表示されている。

#参考 ちなみにhttp://rest.kegg.jp/list/athだと遺伝子リスト。
4、このページ全体をコピペするか、wgetを叩きダウンロードする。wgetなら下のように打つ。
wget http://rest.kegg.jp/list/pathway/ath -O - |sed -e 's/path://g' > pathway_list
sedで先頭のpath:ath00010のpath:を消しながらダウンロードしている。
5、A.thalianaのkegg pathway IDのリストが入手できた。
user$ head -3 pathway_list
ath00010 Glycolysis / Gluconeogenesis - Arabidopsis thaliana (thale cress)
ath00020 Citrate cycle (TCA cycle) - Arabidopsis thaliana (thale cress)
ath00030 Pentose phosphate pathway - Arabidopsis thaliana (thale cress)
入手したIDリストを使い、全pathwayを描く。
cut -f 1 pathway_list > pathway_ID
ちなみにRの中からunixのコマンドを呼び出して実行するなら
system("cut -f 1 pathway_list > pathway_ID")
Rに戻る(一時停止させているならfg %job番号で再開)。
IDlist <- read.table("pathway_ID", header=FALSE) #pathway IDを読み込み
path <- pathview(gene.data = as.matrix(x$name), pathway.id = unlist(IDlist), species = "ath", gene.idtype = "KEGG")
IDがデータフレームのままだと認識してくれないので、unlist(IDlist)
でベクトルに変換している。使った全てのpathwayのグラフが描画される。アラビだと10分くらいかかる。
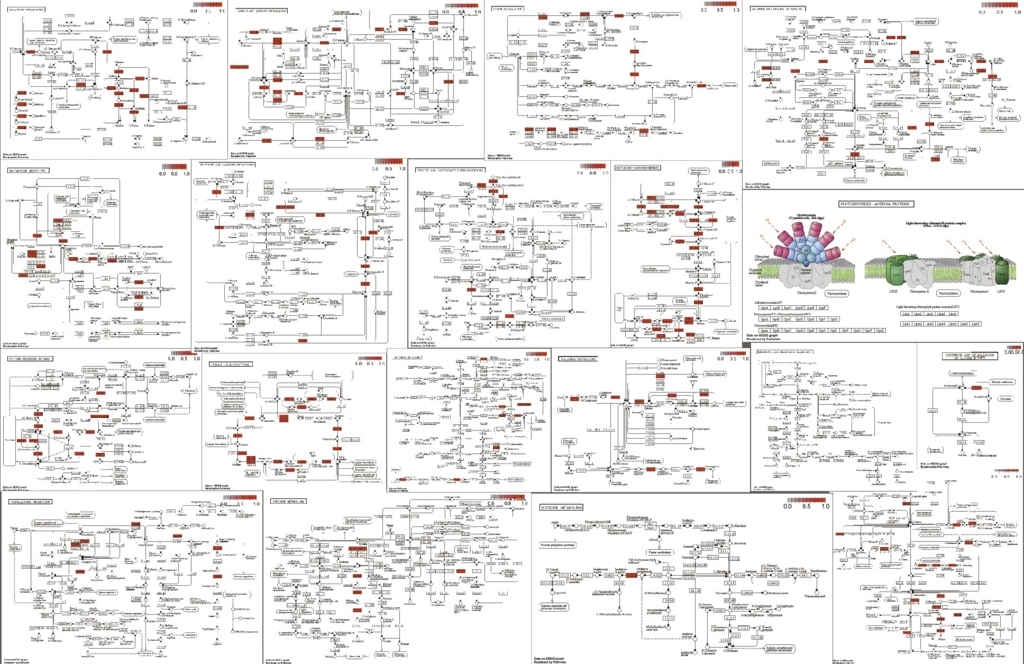
全部は載せられないが、このように一括してpathwayを取得してマップを描画できる。もちろん、エンリッチされたpathwayがGO解析で分かっていれば、それだけ書いてもいい。
誘導、抑制をpathwayで表示できた方が便利そうなので、少し改良する。
significant.txtを開いて、1行目にnameをつけsignificant2.txtとして保存。
"logFC","logCPM","PValue","FDR"
↓
"name","logFC","logCPM","PValue","FDR"
significant2.txtをRに読み込む。
significant <- read.table("significant2.txt", header=TRUE, sep=",")
head(significant, n=3) #確認
name logFC logCPM PValue FDR
1 AT5G03820 3.811659 6.257744 0.000000e+00 0.000000e+00
2 AT2G02010 3.590547 5.904101 0.000000e+00 0.000000e+00
3 AT1G71380 2.365841 5.759736 0.000000e+00 0.000000e+00
genelist <- as.vector(significant$logFC)
names(genelist) <- as.vector(significant$name)
path2 <- pathview(gene.data = genelist, pathway.id = unlist(IDlist), species = "ath", gene.idtype = "KEGG")
誘導、抑制が色でわかるようになった。

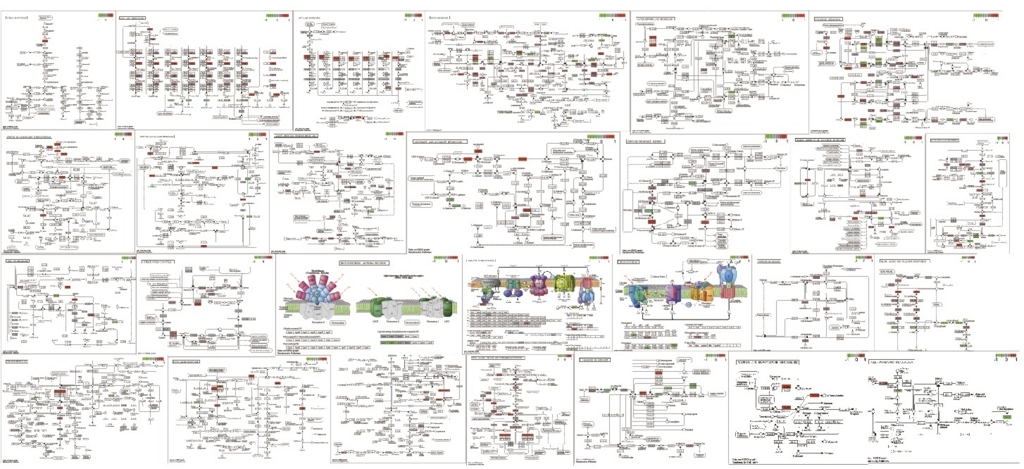
全部では無いが、その他の系。
resulttxtを開いて、1行目にnameをつけ保存。
"logFC","logCPM","PValue","FDR"
↓
"name","logFC","logCPM","PValue","FDR"
library("clusterProfiler") #リンク
library(org.At.tair.db)
all = read.table("result.txt", header = TRUE) #全遺伝子(バックグラウンドとして使う)
head(all, n=3)
name logFC logCPM PValue FDR
1 AT5G03820 -3.967569 6.623068 0 0
2 AT2G02010 -3.770837 6.244671 0 0
3 AT4G23690 -2.719579 6.855122 0 0
allgene <- bitr(all$name, fromType="TAIR", toType="ENTREZID", OrgDb="org.At.tair.db") #比較できるようにIDをTAIRからENTREZIDに変換。
head(allgene)
x <- read.csv("induction.txt", header=TRUE)#上の方でlogFC>=0条件で抽出したデータを読み込む。
head(x)
y <- read.csv("repression.txt", header=TRUE)#上の方でlogFC<0条件で抽出したデータを読み込む。
head(y, n=3)
induction <- bitr(x$name, fromType="TAIR", toType="ENTREZID", OrgDb="org.At.tair.db") #コマンドbitrで誘導TAIR IDをENTREZIDへID変換する。
head(induction)
repression <- bitr(y$name, fromType="TAIR", toType="ENTREZID", OrgDb="org.At.tair.db") #コマンドbitrで抑制TAIR IDをENTREZIDへID変換する。
head(repression)
egoBP <- enrichGO(gene = induction[,2],
universe = allgene[,2], #allgeneと比較
OrgDb = org.At.tair.db,
ont = "BP",
pAdjustMethod = "BH",
pvalueCutoff = 0.01,
qvalueCutoff = 0.05,
readable = TRUE)
write.table(egoBP[,-8], "GO_BP_over-representation.txt", quote=F, col.names=TRUE, sep ="\t") #保存
#CC、MFについても同様に実行する。また抑制についても行う。
このようになった。
#誘導 最初の5行 (合計294)
| |
ID |
Description |
GeneRatio |
BgRatio |
pvalue |
p.adjust |
qvalue |
Count |
| GO:0001510 |
GO:0001510 |
RNA methylation |
141/2099 |
172/20469 |
2.76E-109 |
4.17E-106 |
2.90E-106 |
141 |
| GO:0042254 |
GO:0042254 |
ribosome biogenesis |
179/2099 |
338/20469 |
1.51E-87 |
1.14E-84 |
7.91E-85 |
179 |
| GO:0022613 |
GO:0022613 |
ribonucleoprotein complex biogenesis |
179/2099 |
344/20469 |
7.14E-86 |
3.59E-83 |
2.50E-83 |
179 |
| GO:0009451 |
GO:0009451 |
RNA modification |
169/2099 |
325/20469 |
5.55E-81 |
2.09E-78 |
1.46E-78 |
169 |
誘導された遺伝子についてGO絶対数のグラフと、全遺伝子に対して有意差検定したグラフの2つを描く。
ggoCC <- groupGO(gene = induction$ENTREZID, OrgDb = org.At.tair.db, ont = "CC", level = 3, readable = TRUE) #ほか2つも実行
barplot(ggoCC, drop=TRUE, showCategory=20, font=12) #検出されたGOの数のグラフ

#全IDと比較してp値を計算しバーの色で表現する(上で計算したegoBPを使う)
barplot(egoBP, showCategory=20, font=12)

dot plotでグラフ化することも可能。
dotplot(egoBP,showCategory=20, font=12)

plotGOgraph(egoCC)
enrichMap(ego, n=10)

Bioconductor のpathway解析ツール(117種類)
Bioconductor - BiocViews
Bioconductor のenrichment解析ツール(82種類)
Bioconductor - BiocViews
引用
TMM正規化
http://www.nathalievilla.org/doc/pdf/tutorial-rnaseq.pdf
edgeR
二群間比較(edgeR) | RNA-Seq による比較トランスクリプトーム解析
クラスタリング
階層クラスタリング | スピアマンの順位相関係数を利用したクラスタリング
主成分解析
主成分分析 | RNA-Seq リードデータをクラスタリング
GO profile
goProfiles | 遺伝子セットの GO term の出現頻度を調べ棒グラフで視覚化
org.At.tair.db | シロイヌナズナのアノテーション Bioconductor
pathview
pathview | KEGG パスウェイの視覚化パッケージ
ヒートマップ作成
どんな図を作成すべきかのナビゲート。
コマンドが難しければCyVerseを使うのも手です。
CYVERSE RNA seqチュートリアル
- RNA-seq Tutorial- STAR, StringTie and DESeq2
https://pods.iplantcollaborative.org/wiki/display/TUT/RNA-seq+Tutorial-+STAR%2C+StringTie+and+DESeq2
- RNA-Seq with Kallisto and Sleuth
https://cyverse-kallisto-tutorial.readthedocs-hosted.com/en/latest/
どれだけrepicatesやリード数は必要か?
ENCODEのガイドライン
https://www.encodeproject.org/about/experiment-guidelines/
=> RNA seq
ENCODE Experimental Guidelines for ENCODE3 RNA-seq
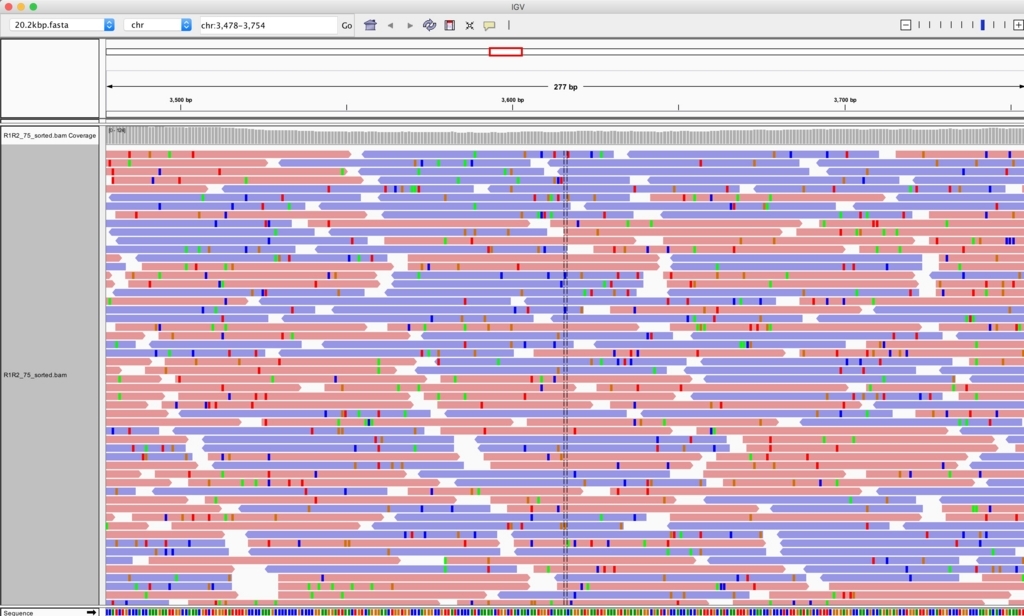









 矢印部分がwiuggleファイルで表記されている部位である。
矢印部分がwiuggleファイルで表記されている部位である。