2018 10/2 誤字修正
2018 11/4 追記
2019 6/2 リンク追加
2020 4/7 補足説明追加
2020 11/4 ツイート追加
2021 6/13 コマンド追加
バイオインフォマティクスは、新しいアルゴリズムの開発、複雑なパイプラインへの様々なツールの接続(Perez-Riverol et al、2014)、それらの蓄積と普及により、システム生物学の機能と振る舞いの理解への重要な貢献をしている。これらの開発は、単一のツールと個別ツールからOpenMS(Röstet al、2016)、Taverna(Wolstencroft et al、2013)、Galaxy(Afgan et al、2016)などの複雑かつ統合されたワークフローシステムに移行してきているが、ソフトウェア開発者やバイオインフォマティクスコミュニティにとっての主な2つの課題は、(i)ソフトウェアが利用可能なこと、(ii)再現性ある実験ができること、である。バイオインフォマティクスにおけるいくつかのアルゴリズムソフトウェアおよびパイプラインは、適切なinstallationおよびconfiguration(例えば、システム依存性のコンフリクト)のために多大な努力を必要とする。オリジナルの結果の複製可能性と再現性が保証される良い出発点というのは、十分に文書化(ソフトウェアパラメータ、依存性など)され、簡単にインストールできるものである。 Docker(docker.com)やrkt(https://coreos.com/rkt)などのコンテナベースのテクノロジーは、いわゆるソフトウェアコンテナ内でのアプリケーションの展開を自動化することで、これらの課題を克服している。ソフトウェアコンテナは、システムの他の部分に影響を与えることなく、特定のソフトウェアのインストールと実行のための独立した環境を提供する。異なる複数のグループが、バイオインフォマティクスの問題を解決するためにDockerコンテナを使用することを提案している(Belmann et al、2015; Moreews et al、2015)。しかし、これらのプロジェクトのほとんどは個々の努力にとどまっており、バイオインフォマティクスでのDocker技術の可能性はまだ探索段階にある。
ここでは、バイオインフォマティクスコンテナを作成、管理、配布するためのインフラストラクチャとガイドラインを提供するコミュニティ主導のプロジェクトであるBioContainers(biocontainers.pro)を紹介する。 BioContainersアーキテクチャは、バイオインフォマティクスコンテナの要求と保守、およびユーザとコミュニティとの相互作用を容易にする。貢献する人物が30人を超え、コミュニティ主導のアプローチはプロジェクトの持続可能性とスケーラビリティを保証している。さらに、BioContainersはBioConda(https://bioconda.github.io/)プロジェクトと統合され、各BioCondaレシピのコンテナの自動生成が可能になった。執筆の時点で、BioContainersは2076以上のコンテナを提供し、共通のウェブレジストリ(biocontainers.pro/registry/)を通じて検索、タグ付け、アクセスすることができる。最後に、BioContainerをGalaxy(https://galaxyproject.org/)やPhenoMeNal H2020(http://phenomenal-h2020.eu/home/)などのオープンソースプロジェクトとコンテナプロバイダとして統合する方法について説明する。
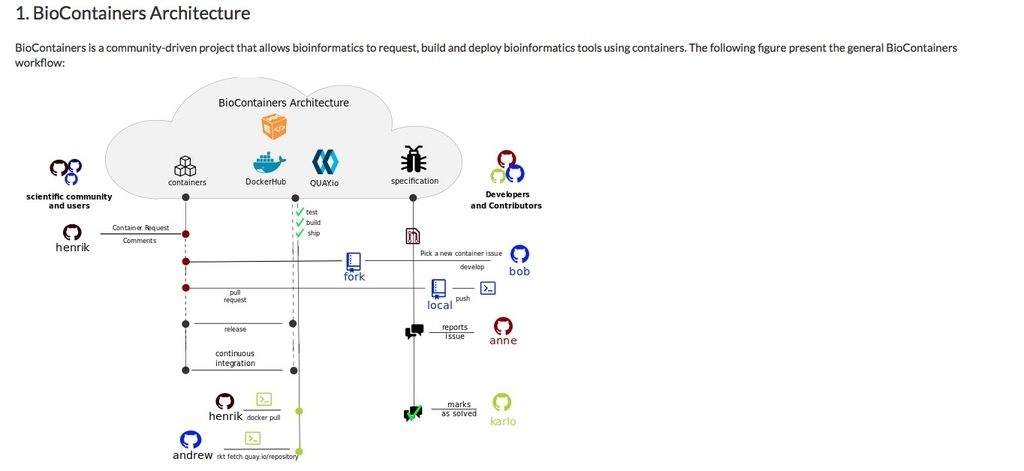
BioContainersアーキテクチャは主要な2つのコンポーネントからなる、(i)すべてのDockerfiles(Dockerfileベースのコンテナ用)、仕様、およびコンテナの作成/管理ツールを含むGitHub組織(github.com/BioContainers/)、 (ii)利用可能なコンテナが自動システムによって構築され、DockerまたはrktによってすぐにダウンロードできるようになっているBioContainersレジストリおよびレジストリUI(biocontainers.pro/registry/)(例http://biocontainers.pro/docs/101/running-example/)。 論文図1は、ユーザーレクエストからコンテナの最終展開までのBioContainersインフラストラクチャを示している。
BioContainerのユーザーは、ソフトウェア(名前、URL、またはパッケージ化するバイナリ)に関する情報を含む問題をコンテナのリポジトリに開くことによって、ソフトウェアコンテナを要求できる。 BioContainersコミュニティのメンバーが問題を選択し、特定のコンテナを生成する。 自動化されたビルドシステムが設定/配備され、新しいコンテナが数時間以内に利用可能になる。 両方のレジストリを統合するために、著者らはユーザが配置された場所から独立してBioContainerを検索、タグ付け、検索することを可能にするRegistry-UI(biocontainers.pro/registry/)を開発した。 ユーザーは、dockerまたはrktを使用して、対応するコンテナをpullまたはfetchできる。
We just received this email from @Docker "BioContainers has been added to our Docker Open Source program. @Biocontainers is now a whitelisted namespace". Thanks to @Docker for the support to the Bioinformatics community. Thanks to Olivier Sallou for leading the conversations 🏆
— BioContainers (@BioContainers) 2020年11月3日
Overview of the BioContainers architecture。webマニュアルより転載。
- マニュアル
- 追加するための規則
https://github.com/BioContainers/specs/blob/master/CONTRIBUTING.md
BioContainersに関するツイート
base image
The #Bioconductor team maintains base #docker images to enhance reproducibility. https://t.co/dVxhPjDp8i pic.twitter.com/3cLD0pWrMj
— Sean Davis (@seandavis12) 2020年3月12日
以下はDokcerに触れた経験がほぼ無い人向けに書いています。
使い方
step1、dockerのインストール
すでに優れた記事がたくさん書かれているので、ググってdockerをインストールし、起動しておいてください。Qiita(キータ)に上がっている最近の記事リンクを張っておきます(リンク)。
step2、準備
OSログイン時点でdockerが起動するように設定できるが、起動してなければ、OSX右上のランチャからdockerを起動する。Runを選択して10秒程度待つ。ランチャの設定から、CPUは最大12、メモリは最大16GB、とかなり高めのリソース制限にした。NGS関連のツールはワークステーションクラスのマシン性能を要求するツールも多いためです。環境に合わせて変えてください。

step3、イメージの入手: イメージをダウンロードするか、自分でイメージをビルドする。
BioContainersのHPのContainersをクリックするとDockerfiles(イメージビルドのための仕様書)が構築済みのツール一覧を確認できる。 ContainersをクリックするとBioContainersの管理するGIthubレポジトリにジャンプする。
最初にdockerイメージ共有サイトからBioContainersのツールをpullする手順を3パターン紹介し、最後にBioContainersのツールイメージを自分でビルドして使う方法を紹介する。
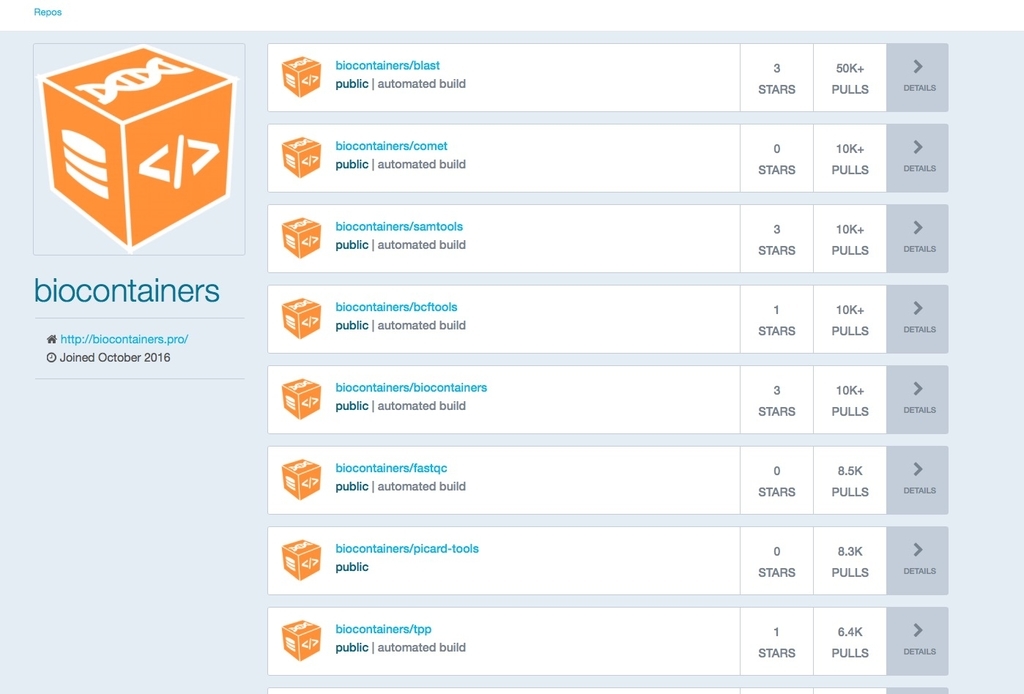
方法1、Docker hubのbiocontainersからイメージを探してpull
https://hub.docker.com/u/biocontainers/
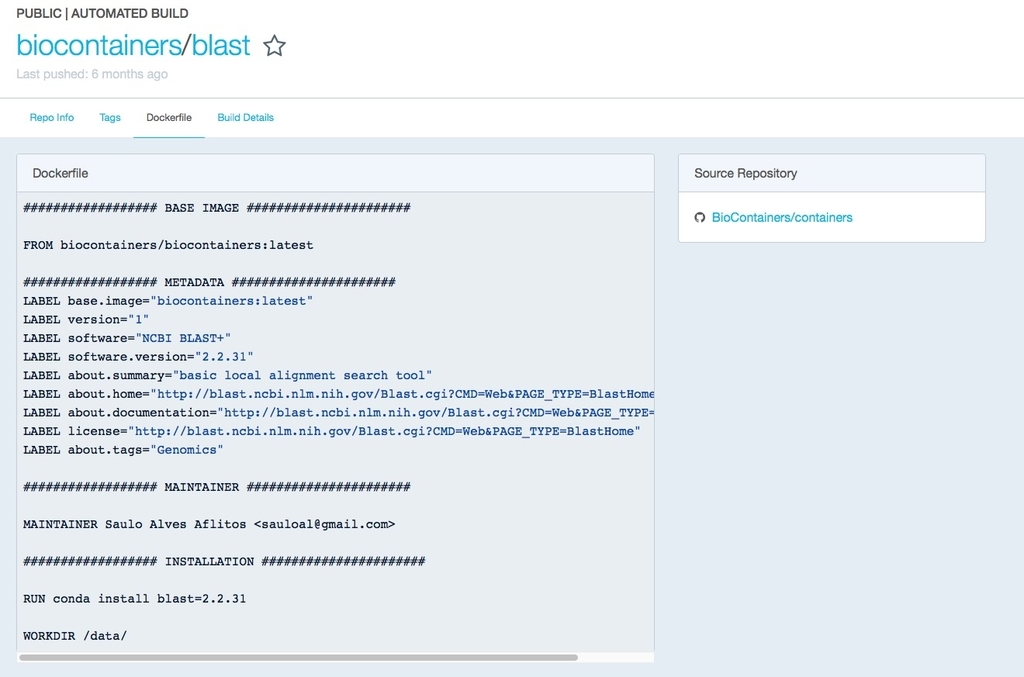
現状、バイオインフォマティクス研究者の努力により、1000を超えるツールにDockerfilesが準備されている。一番上のblastをクリックする。
BioContainersの各種リンク、Dokerfileの記載内容など確認できる。ここでは右のコード"docker pull biocontainers/blast"を端末にコピペする。
docker pull biocontainers/blast
$ docker pull biocontainers/blast
Using default tag: latest
22dc81ace0ea: Already exists
1a8b3c87dba3: Already exists
69fea7e39cc9: Downloading [============> ] 23.57MB/98MB
ダウンロードが終わるまで待つ。
ダウンロードが終わったらイメージ一覧を表示。
docker images #全イメージを確認
$ docker images
REPOSITORY TAG IMAGE ID CREATED SIZE
blast latest 06e20d67e8c5 Less than a second ago 2GB
biocontainers/blastが追加されている。イメージサイズは2GBだった。ubuntuイメージはGUIなしで100MB以下の容量しかないが、biocontainersには色々なものが導入されているため、重くなる(biocontainersのベースはubuntu16.04。biocontainersそのもののDockerfilesはこちら、またはGithubレポジトリのBioContainers/biocontainers/1.0.0/Dockerfiles link)。
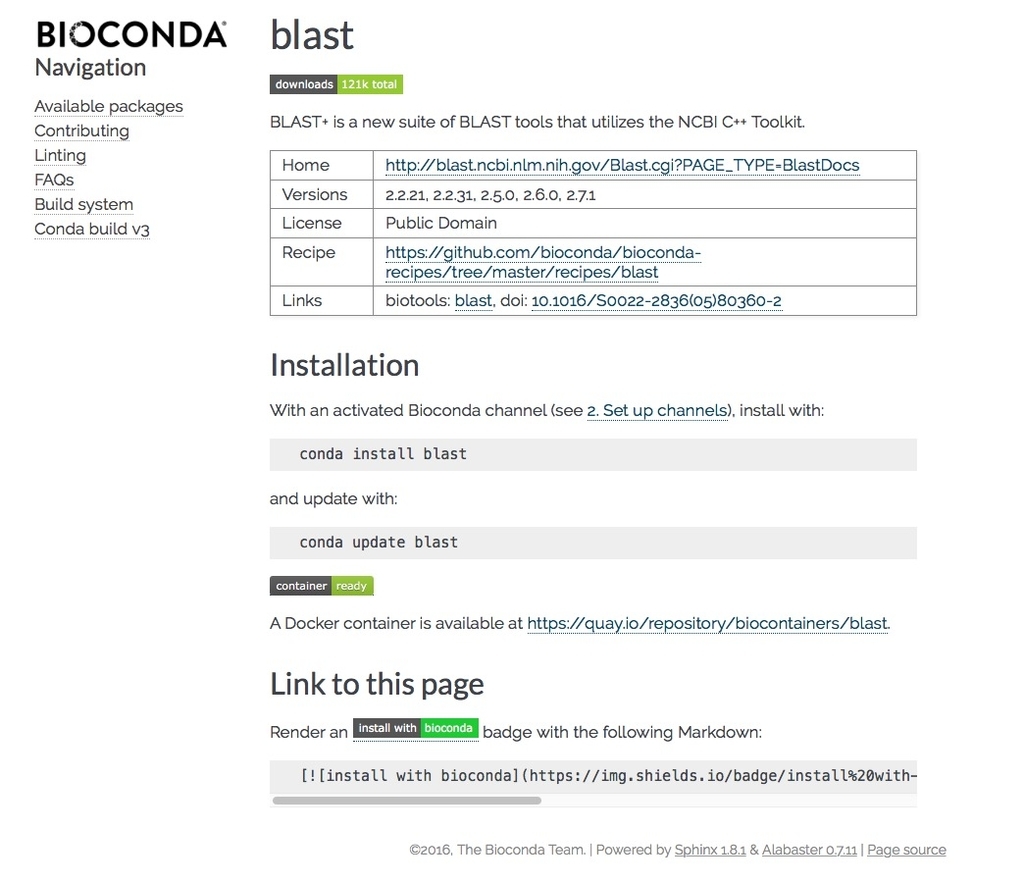
方法2、Biocondaからイメージを探してpull
BioContainersはBiocondaと統合されているので、BiocondaのHPからツールを探せばdockerイメージリンクも見つかる。ただし、Bioconda全てのツールにイメージが用意されているわけではないし、dockerイメージからの検索にも対応していないので使いにくい。あればラッキー、くらいのイメージ。
https://bioconda.github.io/recipes.html#recipes
Dockerfiesが作成されているツールだと、中央付近にイメージへのリンクがある。
リンク先からdocker pullのコードをコピペする。手順は方法1と同じになるので省略する。
方法3、Docker hubからイメージを探してpull
Docker hubはpublicイメージ共有サイトなので、アップされているイメージのほとんどは基礎研究とは無関係のものになる。ツール名で検索はできるものの、たくさんの人が"public"アクセスでイメージを上げており、ツール名だけで検索すると、同名のイメージが複数見つかったりする(例えばxxx/samtoolsがたくさん見つかる)。pull数や前半のxxxであたりはつけられるが(例えば biocontainers/blastならBioContainersのイメージ)、中にはツールがビルドできてないもの、テスト中のもの、動きはするが説明がゼロで信頼性が不明のものもある。論文中で言及されたり、オーサーが直接上げたイメージをpullするのであれば問題ないが、そうでないなら、言い方は悪いが、知らないままジョーカーを引っぱっている可能性もある。
方法4、Dockerfilesからイメージをビルド
Dockerfileの定義に従ってイメージをビルドする。この方法であれば不要なコマンドが仕込まれて入れば排除できるし、個人的に使うツールが入ってなければビルドするようにカスタマイズできる。何よりdokcerの勉強になる。
このページは基本的にBioContainersの紹介なので、BioContainersのHPからDockerfileをコピペする手順と、BioContainersのGithubレポジトリを丸ごとcloneして、そこからビルドする手順を紹介する。また、最後に、BioContainersには関わっていないツールのイメージビルドについてかんたんに触れる。
"方法1"と同じようにBioContainersのツール一覧にアクセスし、そこからblastのコンテナページにジャンプする。
https://hub.docker.com/u/biocontainers/
blastのコンテナページに飛んだら、上からDockerfilesのタブを選択する。
https://hub.docker.com/r/biocontainers/blast/~/dockerfile/
表示されたコードをプレーンテキストエディタに貼り、Dockerfileという名前で新しく作ったディレクトリに保存する。そのままビルドする。
emacs Dockerfile
#emacs編集ウィンドウ内にペースト、保存して閉じる
#イメージ名をblastとしてビルド
docker build -t blast .
Dockerfile記載内容が1行ずつ実行され、ビルドされる。
イメージの作成手順は以上となる。しかしこの手順だと、外部ファイルが必要な命令が書いてあるとうまく行かない。ということで、BioContainersのコンテナを前もってcloneしておく方が無難だし楽だと思う。ツールは1000以上あるようだが、Dockerfileはただのテキストファイルなので容量はしれている。
#名前をBioContainersとしてclone
git clone https://github.com/BioContainers/containers.git BioContainers
cd BioContainers/blast/2.2.31/
#イメージ名をblastとしてビルド
docker build -t blast .
ベースイメージのダウンロードの他、必要なツールのダウンロード&ビルドが行われたりする。時間はDockerfileの記載内容により変わる。しばらく待つ。
ビルドが終わったらイメージがあるか確認する。
docker images
docker images
REPOSITORY TAG IMAGE ID CREATED SIZE
blast latest 06e20d67e8c5 21 minutes ago 2GB
blastのイメージができている。
pullするかbuildしてイメージが準備したら、いよいよコンテナを立ち上げる。
step4、コンテナを立ち上げる
dockerのrunコマンドでコンテナを立ち上げ、そのまま中に入る(run -iで同時実行)。
docker run -i -t blast
$ docker run -it blast
biodocker@12cf739e8957:/data$
ユーザーbiodockerとしてログインしている。
blastにパスが通っているか確認。
> which blastn
$ which blastn
/opt/conda/bin/blastn
blastの実行ファイルは/opt/conda/bin/bin/にある。パスが通っているので、すぐに使用できる。抜けるときは"exit"とうつ。起動したまま一旦抜けるにはデタッチする(Ctrl キー+ PQ)。
手順は以上だが、もう少しコンテナの扱い方の基本を書いておく。
デタッチしたら、コンテナのSTATUSとIDを確認。
docker ps -a #全コンテナを確認。-aをつけないと起動中のコンテナだけ
$ docker ps -a
CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES
89bd500c45088 blast "/bin/bash" 37 hours ago Exited (0) 2 hours ago unruffled_bose
再びコンテナを立ち上げるときはstartとexecコマンドを使う。
#docker ps -aで確認したIDを指定
docker ps -a
#IDを指定してstart
docker start 9bd500c45088
#startしただけでは中に入らない。ここで起動中のイメージを確認
docker ps
docker ps
CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES
9bd500c45088 blast "/bin/bash" 37 hours ago Up 2 minutes unruffled_bose
起動している。
アタッチして中に入る。
docker attach 9bd500c45088
$ docker attach 9bd500c45088
biodocker@9bd500c45088:/data$
中に入った。exit、またはCtrl + Dでコンテナを終了する。
exitしたら、もう使わないのでこのコンテナを消去する。イメージを消すのは"docker rmi"、コンテナを消すのは"docker rm"。
docker rm 9bd500c45088
$ docker rm 9bd500c45088
9bd500c45088
——
#イメージも消す
docker rmi blast
説明は以上。
まとめ
導入が簡単なblastを例にしたのであまりメリットが伝わらなかったかもしれませんが、一般に、ある分野の方法論/アルゴリズムが飽和してくると、今度はそれを全自動で実行するユーザーフレンドリーなツールや、たくさんのツールを結びつけて感度や精度を引き上げるツールの発表が増えてきます。後者のようなツールは依存するツールも多く、全てのツール、ライブラリのビルドには数時間かかる場合もあります。それに伴い、ツールのバージョン違いによるコンフリクトが発生してビルドに失敗する確率も増えてきます。そうなると、当然、数時間もかけたビルド作業が水の泡になります。また、ビルドできても、動かすとエラーが出つつ結果を出したりして不安になったことはありませんか?
dockerのイメージを使うことで、導入の障壁を解決し、開発者と同じ環境で解析を始めることができるようになります。後者が一番重要で、ツールの再現性が担保されます。もう一つのメリットは、散々言われていることですが、すぐに使えることでしょう。pullはあっという間に終わりますし、起動も一瞬です。私の場合、一番多い使い方は、ツールの使い捨て環境としての場です。素のlinuxマシンをすぐに立ち上げ(*2)、コンフリクトを心配せずツールをテストし、終わったらすぐ廃棄できるのは便利だと思いませんか?これらが、インフォマティクスの研究者にとってdockerを使うことの主要なメリットだと考えています。実際、BioContainersに限らずDockerfilesを準備するツールも増えてきています。
もちろん、メリットがあればデメリットありで、メリットをコインの表にできる使い方を知ることが肝要になります。なんでもかんでもdockerでいいという訳ではありません。DockerはカーネルなどホストOSと重複する部分を省略してオーバーヘッドも軽減することでクラシックな仮想環境より軽くなっています。しかし、当然ネイティブ環境ほど速くはありません。むやみに使うと、オーバーヘッドから予想される以上に著しくパフォーマンスが低下する可能性があります(highly subjectiveだが、色々テストしているとそう感じることがある)。また、コンテナを共有していても、増えてくればあっという間に数百GB容量を食ってきます。使って、どのようなことに向いているか自分なりに答えを出すことが重要です。
というわけでdokcerに興味が出たら自分で様々なソースを調べてみてください。関連本やまとめ、学習動画はたくさん出てきています。linuxのファイル構造を調べたり、ユーザを作ったり、ポートを開けたりして遊んでいるうちに、unixの実践的な勉強にもなったりします。私自身、調べていると、使えると便利そうだなという使い方がいくつも出てきます。これからも勉強して紹介していきたいと思います。
エキストラstep
基本的な使い方がわかったら、簡単なのでDockerfileを自分で作れるようにする。検索して出てきたStrainEstというツールのDockerfileを開いてみる。
https://github.com/compmetagen/strainest/blob/master/docker/Dockerfile
色々書かれているが、必須なのは1行目のFROM命令のところだけで、FROM xxxで、ベースイメージが決まる(FROM説明リンク)。この1行だけあれば、最小限の構成のlinuxイメージを作成することができる。例えば、
FROM ubuntu:16.04
という1行だけ書かれたDockerfileを用意すれば、docker buildの実行で最小構成のubuntuイメージを作成できる。最新のubuntuが欲しければ、FROM ubuntu:latestに書き換える(link)。上の例のようにFROM debian:9ならdebian9(link)、FROM centos7ならCentOS7 (latest)がベースイメージになる(link)。Red Hatも使えるが、サブスクリプションが必要。
"FROM ubuntu:16.04"1行だけのDockerfileを作ってビルドしてみる。データをfetchしてくるので実行時はネットに繋がっている必要がある。
#testディレクトリで実行する
mkdir test && cd test/
#Dockerfile作成
echo FROM ubuntu:16.04 > Dockerfile
#イメージ名はtestでビルド(build)。-tで作成するイメージ名指定
docker build -t test .
$ docker image build -t test .
Sending build context to Docker daemon 2.048kB
Step 1/1 : FROM ubuntu:16.04
16.04: Pulling from library/ubuntu
3b37166ec614: Pull complete
504facff238f: Pull complete
ebbcacd28e10: Pull complete
c7fb3351ecad: Pull complete
2e3debadcbf7: Pull complete
Digest: sha256:45ddfa61744947b0b8f7f20b8de70cbcdd441a6a0532f791fd4c09f5e491a8eb
Status: Downloaded newer image for ubuntu:16.04
---> b9e15a5d1e1a
Successfully built b9e15a5d1e1a
Successfully tagged test:latest
——
ビルドできた。
イメージができているか確認する。
$ docker images
REPOSITORY TAG IMAGE ID CREATED SIZE
test latest b9e15a5d1e1a 3 weeks ago 115MB
できている。
Dockerfileの 2行目以降では、apt-getの更新や依存ツール導入を行なっている。"RUN命令 + commnad"で実行できる。

RUN apt-get update
RUN apt-get install -y build-essential
RUN apt-get install -y pkg-config
というように1行ずつ実行してOK(RUN命令は何回でも使える)。

後半はENVで環境変数を定義し、WORKDIRで現在のパスを定義し(要は/temp/sickle-1.33に移動している)、再びRUNでコマンドを実行している。あとはrunすればStrainEstをローカルで使うことができる。Dockerfileの詳細はベストプラクティスで確認してください。
Dockerfile のベストプラクティス — Docker-docs-ja 1.9.0b ドキュメント
一度exitしたイメージにもう一度入るには、スタートしてアタッチします。
#IDを確認
docker ps -a
#start
docker run <ID>
#attach
docker attach <ID>
追記
ビルドで全ての作業を行わなくても、立ち上げて中で追加インストールを行い、それからcommitし直すのが簡単です。commitするには、中で作業後、exitしてcommitコマンドを実行するだけです。同じ名前で上書きもできます。
docker commit <ID> ubuntu
コマンドがhost osでも動くなら、dokcerコンテナ内でビルドされた実行ファイルを、ホスト側に持っていき、ホスト側でランする方がパフォーマンスが出ます。
docker run --rm -v $(pwd):/data ubuntu cp xxx /data
dockerイメージとしてしか配布されていないプログラムで利用できるtipsです。
sudo usermod -aG docker <YOUR_USER_NAME>
dockerイメージとしてしか配布されていないプログラムで利用できるtipsです。
追記
引用
BioContainers: an open-source and community-driven framework for software standardization
da Veiga Leprevost F, Grüning BA, Alves Aflitos S, Röst HL, Uszkoreit J, Barsnes H, Vaudel M, Moreno P, Gatto L, Weber J, Bai M, Jimenez RC, Sachsenberg T, Pfeuffer J, Vera Alvarez R, Griss J, Nesvizhskii AI, Perez-Riverol Y
Bioinformatics. 2017 Aug 15;33(16):2580-2582
参考
*1
Dockerについてちゃんと勉強するなら、市販のDockerマニュアル、ドットインストールや講習会、you tuberやネットワーク管理者?の方々の動画が聞きやすくお勧めです。
ドットインストール
https://dotinstall.com/lessons/basic_docker
https://www.youtube.com/results?search_query=docker+linux+学習
*2
virtual machineや、parallels,fusionsは、早くても起動に20秒ほどかかる。その間、CPUリソースをたくさん占有する。
追記
@Damjana_Kas @fanofactor We are trying to add Docker/Singularity containers to @BioContainers for #Bioinformatics training. These efforts will help trainnes to easily find and discover the containers and materials. Here the chat for discussing the details https://t.co/FDx7j2KKKN
— Yasset Perez-Riverol (@ypriverol) March 14, 2019
windowsユーザーの方はこちらの記事がとても参考になると思います。
関連
2020 11/11
メモ
Dockerファイルをホームディレクトリーにおいてdocker buildを実行しない。