2019 4/30 インストール方法修正
2022 1/23 追記
発現が負の二項分布に従うと仮定した検定法。正規化はTMMで行う。FPKM/RPKM補正のcufflinksより正しくDEGの検出ができる検定法とされる。詳細は門多先生のスライドやdry本の序章の正規化の話を読んでください。以下のマニュアルも大変参考になります。後半で正規化法について詳しく書かれています(英語です)。
http://chagall.med.cornell.edu/RNASEQcourse/Intro2RNAseq.pdf
こちらはTMM正規化の流れが丁寧に書かれています。
http://www.nathalievilla.org/doc/pdf/tutorial-rnaseq.pdf
こちらはビギナー向けマニュアルです。FPKMは論文で使われれてても使うべからず、と説明されています。
http://chagall.med.cornell.edu/RNASEQcourse/Intro2RNAseq.pdf
edgeRのマニュアルが一番詳しい。ケーススタディも豊富。
https://bioconductor.org/packages/release/bioc/vignettes/edgeR/inst/doc/edgeRUsersGuide.pdf
インストール
R #Rに入る
#install
> if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("edgeR")
ここでは3反復で行われたstress条件とnormal条件の2群間比較のデータを検定してみる。データは前もってtophat2でアライメントして、featurecountsでカウントしてある。
3反復 2群比較のデータ(リンク)
↓
tophat2でmapping(リンク)
↓
featurecountsでカウント(リンク)。
↓
正規化と検定(今ここ)
featurecountsの出力は以下のようなものである。先頭1行目はコメント行(#で始まる)で、2行目がheader行、3行目以降がデータとなっている。リードのカウント数を記載した列は7-12列である。

読み込む前に、edgeRでは不要な2-6列目を削る。
cut -f 1,7-12 all_featureCounts_strand_specific.txt > read_count.txt
長さ情報も捨てているが、edgeRは長さについて正規化をしないのでこれでよい。
Rの起動とedgeRの読み込み。
>R #R起動
library("edgeR")
リードデータの読み込み
count <- read.table("read_count.txt",sep = " ", header = T, row.names = 1)
先頭のコメント行は無視され、2行目がヘッダーとなる。row.names=1で1 から始まる番号が行名として生成される。
行列データに変換。
count <- as.matrix(count)
dim(count) #行列の行と列数を確認
#normalを要素1-3、stressを要素4-6としたベクトルの識別ラベル、"group"を作成。
group <- factor(c("normal", "normal", "normal", "stress", "stress", "stress"))
edgeRのDGEList(リンク)を使い、tableをオブジェクト化。
d <- DGEList(counts = count, group = group) #DGEListオブジェクトを作成してdに格納
2022 1/23
任意で低発現遺伝子のフィルタリングを行う。上で定義したグループを考慮に入れる(クラシカルな2群間比較ではなく複雑な実験デザインの時はdesignを定義して、それを使う)
keep <- filterByExpr(d, group=group)
#低発現遺伝子をフィルタリング
d <- d[keep, , keep.lib.sizes=FALSE]
=> テストした時は3万遺伝子のうちの1万5千遺伝子ほどが除外された。
続いてedgeRのcalcNormFactors(リンク)を使い、TMM正規化の係数を計算
d <- calcNormFactors(d) #TMM正規化
dの中身を確認。
d$samples
group lib.size norm.factors
tophat_high_sensitivity_control1 normal 25628799 0.8480974
tophat_high_sensitivity_control2 normal 32962605 0.9635817
tophat_high_sensitivity_control3 normal 27119594 0.9932421
tophat_high_sensitivity_stress1 stress 25379772 1.0533018
tophat_high_sensitivity_stress2 stress 32962605 0.9635817
tophat_high_sensitivity_stress3 stress 18642271 1.2138616
dの右端の列(norm.factors) の係数とlib.size(ライブラリーサイズ; トータルのリード数)の積を計算した値を使い補正される。
すなわち
ライブラリサイズ(トータルリード数)x 正規化係数
を計算した値で各サンプルを割ることになる。このように、edgeRはライブラリサイズと変動が少ないハウスキーピング遺伝子の発現量を使った正規化法である。
検定を行う。edgeRのマニュアル通り進める(リンク)。
d <- estimateCommonDisp(d) #全遺伝子commonの分散を計算 (リンク)
d <- estimateTagwiseDisp(d) #moderated tagwise dispersionの計算 (リンク)
result <- exactTest(d) #exact test(リンク)
検定結果のtop10をedgeRのtopTagsコマンドで表示(リンク)。
topTags(result)
下のようになった。
> topTags(result)
Comparison of groups: stress-normal
logFC logCPM PValue FDR
AT1G09350 10.567190 6.101216 3.137694e-08 0.0002715988
AT1G77120 6.341318 8.882087 1.553797e-07 0.0006724831
AT1G04570 4.959992 5.609363 1.371816e-06 0.0039581457
AT1G01060 3.988584 5.278486 2.636780e-06 0.0057059928
AT1G20440 3.344557 8.131254 6.369575e-06 0.0110270090
AT1G09780 2.740267 8.804577 1.122465e-05 0.0132076639
AT1G16850 3.703996 8.951409 1.195946e-05 0.0132076639
AT1G62710 3.903980 6.677934 1.220671e-05 0.0132076639
AT1G20450 2.770247 5.902330 1.708683e-05 0.0156210368
AT1G18900 3.025852 5.706347 1.804648e-05 0.0156210368
解析結果を result.txt ファイルに保存。
table <- as.data.frame(topTags(result, n = nrow(count))) #toptagsで全行指定してデータフレームに変換
write.table(table, file = "result.txt", col.names = T, row.names = T, sep = " ", quote = F)
中身を確認。FDR<0.05を満たす遺伝子を確認する。
| logFC | logCPM | PValue | FDR | |
| AT1G09350 | 10.16307525 | 4.527258796 | 4.27E-09 | 0.000107078 |
| AT4G14690 | 7.354181425 | 4.435418248 | 6.52E-09 | 0.000107078 |
| AT3G22840 | 7.228134725 | 5.012923253 | 1.98E-08 | 0.000216609 |
| AT4G12470 | 6.474020331 | 5.655042141 | 3.42E-08 | 0.000280991 |
| AT3G55580 | 5.653555557 | 5.139781896 | 5.43E-08 | 0.000287357 |
| AT5G52310 | 5.124986828 | 5.916626611 | 5.46E-08 | 0.000287357 |
| AT4G21020 | 10.91063112 | 2.178134308 | 6.13E-08 | 0.000287357 |
| AT1G77120 | 6.138518805 | 7.401568486 | 1.88E-07 | 0.000689566 |
| AT3G09450 | 4.657253152 | 3.423533639 | 1.89E-07 | 0.000689566 |
| AT1G01060 | 4.368122835 | 3.763121902 | 3.53E-07 | 0.00109118 |
| AT2G07715 | 4.257034937 | 3.953448462 | 3.66E-07 | 0.00109118 |
| AT1G04570 | 5.171798484 | 4.292665281 | 4.20E-07 | 0.001149004 |
| AT4G33070 | 6.746791572 | 2.576092914 | 6.86E-07 | 0.001666543 |
| AT5G17300 | 5.254208837 | 2.007723134 | 7.11E-07 | 0.001666543 |
| AT5G37260 | 4.92542501 | 3.35910879 | 7.80E-07 | 0.001707349 |
| AT4G02280 | 4.22594104 | 7.358454251 | 8.83E-07 | 0.00181116 |
| ATCG00190 | 3.645754145 | 5.62775771 | 1.02E-06 | 0.001974423 |
| AT5G59310 | -5.174304818 | 2.241756997 | 1.16E-06 | 0.002122225 |
| AT3G07050 | 3.431999493 | 4.513456519 | 1.41E-06 | 0.002430182 |
| AT4G18690 | 9.538641966 | 0.830755067 | 1.73E-06 | 0.002838395 |
| AT1G04560 | 5.293711871 | 3.142572477 | 2.18E-06 | 0.003398314 |
| AT4G12480 | 5.657010399 | 2.190044061 | 2.28E-06 | 0.003398314 |
| AT5G15960 | 6.905008258 | 6.46085288 | 2.47E-06 | 0.003532814 |
| AT1G26790 | 6.025415721 | 0.792018063 | 2.71E-06 | 0.003703112 |
| AT4G25433 | 5.27934755 | 1.508481739 | 2.89E-06 | 0.003703112 |
| AT2G01008 | 3.91336062 | 3.369718716 | 2.99E-06 | 0.003703112 |
| AT3G12860 | 3.56164772 | 2.72274909 | 3.05E-06 | 0.003703112 |
| AT4G17090 | 4.057594021 | 8.295083362 | 4.03E-06 | 0.004719979 |
| AT1G62710 | 3.833187835 | 5.158604303 | 4.80E-06 | 0.005432695 |
| AT2G37770 | 3.401608114 | 3.427762539 | 5.05E-06 | 0.00552434 |
| AT1G20440 | 3.238103764 | 6.394609506 | 5.31E-06 | 0.005624722 |
| AT5G05270 | 2.944176517 | 6.213369092 | 6.00E-06 | 0.006156491 |
| AT4G15430 | 2.995358995 | 3.898170281 | 6.20E-06 | 0.006168644 |
| AT1G20450 | 2.899460216 | 3.89279605 | 7.87E-06 | 0.007179477 |
| AT3G59670 | 2.900976613 | 4.387722673 | 8.10E-06 | 0.007179477 |
| AT5G16930 | 3.00078392 | 4.028316392 | 8.27E-06 | 0.007179477 |
| ATCG00180 | 3.045154042 | 3.083318107 | 8.28E-06 | 0.007179477 |
| AT1G09780 | 2.784284171 | 6.845328834 | 8.31E-06 | 0.007179477 |
| AT2G33210 | 2.89055242 | 4.722119045 | 8.71E-06 | 0.007262903 |
| AT5G17030 | 3.074060891 | 4.463097295 | 9.06E-06 | 0.007262903 |
| AT5G20830 | 2.957190377 | 5.8454128 | 9.07E-06 | 0.007262903 |
| AT5G07990 | 3.307887967 | 3.536393301 | 9.56E-06 | 0.007470713 |
| AT1G47510 | 2.816286516 | 3.981822721 | 1.01E-05 | 0.007663266 |
| AT1G16850 | 3.623257952 | 7.724435616 | 1.03E-05 | 0.007663266 |
| AT3G12270 | 2.775114741 | 4.098894865 | 1.08E-05 | 0.007791155 |
| AT4G37910 | 3.026726294 | 5.176987655 | 1.09E-05 | 0.007791155 |
| AT4G10120 | 2.688153454 | 4.612048946 | 1.21E-05 | 0.008353586 |
| AT4G19975 | 6.19617109 | 0.664252933 | 1.22E-05 | 0.008353586 |
| AT1G18900 | 2.744659249 | 3.962487564 | 1.28E-05 | 0.008546496 |
| AT5G59320 | -3.604847868 | 3.23617687 | 1.30E-05 | 0.008546496 |
| AT2G23120 | 3.079019743 | 4.469576169 | 1.55E-05 | 0.009847484 |
| AT3G03060 | 3.090251188 | 3.167877441 | 1.57E-05 | 0.009847484 |
| AT1G06720 | 2.803080797 | 3.175083458 | 1.61E-05 | 0.009847484 |
| AT5G15970 | 3.377579491 | 4.03069361 | 1.62E-05 | 0.009847484 |
| AT4G25630 | 2.775891378 | 7.076363693 | 1.68E-05 | 0.010011705 |
| AT2G05520 | 3.782813845 | 5.971256405 | 1.81E-05 | 0.010598322 |
| AT2G40010 | 3.044930685 | 3.582251663 | 1.90E-05 | 0.010921949 |
| AT1G29940 | 2.593259085 | 4.110832625 | 2.01E-05 | 0.011367248 |
遺伝子は合計32834行ある。そのうちP-value が0.01以下のtrasncriptsは1080で、さらにFDR<0.01を満たすのは54trasncriptsだけだった(FDRが太字の行)。
FDR < 0.05 の遺伝子を赤色でプロットする。
is.DEG <- as.logical(table$FDR < 0.05) #logical クラス(true、false)に判定付きで変換。
DEG.names <- rownames(table)[is.DEG] #trueだけDEG.namesに収納
edgeRのplotSmear(リンク)を使いM-A plotを描画する。
plotSmear(result, de.tags = DEG.names, cex=0.3)
plotSmearを使うとM-A plotの図を容易に描画できる。オプションはリンク参照。
FDR < 0.05

上が出力だが、DEGの数が妙に少ない。また以前にテストしたナズナ(Col)のデータとかなりplotの形が違う。下が以前のデータ。
FDR < 0.05

高発現の遺伝子ほどばらつきが少ないため、logFCが0に値が収束していくのだが、今回のデータはそうなっていない。ナズナのゲノムを使ってアライメントしているので、ランダムアライメントを多く拾ってしまっているのかもしれない。
DAVIDを使い、FDR<0.05の種類を調べてみる。
KEGG_PATHWAY でribosome合成系がhitした。Ribosome biogenesisのpathwayだった。
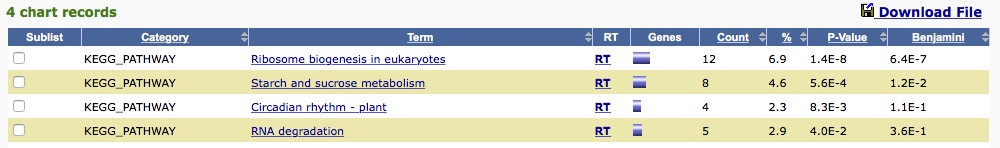
1つ開いてみる。
Ribosome

緑になっているのが、ナズナでアノテーションが付いている遺伝子である。また、緑のobxの一部には赤色の星が付いてる。この星がDABIDの検索で見つかった今回DEGと判定された遺伝子になる。
Functional Annotation Chartも確認する。

今回調べた遺伝子に載っているpathwayで多く見つかるもの。
FDR < 0.05のread_count.txtを使い、クラスター解析。あらかじめ正規化してある。
count <- read.table("read_count_TMM_cali.txt", sep = " ", header = T, row.names = 1)
count <- as.matrix(count)
rho <- cor(count, method = "spearman")
d <- dist(1 - rho)
h <- hclust(d, method = "ward.D2")
plot(h)
階層クラスタリング | スピアマンの順位相関係数を利用したクラスタリング

controlとstressのtriplicateで分類されなかった。普通ならこのデータには何か問題がある可能性がある、となるが今回はアライメントとgtfに穴があるのでそこも怪しい。
NCBI GEOで、DEG判定された遺伝子が、どのような条件で変動する遺伝子なのか調べてみる。
NCBI GEOにアクセス。
https://www.ncbi.nlm.nih.gov/geo/
↓
search by GEO profileに移動(リンク)。
↓
TAIR IDを入れて検索を開始(例)。

発現が有意に変動しているトランスクリプトーム実験のデータが表示される。右の方にあるオレンジのバーが、検索した遺伝子の発現量の変化を表す。
↓
どれか1つの実験を選び中に入る。ここでは一番上を選んだ(リンク)。
この実験で変動している遺伝子を調べてみる。下の方にあるCompare 2 sets of samplesをクリック。

step1、両側t検定を選び、例えばp<0.01を選択。
↓
step2、対象とするデータセットを選択(おかしなデータがない限り、基本全て選択)。

↓
step3、DEGを検索。
↓
125遺伝子がDEGと検出された(リンク)。検出されたのは全てこの実験のデータであることを確認する。
↓
遺伝子名をダウンロードする。その前に全部収まるよう表示件数を200に変更。

↓
右のdownloadからダウンロード。
↓
遺伝子名を一括で入手できた。

↓
pathwayも調べておく。右のpathwayをクリック。
↓
pathwayが10検出された。KEGGでのhitが2(1つは全体図)、REACTOMEが8検出されている。

どれか1つクリックしてみる。ここではKEGGのsecondary metaboliteを選択。
↓
http://www.kegg.jp/pathway/ath01110

左端のsecondary metaboliteをクリックして場所を確認する。
↓
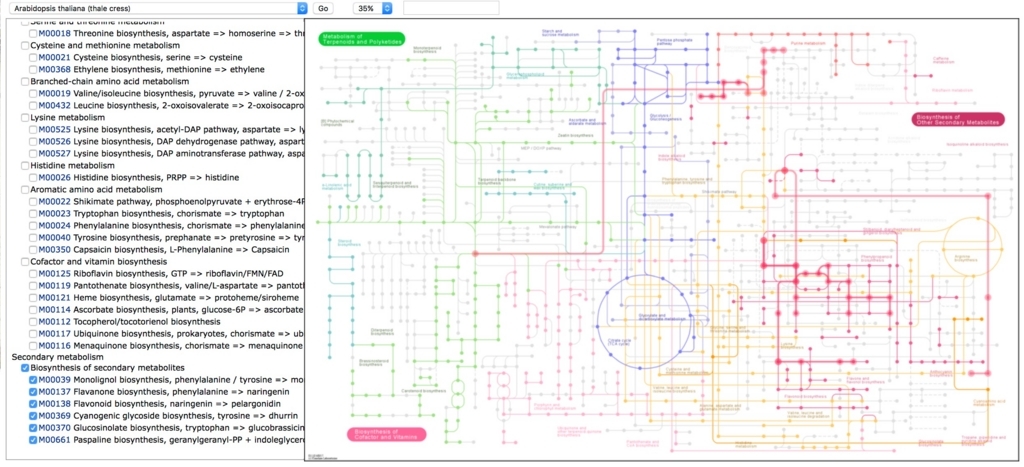
右のほうにある。ここから先はわかりにくいので、kEGG pathwayのtopに移動してsecondary metaboliteで再検索する。
↓

下の方の
01060 Biosynthesis of plant secondary metabolites
を選択。
↓

Referenceしかない。研究が不十分でアノテーションが進んでいないようである。
PANTHERでも機能を解析してみる。
リストを入れ、下のFunctional classification viewed in pie chartにチェックをつけてsubmit。

GOの第一階層を選択する。Biological processを選択した。

response to stimulus (GO:0050896) ![]()
![]() というtermも出てきている。
というtermも出てきている。
似た機能として、PANTHER Overrepresentation Testなどがあり、top pageから実行できる。
- 植物の転写因子データベースPlantTFDB
http://planttfdb.cbi.pku.edu.cn/tf.php?sp=Ath&did=AT3G02310.1
既知転写因子にhitするかどうか、blastで探索することができる(リンク)。同様のサービスとして、PlnTFDBにもBLAST検索機能がある(リンク)。
- BiomartでID変換(現在工事中らしい)。
http://www.biomart.org (=> ID変換リンク)
- 何らかの転写因子や相互作用しそうな遺伝子がDEGとして検出されたら、GeneMANIAで関連する遺伝子を探すのも良い。

arabidopsisを選択。遺伝子名で検索できるが、TAIRのIDにも対応している。

結果は、遺伝子名があるものは遺伝子名で表示される。

右にedgeの情報源が表示されている。co-expression以外のチェックを外してみる。

紫の線だけ残る。真ん中のクラスターは共発現することがあるようだ。

右のカラムを展開すると、どのような実験条件か確認することができる。

チェックボックスをクリックして、例えば自分の解析に似た実験だけ選んでedgeを確認することができる。右端のリンクから論文を確認できる。

その他、nodeの上でクリックして、その遺伝子だけで再解析することもできる。
GeneMANIAcytoscapeのpluginもあるようだ。
ヒートマップも書いてみる。ただし、このような2群間比較でヒートマップを書く意味はあまりない。 ヒートマップは、例えば遺伝子Aを壊したA株と遺伝子Bを壊したB株をWTと比較し、 そのfold changeをA/WT、B/WT、で比較して共通のpathway遺伝子などを図示するなどして使う。あくまで忘備録として残しておく。
ここではheatmapなど様々な図を作画できるg.plotsを使う。様々な条件でフィルタリングができるgenefilterも導入する。
まずg.plotsとgenefilterをインストール。
biocLite("genefilter")
biocLite("gplots")
ライブラリをロード。
library(genefilter)
library(gplots)
読み込むデータはedgeRで検定し、TMM正規化したカウントデータ。FDR<0.05でなくp< 0.05を全て使う。読み込むデータは以下のようなフォーマットになっている。1行目は各列の注釈、1列目は遺伝子名となっている。
| Geneid | control1 | control2 | control3 | stress1 | out | stress3 |
| AT1G01060 | 2 | 15 | 3 | 152 | 15 | 104 |
| AT1G01100 | 1002 | 1147 | 859 | 4031 | 1147 | 2427 |
| AT1G01320 | 72 | 170 | 112 | 715 | 170 | 624 |
| AT1G01390 | 1 | 20 | 6 | 24 | 20 | 38 |
| AT1G01470 | 4 | 7 | 1 | 34 | 7 | 68 |
| AT1G01580 | 6 | 10 | 8 | 32 | 10 | 22 |
| AT1G01790 | 197 | 481 | 240 | 948 | 481 | 690 |
| AT1G01950 | 18 | 50 | 30 | 74 | 50 | 67 |
| AT1G02370 | 1 | 5 | 1 | 12 | 5 | 14 |
| AT1G02460 | 3 | 4 | 2 | 75 | 4 | 58 |
| AT1G02690 | 12 | 23 | 17 | 60 | 23 | 53 |
| AT1G02780 | 1400 | 3271 | 1973 | 8204 | 3271 | 6250 |
| AT1G02870 | 12 | 53 | 27 | 82 | 53 | 69 |
| AT1G02920 | 430 | 346 | 773 | 81 | 346 | 259 |
| AT1G02952 | 204 | 198 | 168 | 38 | 198 | 34 |
| AT1G03110 | 42 | 67 | 52 | 225 | 67 | 145 |
| AT1G03360 | 15 | 31 | 11 | 63 | 31 | 35 |
| AT1G03530 | 4 | 12 | 10 | 46 | 12 | 28 |
ファイル名はFDR0.05.txtとした。
データを読み込む。
data <- read.delim("result_FDR0.05.txt", header=TRUE, row.names=1)
先ほどcountとして読み込んだデータが残っているなら、それでもよい。
gplotsのheatmapを使う。データは行列でないといけないので、as.matrixで変換している。
heatmap(as.matrix(data), margin=c(6,4), main="Heat Map 1 (Raw Data)")
- margin numeric vector of length 2 containing the margins
- main, xlab, ylab main, x- and y-axis titles; defaults to none.
以下のようになった。

一部の強い発現に引っ張られており、一部の条件だけ真っ赤になっている。
z-スコア(解説リンク)に変えた方がクラスタリングは綺麗になる。genefilterのgenescaleを使い、Zスコアに変換。
data.z <- genescale(data, axis=1, method="Z")
- axis An integer indicating which axis of m to scale.
- method Either "Z" or "R", indicating whether a Z scaling or a range scaling should be performed.
改めてヒートマップを描画。
heatmap(as.matrix(data.z), margin=c(6,4), main="Heat Map 2")

Zーscore化する前は強い変動をしている遺伝子にクラスタリングが引っ張られてしまっていたが、それが無くなっている。
heatmap.2(as.matrix(data.z), col=greenred(75), scale="none", key=TRUE, symkey=FALSE, density.info="none", trace="none", cexRow=1, margin=c(6,8), main="Heat Map 3", Colv=NA)
- col colors used for the image. Defaults to heat colors (heat.colors).
- scale character indicating if the values should be centered and scaled in either the row direction or the column direction, or none. The default is "none".
- key logical indicating whether a color-key should be shown.
- symkey Boolean indicating whether the color key should be made symmetric about 0. Defaults to TRUE if the data includes negative values, and to FALSE otherwise.
- density.info character string indicating whether to superimpose a 'histogram', a 'density' plot, or no plot ('none') on the color-key.
- trace character string indicating whether a solid "trace" line should be drawn across 'row's or down 'column's, 'both' or 'none'. The distance of the line from the center of each color-cell is proportional to the size of the measurement. Defaults to 'column'.
- cexRow, cexCol positive numbers, used as cex.axis in for the row or column axis labeling. The defaults currently only use number of rows or columns, respectively.
- Colv determines if and how the column dendrogram should be reordered. Has the options as the Rowv argument above and additionally when x is a square matrix, Colv="Rowv" means that columns should be treated identically to the rows.
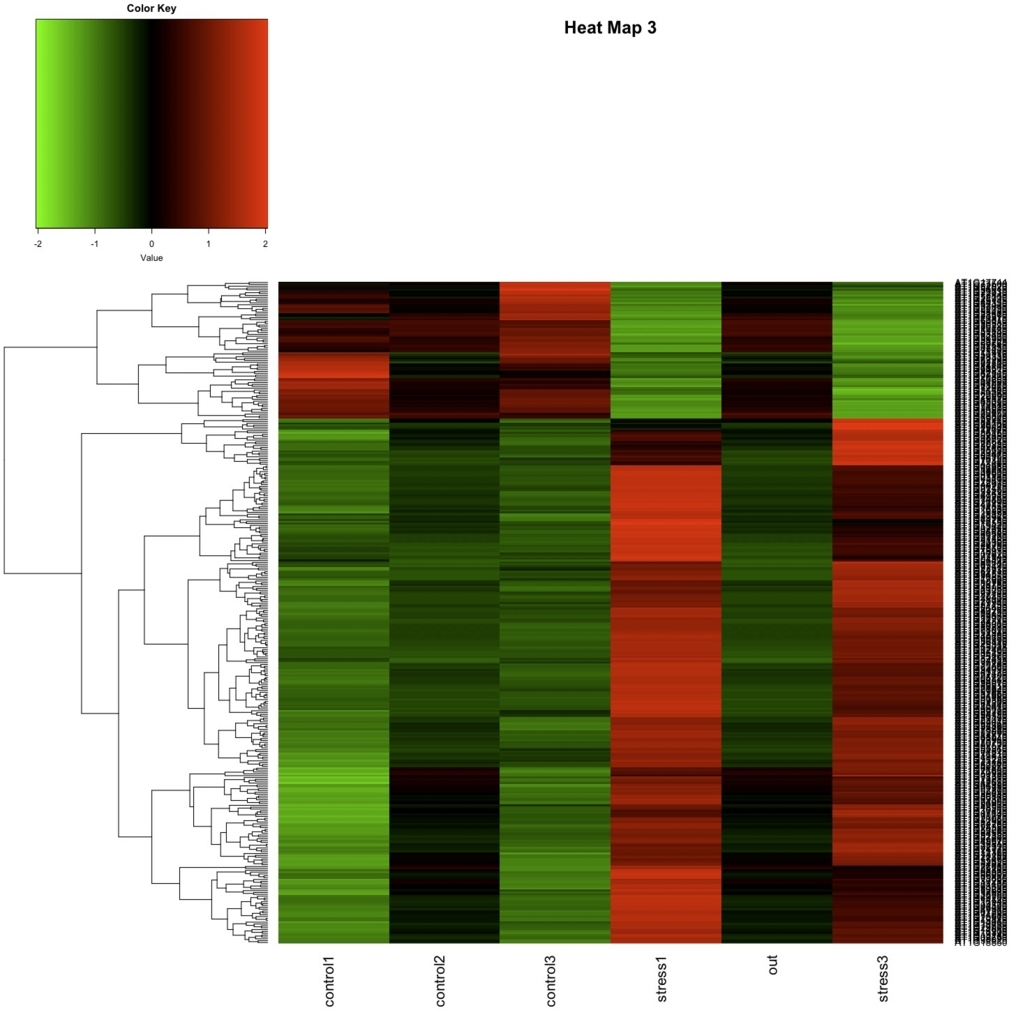
heatmapのリンクには様々なheatmapの例が載っている。コピペするだけで図が書けるので、好みの図がどうなコードで書けるのかテストしてみるとよい。
追記
勉強会用資料
引用
edgeR: a Bioconductor package for differential expression analysis of digital gene expression data.
Robinson MD, McCarthy DJ, Smyth GK.
Bioinformatics. 2010, 26(1):139-40. PubMed Abstract Bioconductor
階層クラスタリング | スピアマンの順位相関係数を利用したクラスタリング
ヒートマップ
DAVIDによるGO解析
DAVID 操作ガイド3 – 遺伝子発現解析(マイクロアレイ解析, RNA-seq)
NCBI GEO
Large-scale gene function analysis with the PANTHER classification system
Huaiyu Mi, Anushya Muruganujan, John T Casagrande & Paul D Thomas
Nature Protocols 8, 1551–1566 (2013) doi:10.1038/nprot.2013.092 Published online 18 July 2013
GO
遺伝子オントロジー | 生物学的プロセス、細胞の構成要素、分子機能
GeneMANIA
2020 11/25 こちらを読んで見てください。
関連