現在のシステムバイオロジー研究では、異なる種類のデータを別々のソースからアクセスし、別々のツールを使って視覚化しなければならないという大きな課題がある。このようなワークフローをナビゲートするために必要な高い認知的負荷は、仮説の生成に悪影響を及ぼす。そのため、すべてのデータを統合し、単一のポータルで検索・解析・可視化の機能を提供する強固な研究プラットフォームが必要とされている。ここでは、シロイヌナズナの複数レベルのデータを、ズーム可能なユーザ・インターフェイスで探索するビジュアル分析ツール、ePlant(http://bar.utoronto.ca/eplant)を紹介する。ePlantは、いくつかの公開されたWebサービスに接続して、1つまたは複数の関心のある遺伝子またはゲノム、プロテオーム、インタラクトーム、トランスクリプトーム、3D分子構造データをダウンロードする。データは、大きなものから小さなものへの概念的な階層を用いて表示される一連の可視化ツールで表示され、多くの可視化ツールは複数のデータタイプからの情報を組み合わせて表示される。この論文では、ePlantの開発について説明し、仮説生成のための統合機能を示すいくつかの例を紹介する。また、Araport上の「アプリ」としてePlantを展開するプロセスについても説明する。ePlantのコードは、他の生物種の研究にも自由に利用できる。
BAR
http://bar.utoronto.ca/eplant/にアクセスする。
遺伝子名かgene IDで検索する。例えば"AT5G60200"とタイプ、

表示された。染色体のように見えるが、線は全て発現の条件を表しており、相対的な発現強度がヒートマップ として表現されたものになる。


カーソルを合わせるとメタデータが表示される。

クリックすると、AT5G60200の発現パターンが、その発現解析の条件に沿って表示される。

左のタブから植物組織を切り替えることができる。
myzus persicaere感染の有無

根端組織

MYBの変異株

上のメニューから図の拡大縮小、

相対から絶対値への変更。

他の遺伝子との発現場所と発現強度の比較

遺伝子を選択

ヒートマップ が選んだ2遺伝子の相対的な発現強度に変わった(2遺伝子間が同じ発現量なら黄色)。

発現変動が大きい場所をマスク

S.Dの大きさを選択、

マスクされた。

パレットのマークからは色指定が行える。また、右端の歯車マークではヒートマップ の色の勾配を指定できる。

興味がある組織があれば、カーソルを合わせるとメタデータが表示される。

クリックすると追加で分析を行える。

Find genes ~ => 同じ発現パターンの遺伝子を取り出す。

遺伝子を選択して、Get data for selected genesを選択。ここではGet five more ~ をクリックして10遺伝子を選択した。
左端に10遺伝子追加され、最初の遺伝子同様の分析ができるようになった。

ヒートマップ が11遺伝子に増えた。

引用や実験条件について知りたければ右上をクリックする。

今までは顕微鏡のマーク(左端中央のボタン)だったが、他のボタンも簡単に見ていく。

iマークはその遺伝子の情報

遺伝子ID 、場所、ATGからTAAまでのコード領域DNA配列と上流下流の数十bpのDNA配列(UTR)、そしgてコード領域のタンパク質配列が表示される。
文献情報

登録されている他の植物へのナビゲーション
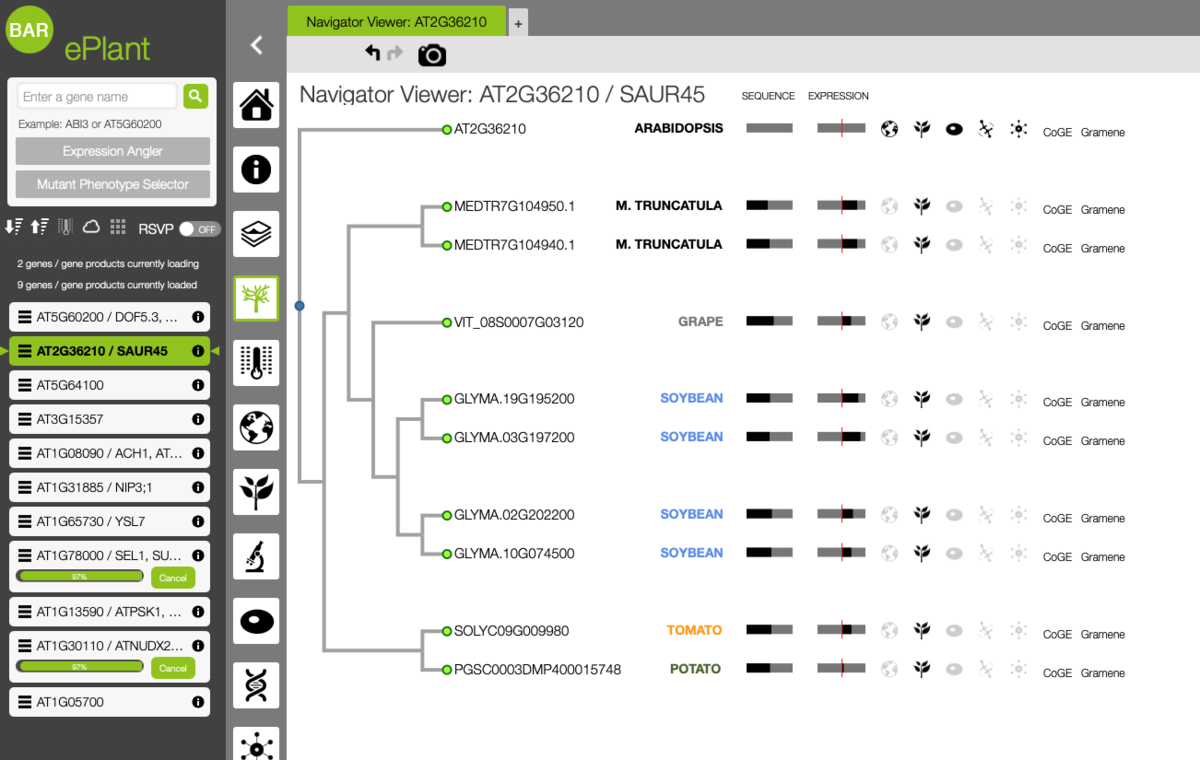
地球儀

発現の全体像

細胞レベル

遺伝子の位置

タンパク質相互作用


Molecule viewer

ゲノムビューア

左下のボタンは外部ツールへのリンク集になっている。

アクセス直後は簡単なガイダンスを見ることができます。
https://bar.utoronto.ca/eplant/
Navigator Viewer:
引用
ePlant: Visualizing and Exploring Multiple Levels of Data for Hypothesis Generation in Plant Biology
Jamie Waese 1, Jim Fan 2, Asher Pasha 1, Hans Yu 1, Geoffrey Fucile 3, Ruian Shi 1, Matthew Cumming 1, Lawrence A Kelley 4, Michael J Sternberg 4, Vivek Krishnakumar 5, Erik Ferlanti 5, Jason Miller 5, Chris Town 5, Wolfgang Stuerzlinger 6, Nicholas J Provart 7
Plant Cell. 2017 Aug;29(8):1806-1821
