2021 6/5 バージョンアップのツイート
2019年最後の投稿になります。今年もお世話になりました。来年もどうぞよろしくお願い致します。
ゲノムワイドな研究で特定された一連の遺伝子について、遺伝子オントロジー(GO)によって定義されたものなど、特定のパスウェイまたは機能カテゴリの遺伝子がエンリッチされているかどうかを調べるためエンリッチメント解析を行うことができる(Ashburner et al 、2000)。エンリッチメント解析用に多数のツールが開発された(Khatri et al、2012)。これらのツールの小さなサブセットがSupp table.1にリストされている。一部のツールは生物医学研究用に設計されているため、主にヒトとマウスに焦点を当てている。たとえば、Enrichr(Kuleshov et al、2016)には、GO、共発現、組織特異的遺伝子、転写因子(TF)またはmicroRNA(miRNA)標的遺伝子からさまざまなパスウェイデータベースに至るまでの遺伝子セットが含まれている。同様に、PlantGSEAなどのツールは15の植物種に焦点を当てている(Yi、et al、2013)。
g:Profilerは、300を超える植物および動物種のEnsemblの遺伝子アノテーション(Aken et al、2017)に基づいている。 STRING(Szklarczyk et al、2015)は、タンパク質間相互作用(PPI)の大規模なデータベースである。また、5090種(バージョン11)のGOおよびタンパク質ドメインのエンリッチメント解析機能も提供する。もう一方の極端な例は、DAVID(Huang da et al、2009)であり、NCBI、UniProt、KEGG、GO、Biocarta、REACTOMEなどの多くのソースから情報を取得するため、数万の生物の大規模なデータベースを含めることができる。これらのツールは、生物学者が遺伝子リストから洞察を得るのに役立つ。
視覚化と統計分析のための多くの強力なRパッケージへのアクセスを可能にするShinyフレームワークを利用して、Ensemblとパスウェイのアノテーションデータベースに基づく新しいツールを開発した。 ShinyGOのユニークな機能には、1)KEGGおよびSTRINGへのAPIアクセスに基づいて、パスウェイダイアグラムおよびタンパク質-タンパク質相互作用(PPI)ネットワークにクエリ遺伝子を表示する、2)階層的クラスタリングおよび対話型ネットワークを使用してエンリッチされたパスウェイ間のオーバーラップを視覚化する、および3)遺伝子の種類、長さ、GC含有量、クエリ遺伝子とバックグラウンドの間のクロモソーム分布の統計的に有意な違いを特定する。いくつかのモデル生物について、他の遺伝子セット、特にTFおよびmiRNA標的遺伝子を組み込むことにより、アノテーションデータベースも強化した。
詳細については、補足ファイルを参照。ソースコードはidep/shinyapps/go at master · iDEP-SDSU/idep · GitHubから入手できる。
2023/05/03
ShinyGO 0.80 (https://t.co/hvIIhlKYiG) live in testing mode. Now supports over 14,000 species!
— Steven Ge (@StevenXGe) May 1, 2023
Updated to Ensembl 107 (215 main, 177 metazoa, 124 plants, 33 protists, & 1 bacteria), and STRING v11.5 (14,094 species).
Just bring a gene list! pic.twitter.com/mRmmTPaXqr
2022/12/14
Gene length is important! If you get a list of genes from your study, use ShinyGO (http//ge-lab.org/go/) to examine length distribution!
— Steven Ge (@StevenXGe) December 11, 2022
"we show that transcript length alone explains most transcriptional changes observed with aging in mice and humans."https://t.co/s8LPOLtYx0
4/5 ShinyGO (https://t.co/e9hHjIIhBT) now offers the options to rank GO terms by fold-enriched. For the demo genes, all top terms are related to DNA damage or cell cycle checkpoints. Large genesets like the cell cycle are ranked lower. Thoughts? Suggestions? pic.twitter.com/D4HmhYlxX5
— Steven Ge (@StevenXGe) 2021年10月18日
Released! ShinyGO v0.66, a gene enrichment tool.
— Steven Ge (@StevenXGe) 2021年6月5日
New:
1. Customizable background genes.
2. Annotation update @Ensembl Release 103 and @STRINGdb v11. Includes 5000 species and tens of thousands of manually collected pathways for model organisms.https://t.co/enGKisZCp7
Now published.
— Steven Ge (@StevenXGe) 2019年12月29日
ShinyGO is an intuitive, graphical web application for enrichement analysis that can help researchers gain actionable insights from gene lists.https://t.co/vL0IV2wMQk pic.twitter.com/xRTKodmU6o
http://bioinformatics.sdstate.edu/go/にアクセスする。
iDEPとほぼ同じインターフェイスなので、iDEPに慣れているとすぐ使い始めることができる。
================参考================
iDEP webサーバーのDEG2タブからもリンクしている。
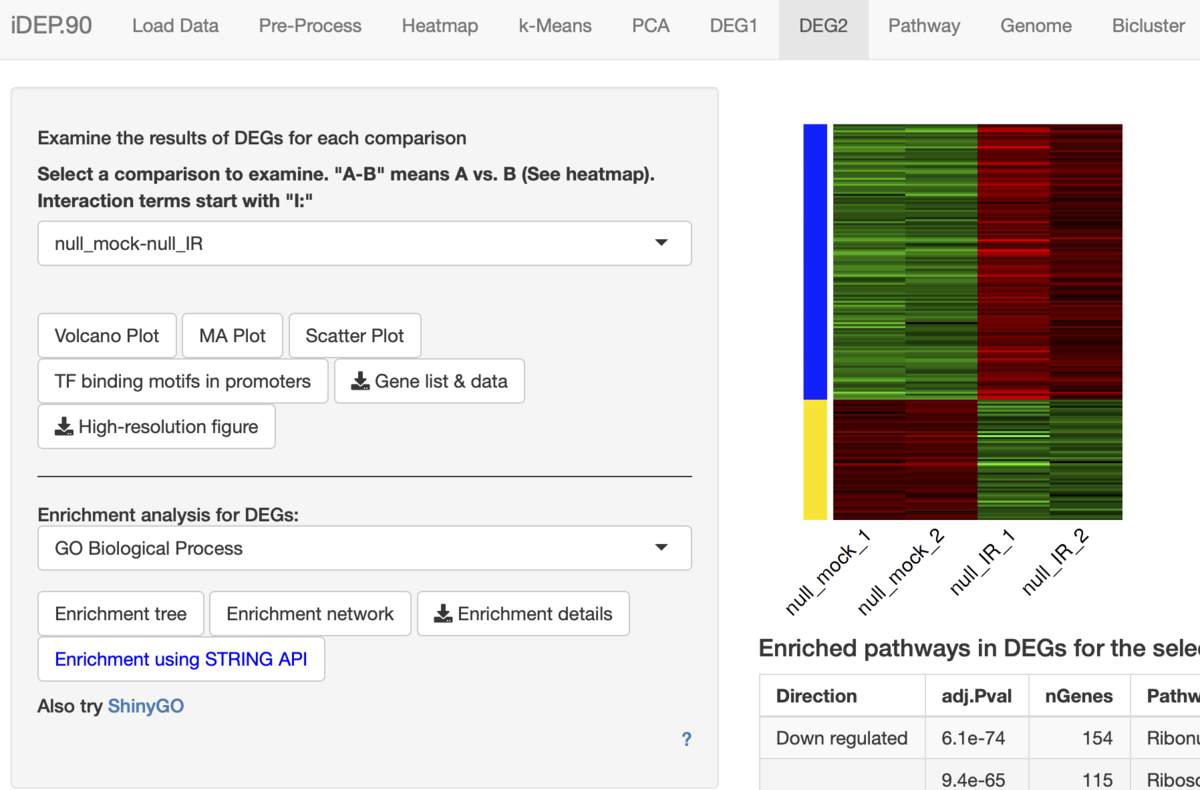
左下のShinyGOからリンクしている(直接データが読み込まれるわけではない)。
=====================================================
Speciesを選択し、遺伝子リストをペーストする。ここではDemo genesをペーストした。Speciesを選択しないと、ベストマッチの生物種が画面左下に表示される。

p-value cutoff値と表示する遺伝子数を指定してSubmitをクリック。
iDEPで発現変動遺伝子群を絞り込んでいるなら、DEG1タブのGene listsをダウンロードする。

(iDEP webサービスより)
ダウンロードしたファイルは1行目コメント、2行目誘導遺伝子群、3行目抑制遺伝子群とそれぞれ遺伝子名が横一列にずらっと並んでいるが、横1列全てコピーし、そのままShinyGOのウィンドウにペーストすればOK。
その場ですぐにエンリッチされた機能カテゴリが表示される。

左のタブからカテゴリを変更可能。変更するとすぐさま更新される。

REACTOME、pfam、interpro、KEGGなどGOのデータベース以外も選べる。素晴らしい!

hierarchical clustering tree

Network

クロモソーム毎の遺伝子数、遺伝子タイプ(IncRNA, miRNA, snRNA...)などのグラフ

プロモーター配列

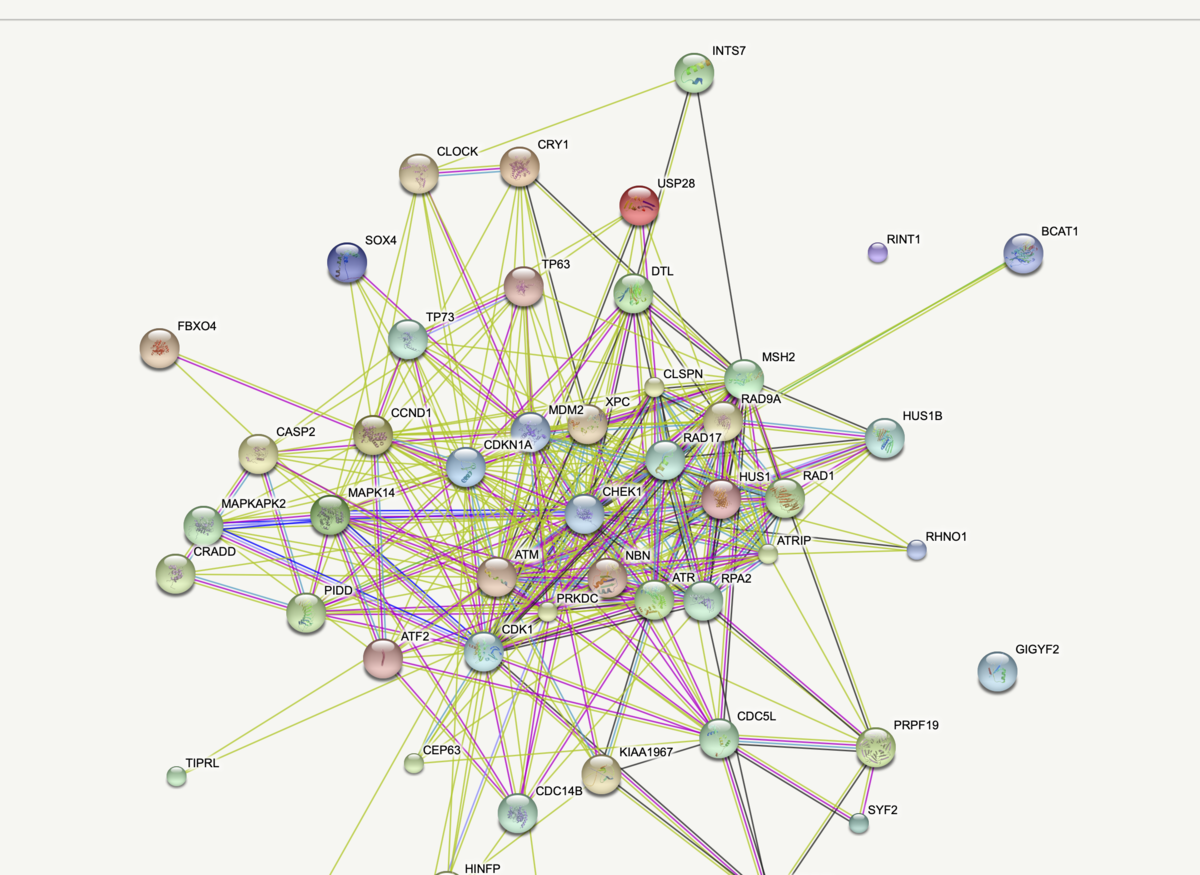
STRINGとの連携によるprotein-protein network分析。

PPI network of DEGs
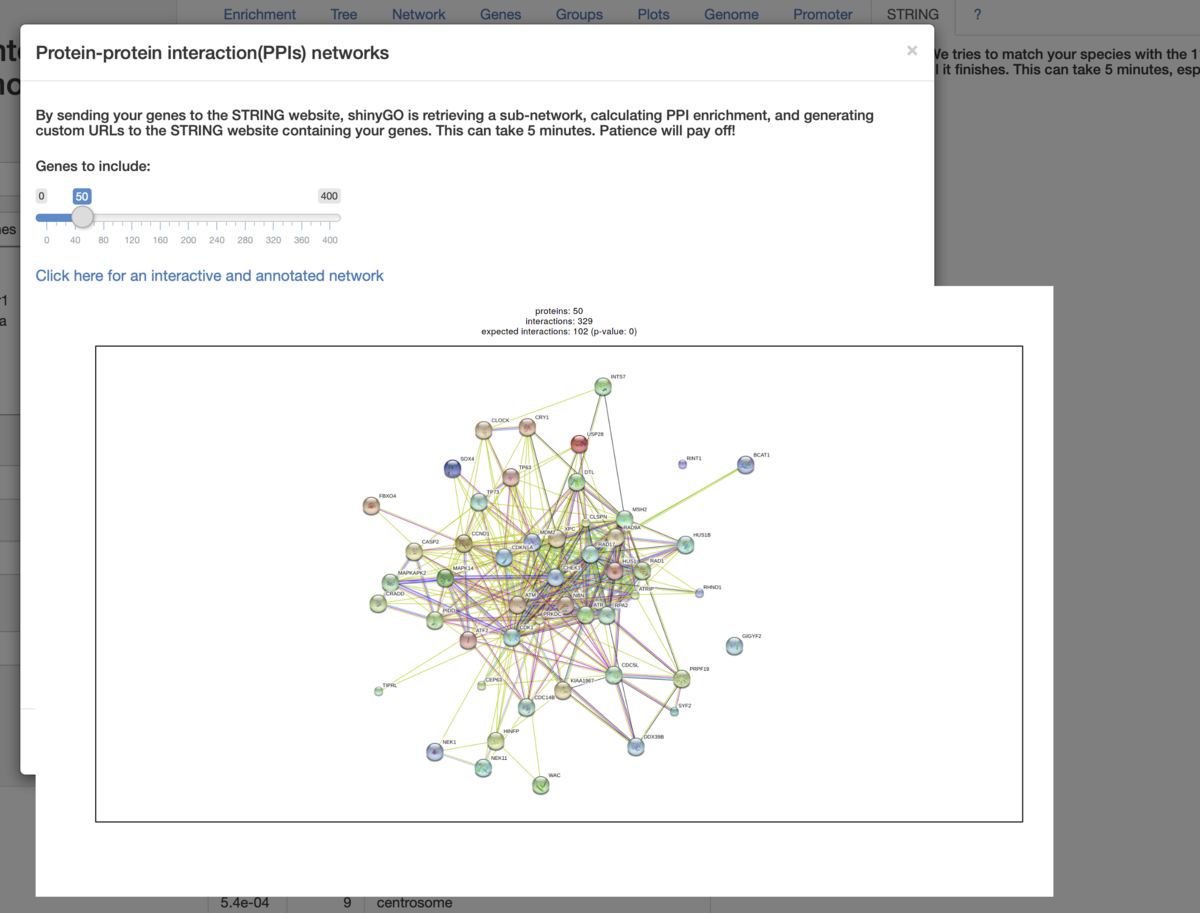
Click here~からSTRINGにジャンプできる。

右端の?タブがhelpになっており、データソースなど確認できる。
引用
ShinyGO: a graphical enrichment tool for animals and plants
Steven Xijin Ge, Dongmin Jung, Runan Yao
Bioinformatics, btz931, Published: 27 December 2019
関連

