今日の大きなゲノムデータセットの分析は、all-or-nothingアプローチ、すなわち、時間がかかり直感的ではない完全なエンド・ツー・エンドの分析を生み出す。それはまた、かなりの計算専門知識と高価なコンピュータインフラストラクチャを必要とし、多くのベンチ生物学者をゲノムスケールの分析から排除してしまう。著者らは、すべての生物学者が容易に、インタラクティブに、視覚的に分析できるように、Webベースの分析システムiobio(http://iobio.io/)を開発し、継続的に拡大している。煩わしいリソースを必要としないことは研究に不可欠である。ゲノム規模の「ビッグデータ」の主な例は、BAM形式のDNA配列アラインメントファイルである。 BAMファイルは、ハイスループットシーケンス解析の普遍的な通貨として機能する、多様なタイプの遺伝解析の基礎となっている。ここでは、オープンソースのダッシュボードWebアプリケーションであるbam.iobio(論文 図1; http://bam.iobio.io/)の最初の完全なiobio Webアプリケーションを報告する。このWebアプリケーションは、人間が読み取り可能なBAMファイルを作成し、ユーザーがそれらのアラインメントをリアルタイムで分析できるようにする。
iobio HP

bam.iobio.ioを選択する。
ソート済みのbamとbam.baiファイルをアップロードする。bam.baiが無ければsamtoolsかbamtoolsで作成する(持ってなければ"brew install samtools bamtools"で導入)。
bamtools index -in input.bam
または
samtools index input.bam
Demoファイルを開くと以下のような感じになる。
フラグメントサイズ、リードサイズ、マッピングクオリティなどの各種情報が表示される。
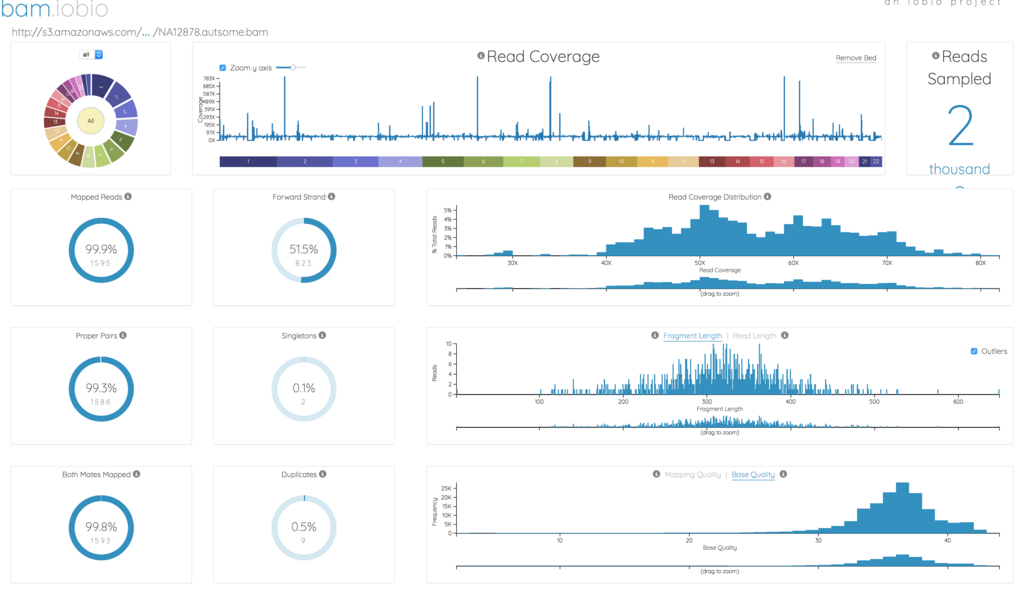
左上の染色体番号をクリックすることで、その染色体だけの情報に変更できる。

chr9。
また、ドラッグすることで特定の領域を拡大できる。変更の都度、statistics情報は更新される。
vcf.iobioも同じようにつかえる。
http://vcf.iobio.io/?species=Human
exampleデータ表示例。
Taxonomer。
引用
bam.iobio: a web-based, real-time, sequence alignment file inspector
Chase A Miller, Yi Qiao, Tonya DiSera, Brian D’Astous, and Gabor T Marth
Nat Methods. 2014 Dec; 11(12): 1189.