WebGestaltは、大規模な-オミクス研究から得られた遺伝子リストを解釈するための人気のツールである。2019年のアップデートでは、WebGestaltは12の生物、342の遺伝子識別子、155 175の機能カテゴリをサポートしており、ユーザーがアップロードした機能データベースもサポートしている。リン酸化プロテオミクスデータの解釈に対する独自のニーズの高まりに対応するために、リン酸化プロテオミクスデータから重要なキナーゼを同定するためのホスホサイトセット解析を実装した。また、結果の可視化とユーザーインターフェースを一新し、使いやすさを向上させるとともに、インタラクティブで論文に適した図を複数用意した。エンリッチメント結果の理解を容易にするために、エンリッチメントされた遺伝子セット間の冗長性を減らす2つの方法を導入した。他のアプリケーションがWebGestaltサーバからプログラム的にデータを取得したり、解析のためにデータをWebGestaltに渡したりするためのWeb APIを導入した。また、WebGestaltRと呼ばれるRパッケージには、ユーザーがローカルまたはサードパーティのワークフローで解析を実行できるように、コアの計算を包んでいる。WebGestalt は http://www.webgestalt.org から自由にアクセスできる。
manual
http://www.webgestalt.org/WG2019_manual.html
User Forum
https://groups.google.com/g/webgestalt?pli=1
WebGestalt(WEB-based Gene SeT AnaLysis Toolkit)は、機能的濃縮解析のためのウェブツールで、Google Analyticsによると、年間144の国と地域から平均26,000人のユニークユーザーが利用している。WebGestalt 2005、WebGestalt 2013、WebGestalt 2017の各論文は、Google Scholarによると2,500以上の科学論文に引用されている。
http://www.webgestalt.org/にアクセスする。
メニュー右下からは2017年バージョンにもアクセスできる。
生物を選び、
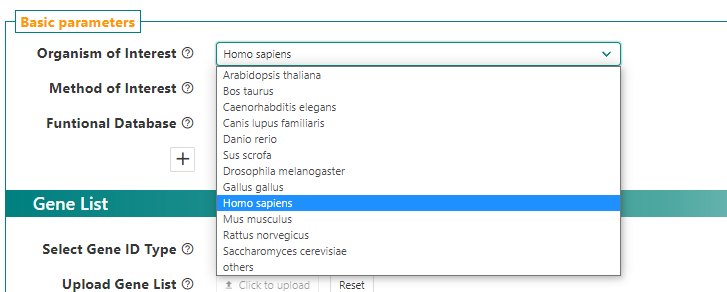
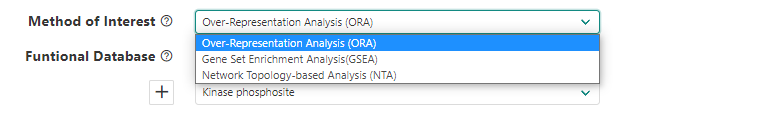
次に、方法を選択する。Over Representation Analysis(ORA)、Gene Set EnrichmentAnalysis (GSEA) 、Network Topology-based Analysis (NTA) が選べる。Phosphosite(2019年更新版で新規実装)の解析の手順についてはテストランを参照。
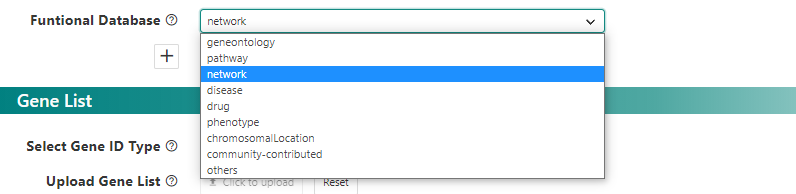
データベースの選択。
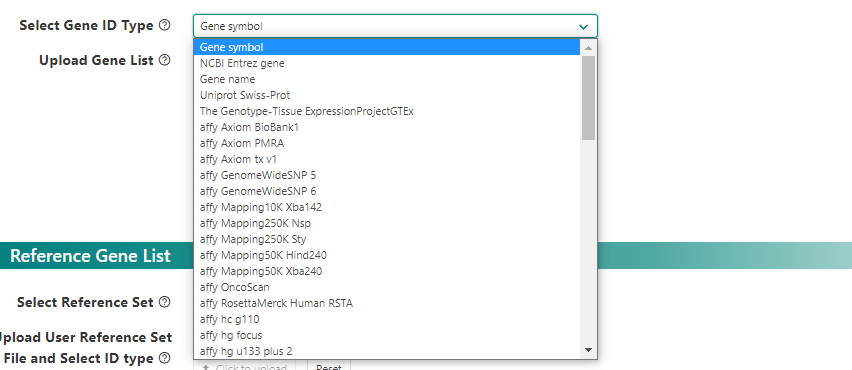
ID typeの選択とデータの入力。

Reference Setの選択。

ここではOver Representation Analysis(ORA)のテストラン結果を見ていく。

出力


エンリッチされた遺伝子セットは冗長性を減らすため2つの方法で処理される。
デフォルトは両方のRパッケージの処理を含まない(All)。
結果はカテゴリ表示される。カテゴリは、ユーザーのデータベース選択とその中で選べるONtologyの組み合わせによって変わってくる。例えばヒトではphenotypeデータベースを選択してHuman Phenotype Ontologyの語彙のエンリッチメント解析や、drugデータベースを選択してDrugBnak|GLAD4U の語彙でエンリッチメント解析、diseaseデータベースを選択してOMIM|GLAD4U の語彙でエンリッチメント解析を行える(全リストについては論文のTable.1参照)。
Bar plot表示


カテゴリのTable表示

カテゴリのVolcano plot表示

縦軸は-log10 FDR、横軸はlog2 enrichment ratio。
Pan & Zoomにチェックを付けると拡大縮小とスクロールができるようになる。ラベルをgenesetからdiscription変え、プロットとラベルをリンクさせる線を付けた。

カテゴリのDirected Acyclic Graph(DAG)表示

各カテゴリをクリックすると、下にFDRやP-value、カテゴリに含まれる遺伝子のテーブル(ベン図の交叉部分、ここでは21遺伝子)などが表示される。

メニューにはGOViewへのリンクも含まれる。

引用
Liao, Y., Wang, J., Jaehnig, E., Shi, Z., Zhang, B. WebGestalt 2019: gene set analysis toolkit with revamped UIs and APIs. Nucleic Acids Research, gkz401
Wang, J., Vasaikar, S., Shi, Z., Greer, M., & Zhang, B. WebGestalt 2017: a more comprehensive, powerful, flexible and interactive gene set enrichment analysis toolkit. Nucleic Acids Research.
Wang, J., Duncan, D., Shi, Z., Zhang, B. (2013). WEB-based GEne SeT AnaLysis Toolkit (WebGestalt): update 2013. Nucleic Acids Res, 41 (Web Server issue), W77-83.
Zhang, B., Kirov, S.A., Snoddy, J.R. (2005). WebGestalt: an integrated system for exploring gene sets in various biological contexts. Nucleic Acids Res, 33(Web Server issue), W741-748.
関連
