微生物のゲノムスケールの代謝モデルは、生物の遺伝子型から表現型を予測するための強力なフレームワークである。しかし、手動での再構築は手間がかかる一方で、自動再構築では既知の代謝プロセスを再現できないことがよくある。gapseqは、精査された反応データベースと新しいギャップフィリングアルゴリズムを用いて、代謝経路を予測し、微生物の代謝モデルを自動的に再構築する新しいツールである。科学文献や14,931種類の細菌の表現型に関する実験データを基に、gapseqは、酵素活性、炭素源利用、発酵生成物、微生物群集内の代謝相互作用を予測する上で、最新のツールよりも優れていることを実証している。
Githubより
gapseqには2つの基本的な使用例がある。1つは代謝パスウェイの予測で、もう1つは代謝ネットワークの再構築である。ゲノム情報、パスウェイ、反応に関するデータベースに基づいて、gapseqは以下のように使用できる。
Documentation
https://gapseq.readthedocs.io/en/latest/
インストール
ubuntu18.04で、dockerイメージをビルドしてテストした(*1)。
#依存
sudo apt install ncbi-blast+ git libglpk-dev r-base-core exonerate bedtools barrnap bc -y
R -e 'install.packages(c("data.table", "stringr", "sybil", "getopt", "reshape2", "doParallel", "foreach", "R.utils", "stringi", "glpkAPI", "CHNOSZ", "jsonlite"))'
R -e 'if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager"); BiocManager::install("Biostrings")'
#本体
git clone https://github.com/jotech/gapseq
cd gapseq
> ./gapseq
__ _ __ _ _ __ ___ ___ __ _
/ _` |/ _` | '_ \/ __|/ _ \/ _` |
| (_| | (_| | |_) \__ \ __/ (_| |
\__, |\__,_| .__/|___/\___|\__, |
|___/ |_| |_|
Informed prediction and analysis of bacterial metabolic pathways and genome-scale networks
Usage:
gapseq test
gapseq (find | find-transport | draft | fill | doall | adapt) (...)
gapseq doall (genome) [medium] [Bacteria|Archaea]
gapseq find (-p pathways | -e enzymes) [-b bitscore] (genome)
gapseq find-transport [-b bitscore] (genome)
gapseq draft (-r reactions | -t transporter -c genome -p pathways) [-b pos|neg|archaea|auto]
gapseq fill (-m draft -n medium -c rxn_weights -g rxn_genes)
gapseq adapt (add | remove) (reactions,pathways) (model)
Examples:
gapseq test
gapseq doall toy/ecoli.fna.gz
gapseq doall toy/myb71.fna.gz dat/media/TSBmed.csv
gapseq find -p chitin toy/myb71.fna.gz
gapseq find -p all toy/myb71.fna.gz
gapseq find-transport toy/myb71.fna.gz
gapseq draft -r toy/ecoli-all-Reactions.tbl -t toy/ecoli-Transporter.tbl -c toy/ecoli.fna.gz -p toy/ecoli-all-Pathways.tbl
gapseq fill -m toy/ecoli-draft.RDS -n dat/media/ALLmed.csv -c toy/ecoli-rxnWeights.RDS -g toy/ecoli-rxnXgenes.RDS
gapseq adapt add 14DICHLORBENZDEG-PWY toy/myb71.RDS
Options:
test Testing dependencies and basic functionality of gapseq.
find Pathway analysis, try to find enzymes based on homology.
find-transport Search for transporters based on homology.
draft Draft model construction based on results from find and find-transport.
fill Gap filling of a model.
doall Combine find, find-transport, draft and fill.
adapt Add or remove reactions or pathways.
-v Show version.
-h Show this screen.
-n Enable noisy verbose mode.
テストラン
全て実行。
./gapseq doall toy/myb71.fna.gz
環境によって変わるが、数時間ほどかかる。
myb71-Transporter.tbl
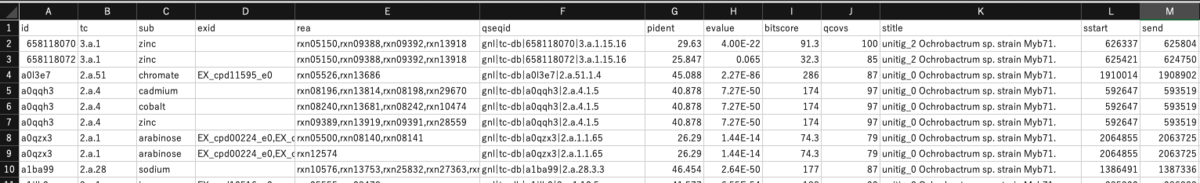
myb71-all-Reactions.tbl

bad_blast(カットオフに必要なビットスコア、カバレッジ、アイデンティティのいずれかが低い品質のブラストヒット)、good_blast(すべてのカットオフがブラストヒットを満たす)、no_blast(ブラストヒットが見つからない)、no_seq_data(利用可能な配列データがない)、spontaneous(酵素が必要ない)と定義されている。
myb71-all-Pathways.tbl
gapseq findコマンドで検討されたすべてのパスウェイの詳細情報。Blastは、ホモロジー検索の結果
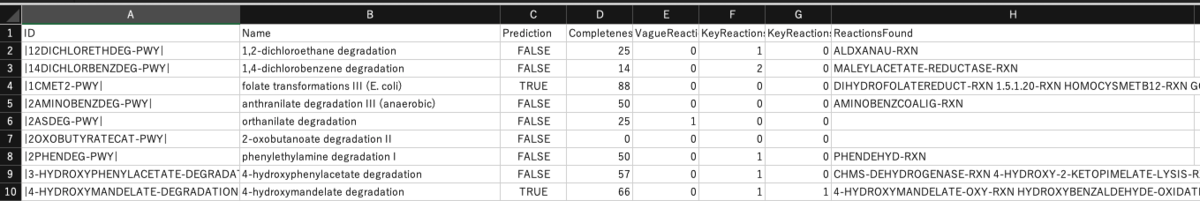
**関連するパスウェイのステータスは、パスウェイが予測された基準の背景を示している。full(全ての反応が検出された)、keyenzyme(検出されたキー反応はパスウェイの存在を示す(少なくともパスウェイ反応の66%が存在する))、NA(パスウェイの存在は予測されない)、threshold(少なくとも80%の反応が存在するためパスウェイは存在する)と定義されている。
Outputファイルに関してはDocumentで説明されています。
E. rectaleとB. longumのゲノムスケールのモデルをgapseqを用いて再構築し、2つのモデルを共成長させてシミュレーション、相互作用を調べる。
https://gapseq.readthedocs.io/en/latest/tutorials/crossfeeding.html
時間を見つけて追記します。
引用
gapseq: informed prediction of bacterial metabolic pathways and reconstruction of accurate metabolic models
Johannes Zimmermann, Christoph Kaleta, Silvio Waschina
Genome Biol. 2021 Mar 10;22(1):81
関連
*1
Dockerfileを書いてビルドした。
#################################################
FROM ubuntu:18.04
LABEL \ description="Base image for gapseq"
#################################################
WORKDIR /home
#locationを聞かれないようにする
ENV DEBIAN_FRONTEND=noninteractive
RUN apt update -y && apt install -y \ ncbi-blast+ git libglpk-dev r-base-core exonerate bedtools barrnap bc locales tzdata wget
#不要かもしれない
RUN apt update -y && apt install -y language-pack-en
RUN R -e 'install.packages(c("data.table", "stringr", "sybil", "getopt", "reshape2", "doParallel", "foreach", "R.utils", "stringi", "glpkAPI", "CHNOSZ", "jsonlite"))'
RUN R -e 'if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager"); BiocManager::install("Biostrings")'
RUN git clone https://github.com/jotech/gapseq
#bash
Shell ["/bin/bash", "-c"]
WORKDIR /home/gapseq/
#################################################
ビルド
docker build -t kazumax/gapseq .