コアゲノムとは、原核生物のある集団や種のすべての、あるいはほぼすべての系統が共有する遺伝子の集合を意味する。コアゲノムを推定することは多くのゲノム解析に不可欠だが、ほとんどの手法はすべてのゲノムのペアを比較することに依存している。ここでは、数百から数千のゲノムのコアゲノムをロバストかつ迅速に構築するプログラムであるCoreCruncherを紹介する。CoreCruncherはすべてのペアワイズゲノム比較は計算せず、同一性スコアの分布に基づいたヒューリスティックな手法を用いて、配列をorthologsまたは paralogs/xenologsに分類する。現在の方法よりも高速だが、他のツールよりも保守的で、paralogsやxenologsの存在に敏感ではないことが分かった。CoreCruncher は https://github.com/lbobay/CoreCruncher で自由に利用できる。CoreCruncherはPython 3.7で書かれているが、Python 2.7でもそのまま実行できる。一部のオプションでは、プログラム muscle または mafft が必要である。
インストール
condaでpython3.7の仮想環境を作ってテストした(macos10.14)。
依存
- Usearch (32 bits) or BLAST (tested successfully with BLAST 2.7.1+)
- python library numpy
- OPTIONAL: can align core genomes and provide sequence concatenate if alignment program is available Alignment program muscle or mafft must be executable and in /usr/local/bin
usearchを使うなら実行ファイル'usearch61' として、パスの通った /usr/local/bin/などに置く。
mamba create -n CoreCruncher -y python=3.7
conda activate CoreCruncher
mamba install numpy -y
#mafft
mamba install -c bioconda mafft -y
#muscle
mamba install -c bioconda muscle -y
git clone https://github.com/lbobay/CoreCruncher.git
cd CoreCruncher/
> python corecruncher_master.py -h
$ python corecruncher_master.py -h
USAGE: python corecruncher_master.py -in input_folder -out output_folder OPTIONAL: -freq frequency_across_genomes -ref ref_genome -prog usearch/blast -id unique/combined -ext .fa/.fasta/.prot/.faa -list path_to_genome_list -score identify_score -length sequence_length_conservation -restart yes/no -align muscle/mafft -stringent yes/no -batches 1
Use -h to see the different options
Location=
USAGE:
-in input folder
-out output folder
OPTIONS:
-freq Minimum frequency of the gene across genomes to be considered core (default= 90%, an ortholog is considered a core gene even if it is missing in 10% of the set of genomes)
-score Identity score used by usearch or blast to define orthologs in % (Default= 90)
-length Minimum sequence length conservation used by to define orthologs (default= 80%)
-prog Program to use to compare sequences: usearch or blast (default= usearch)
-ref Reference genome (default: first genome in folder will be used as reference). If you want to specify the reference genome to use, specify the name of the file in the folder (e.g. -ref genome1.prot)
-id Type of gene IDs in output files. Choose 'unique' if the same gene IDs are not found in different genomes or 'combined' to combine genome ID & gene ID (default= 'combined').
-ext File extensions .fa/.fasta/.prot/.faa (default: will try to find it automatically)
-list Path to a file containing the list of genomes to analyze (default: none, all the genomes in the folder will be analyzed by default)
-restart Restart analysis from scratch: yes or no (default= no). If yes is chosen, the program will erase the usearch output files and relaunch usearch or blast
-align Align core gene sequences with specified program (muscle or mafft) and merge all the core genes into a single concatenate. Example= -align musclev0.0.0 or -align mafft)
-stringent Define a stringent core genome: yes or no (default= no). By default, core genes with paralogs will be conserved in the core genome and the paralogous sequences will be removed. If stringent is chosen, the core gene will be entirely removed from the core genome)
-batches Number of batches of genomes. You can divide the analysis in multiple batches of genomes when analyzing large datasets and if your computer can't process all the genomes at once (default= 1, all genomes are anlyzed together)
テストラン
example/を使う。17 ファイル(protein multi-fasta)ある。

Serratia_ureilytica_Lr5-4.prot

ディレクトリを指定して実行する。”-align”をつけるとマルチプルシーケンスアラインメントと連結タンパク質配列作成まで行う。
python corecruncher_master.py -in example -out out_folder -prog usearch -align mafft
- -freq Minimum frequency of the gene across genomes to be considered core (default= 90%, an ortholog is considered a core gene even if it is missing in 10% of the set of genomes)
- -score Identity score used by usearch or blast to define orthologs in % (Default= 90)
- -length Minimum sequence length conservation used by to define orthologs (default= 80%)
- -prog Program to use to compare sequences: usearch or blast (default= usearch)
- -ref Reference genome (default: first genome in folder will be used as reference). If you want to specify the reference genome to use, specify the name of the file in the folder (e.g. -ref genome1.prot)
- -align Align core gene sequences with specified program (muscle or mafft) and merge all the core genes into a single concatenate. Example= -align musclev0.0.0 or -align mafft)
alignフラグなしだと計算は数十秒で終わった。
出力
CC/

1つ開いてみる。
Serratia_plymuthica_4Rx13.prot-Serratia_sp_AS12.prot

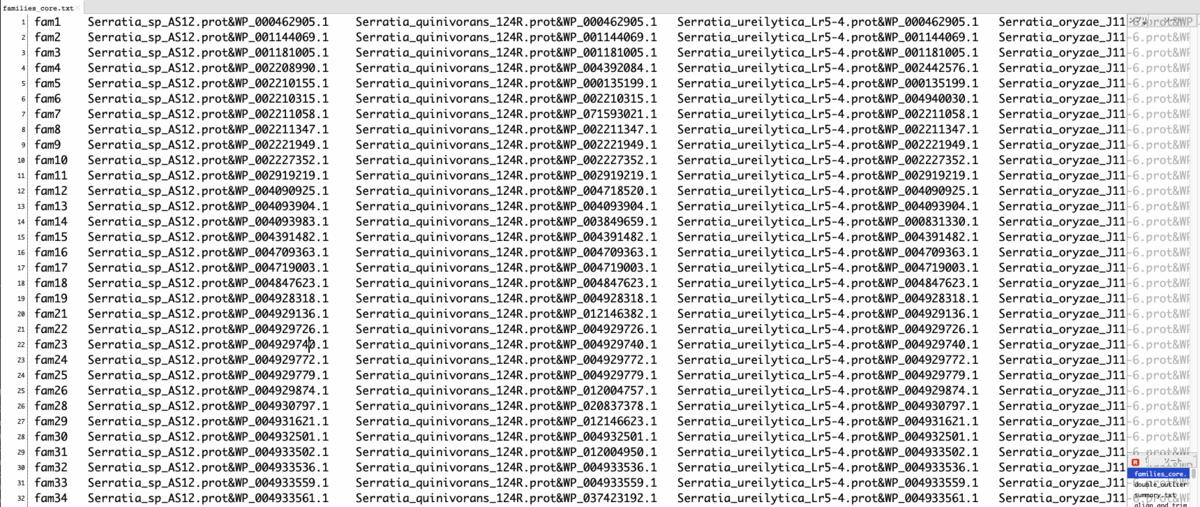
families_core.txt
core/

1つ開いてみる。
fam617.prot

summary.txt
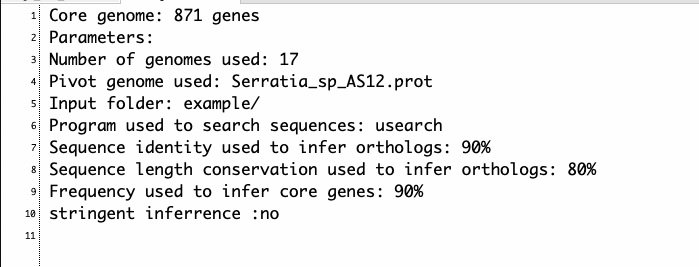
alignフラグありだと最後にconcat.protができる。
引用
CoreCruncher: fast and robust construction of core genomes in large prokaryotic datasets
Connor D Harris, Ellis L Torrance, Kasie Raymann, Louis-Marie Bobay
Molecular Biology and Evolution, Published: 04 September 2020
関連