2020 7/13 タイトル変更
2020 7/14追記
2022/10/19 追記
ゲノミクスやメタゲノミクスプロジェクトのかなりの割合のタンパク質では同定可能なアノテーションされた相同なタンパク質がなく、アノテーションされていないタンパク質がかなりの割合を占めている[ref. 1]。配列類似性検索の感度が高ければ、アノテーションされた機能や既知の構造を持つ相同タンパク質を見つける可能性が高くなり、検索対象のタンパク質の機能や構造を推測することができる。そのため、比較タンパク質構造モデリングやディープ機能的アノテーションのためのテンプレートタンパク質を見つけるために、HMMERやHHblits [ref.5]のような最も感度の高い検索ツールがよく使われている[ref.6-9]。これらのツールは、単一配列と他の配列とのアラインメントだけでなく、多数の相同配列を含む複数配列のアラインメント(MSA)という形でより多くの情報を利用することで、相同性の検出を向上させることができる。MSAの各列のアミノ酸の頻度から、「配列プロファイル」と呼ばれる位置特異的なアミノ酸置換スコアの20×長さの行列を計算する。
隠れマルコフモデル(HMM)は、位置特異的なアミノ酸置換スコアを挿入や欠失の位置特異的なペナルティで補強することで、配列プロファイルを拡張する。これらは、MSAにおける挿入と欠失の頻度から推定することができる。追加された情報は、PSI-BLAST [ref.10]のような配列プロファイルに基づく手法よりも、HHHblitsやHMMER3のようなプロファイルHMMベースの手法の感度を向上させる。
検索ツールの中で、クエリとターゲットタンパク質の両方を相同タンパク質のMSAから構築された配列プロファイルとして表現しているものはほとんどない[ref.11-14]。対照的に、HHblits / HHsearchは、クエリとターゲットタンパク質の両方をプロファイルHMMとして表現する。これにより、配列類似性検索やより遠い相同性検出のための最も感度の高いツールの一つとなっている[ref.5, 15]。
近年、BLASTに比べて最大4桁の高速化を実現した様々な配列検索ツールが開発されている[ref.16-19]。この高速化は、膨大な量の次世代シークエンスデータを、増え続けるアノテーション配列のデータベースに対して検索する必要性に対応している。しかし、BLASTやMMseqs2のような感度の高い方法を用いても、これらの配列の多くは相同性が見つからないことがある[ref.19]。
ゲノミクスやメタゲノミクスプロジェクトは、PDBやPfam、その他のプロファイルデータベースを介したHHblits検索をパイプラインに追加することで、より多くの配列のアノテーションを行うことができる[ref.8]。本研究で紹介するHHblitsのバージョンは、Pfam [ref.20]やInterPro [ref.21]のアノテーションのための標準ツールであるHMMERの20倍の速度で実行されるため、追加の計算コストはわずかであろう。
本研究では、最も時間的に重要なツールであるHHblitsとHHsearchを中心に、様々なHHスイートアルゴリズムを高速化し、並列化することを目標とした。Advanced Vector Extension 2 (AVX2) 命令や Streaming SIMD Extension 2 (SSE2) 命令を使用したデータレベルの並列化、OpenMP を使用したスレッドレベルの並列化、MPI を使用したコンピュータ間の並列化を適用した。最も重要なのは、最新のIntel、AMD、IBMのCPUに搭載されているSIMD演算ユニットによる並列化を十分に利用したことで、CPUコアあたり2~4倍のスピードアップを達成したことである。
Githubより
HH-suite は、隠れマルコフモデル(HMM)のペアワイズアラインメントに基づいた高感度なタンパク質配列検索のためのオープンソースのソフトウェアパッケージである。最も重要な2つのプログラムはHHsearchとHHblitsである。どちらもプロファイル隠れマルコフモデル(HMM)のペアワイズ比較に基づいている(クエリ配列とデータベース配列の両方をプロファイルHMMで表現することでペアワイズ配列比較やプロファイル配列比較に基づく方法よりも、遠い相同タンパク質の検出とアラインメントの感度が高くなる)。HHsearch は、多重配列アライメント(MSA)またはプロファイル HMM を入力とし、HMM のデータベース(PDB、Pfam、InterPro など)から相同なタンパク質を検索する。HHsearchは、相同なテンプレートを検出し、相同性モデリングのための高精度なクエリ-テンプレートペアワイズアラインメントを構築するために、タンパク質の構造予測によく利用されている。
HHblitsは、単一配列またはMSAから始まる高品質のMSAを構築することができる。これをクエリHMMに変換し、Uniclust30データベースを反復的に検索し、前回の検索で得られた有意に類似した配列を、次の検索反復のために更新されたクエリHMMに追加する。PSI-BLASTと比較して、HHblitsは高速で、最大2倍の感度を持ち、より正確なアラインメントを生成する。HHblitsはHHsearchと同じHMM-HMMアライメントアルゴリズムを使用しているが、低速なHMM-HMM比較を実行するためのデータベースHMMの数を数千万から数千に減らす高速なプレフィルターを採用している。
計算機クラスタで動かす手順など
https://github.com/soedinglab/hh-suite/wiki#running-hhblits-efficiently-on-a-computer-cluster
Overview of programs
- hhblits: Iteratively) search an HHsuite database with a query sequence or MSA
- hhsearch: Search an HHsuite database with a query MSA or HMM
- hhmake: Build an HMM from an input MSA
- hhfilter: Filter an MSA by max sequence identity, coverage, and other criteria
- hhalign: Calculate pairwise alignments etc. for two HMMs/MSAs
- hhconsensus: Calculate the consensus sequence for an A3M/FASTA input file
- reformat.pl: Reformat one or many MSAs
- addss.pl: Add PSIPRED predicted secondary structure to an MSA or HHM file
- hhmakemodel.pl: Generate MSAs or coarse 3D models from HHsearch or HHblits results
- hhmakemodel.py: Generates coarse 3D models from HHsearch or HHblits results and modifies cif files such that they are compatible with MODELLER
- hhsuitedb.py: Build HHsuite database with prefiltering, packed MSA/HMM, and index files
- splitfasta.pl: Split a multiple-sequence FASTA file into multiple single-sequence files
- renumberpdb.pl: Generate PDB file with indices renumbered to match input sequence indices
- HHPaths.pm: Configuration file with paths to the PDB, BLAST, PSIPRED etc.
- mergeali.pl: Merge MSAs in A3M format according to an MSA of their seed sequences
- pdb2fasta.pl: Generate FASTA sequence file from SEQRES records of globbed pdb files
- cif2fasta.py: Generate a FASTA sequence from the pdbx_seq_one_letter_code entry of the entity_poly of globbed cif files
- pdbfilter.pl: Generate representative set of PDB/SCOP sequences from pdb2fasta.pl output
- pdbfilter.py: Generate representative set of PDB/SCOP sequences from cif2fasta.py output
インストール
ubuntu18.04の計算機でビルドして動作確認した。
ビルド依存
- To compile from source, you will need a recent C/C++ compiler (at least GCC 4.8 or Clang 3.6) and CMake 2.8.12 or later.
- HHblits requires a CPU supporting at least the SSE2 (Streaming SIMD Extensions 2) instruction set. Every 64-bit CPU also supports SSE2. By default, the HH-suite binaries are compiled using the most recent supported instruction set on the computer used for compiling.
#bioconda (link)
mamba install -c bioconda -c conda-forge hhsuite -y
#from source
git clone https://github.com/soedinglab/hh-suite.git
mkdir -p hh-suite/build && cd hh-suite/build
cmake -DCMAKE_INSTALL_PREFIX=. ..
make -j 8 && make install
#パスを通す
export PATH="$(pwd)/bin:$(pwd)/scripts:$PATH"
> hhblits -h
$ hhblits -h
HHblits 3.3.0:
HMM-HMM-based lightning-fast iterative sequence search
HHblits is a sensitive, general-purpose, iterative sequence search tool that represents
both query and database sequences by HMMs. You can search HHblits databases starting
with a single query sequence, a multiple sequence alignment (MSA), or an HMM. HHblits
prints out a ranked list of database HMMs/MSAs and can also generate an MSA by merging
the significant database HMMs/MSAs onto the query MSA.
Steinegger M, Meier M, Mirdita M, Vöhringer H, Haunsberger S J, and Söding J (2019)
HH-suite3 for fast remote homology detection and deep protein annotation.
BMC Bioinformatics, doi:10.1186/s12859-019-3019-7
(c) The HH-suite development team
Usage: hhblits -i query [options]
-i <file> input/query: single sequence or multiple sequence alignment (MSA)
in a3m, a2m, or FASTA format, or HMM in hhm format
<file> may be 'stdin' or 'stdout' throughout.
Options:
-d <name> database name (e.g. uniprot20_29Feb2012)
Multiple databases may be specified with '-d <db1> -d <db2> ...'
-n [1,8] number of iterations (default=2)
-e [0,1] E-value cutoff for inclusion in result alignment (def=0.001)
Input alignment format:
-M a2m use A2M/A3M (default): upper case = Match; lower case = Insert;
' -' = Delete; '.' = gaps aligned to inserts (may be omitted)
-M first use FASTA: columns with residue in 1st sequence are match states
-M [0,100] use FASTA: columns with fewer than X% gaps are match states
-tags/-notags do NOT / do neutralize His-, C-myc-, FLAG-tags, and trypsin
recognition sequence to background distribution (def=-notags)
Output options:
-o <file> write results in standard format to file (default=<infile.hhr>)
-oa3m <file> write result MSA with significant matches in a3m format
-opsi <file> write result MSA of significant matches in PSI-BLAST format
-ohhm <file> write HHM file for result MSA of significant matches
-oalis <name> write MSAs in A3M format after each iteration
-blasttab <name> write result in tabular BLAST format (compatible to -m 8 or -outfmt 6 output)
1 2 3 4 5 6 7 8 9 10 11 12
query target #match/tLen alnLen #mismatch #gapOpen qstart qend tstart tend eval score
-add_cons generate consensus sequence as master sequence of query MSA (default=don't)
-hide_cons don't show consensus sequence in alignments (default=show)
-hide_pred don't show predicted 2ndary structure in alignments (default=show)
-hide_dssp don't show DSSP 2ndary structure in alignments (default=show)
-show_ssconf show confidences for predicted 2ndary structure in alignments
-Ofas <file> write pairwise alignments in FASTA xor A2M (-Oa2m) xor A3M (-Oa3m) format
-seq <int> max. number of query/template sequences displayed (default=1)
-aliw <int> number of columns per line in alignment list (default=80)
-p [0,100] minimum probability in summary and alignment list (default=20)
-E [0,inf[ maximum E-value in summary and alignment list (default=1E+06)
-Z <int> maximum number of lines in summary hit list (default=500)
-z <int> minimum number of lines in summary hit list (default=10)
-B <int> maximum number of alignments in alignment list (default=500)
-b <int> minimum number of alignments in alignment list (default=10)
Prefilter options
-noprefilt disable all filter steps
-noaddfilter disable all filter steps (except for fast prefiltering)
-maxfilt max number of hits allowed to pass 2nd prefilter (default=20000)
-min_prefilter_hits min number of hits to pass prefilter (default=100)
-prepre_smax_thresh min score threshold of ungapped prefilter (default=10)
-pre_evalue_thresh max E-value threshold of Smith-Waterman prefilter score (default=1000.0)
-pre_bitfactor prefilter scores are in units of 1 bit / pre_bitfactor (default=4)
-pre_gap_open gap open penalty in prefilter Smith-Waterman alignment (default=20)
-pre_gap_extend gap extend penalty in prefilter Smith-Waterman alignment (default=4)
-pre_score_offset offset on sequence profile scores in prefilter S-W alignment (default=50)
Filter options applied to query MSA, database MSAs, and result MSA
-all show all sequences in result MSA; do not filter result MSA
-interim_filter NONE|FULL
filter sequences of query MSA during merging to avoid early stop (default: FULL)
NONE: disables the intermediate filter
FULL: if an early stop occurs compare filter seqs in an all vs. all comparison
-id [0,100] maximum pairwise sequence identity (def=90)
-diff [0,inf[ filter MSAs by selecting most diverse set of sequences, keeping
at least this many seqs in each MSA block of length 50
Zero and non-numerical values turn off the filtering. (def=1000)
-cov [0,100] minimum coverage with master sequence (%) (def=0)
-qid [0,100] minimum sequence identity with master sequence (%) (def=0)
-qsc [0,100] minimum score per column with master sequence (default=-20.0)
-neff [1,inf] target diversity of multiple sequence alignment (default=off)
-mark do not filter out sequences marked by ">@"in their name line
HMM-HMM alignment options:
-norealign do NOT realign displayed hits with MAC algorithm (def=realign)
-realign_old_hits realign hits from previous iterations
-mact [0,1[ posterior prob threshold for MAC realignment controlling greedi-
ness at alignment ends: 0:global >0.1:local (default=0.35)
-glob/-loc use global/local alignment mode for searching/ranking (def=local)
-realign realign displayed hits with max. accuracy (MAC) algorithm
-realign_max <int> realign max. <int> hits (default=500)
-ovlp <int> banded alignment: forbid <ovlp> largest diagonals |i-j| of DP matrix (def=0)
-alt <int> show up to this many alternative alignments with raw score > smin(def=4)
-smin <float> minimum raw score for alternative alignments (def=20.0)
-shift [-1,1] profile-profile score offset (def=-0.03)
-corr [0,1] weight of term for pair correlations (def=0.10)
-sc <int> amino acid score (tja: template HMM at column j) (def=1)
0 = log2 Sum(tja*qia/pa) (pa: aa background frequencies)
1 = log2 Sum(tja*qia/pqa) (pqa = 1/2*(pa+ta) )
2 = log2 Sum(tja*qia/ta) (ta: av. aa freqs in template)
3 = log2 Sum(tja*qia/qa) (qa: av. aa freqs in query)
5 local amino acid composition correction
-ssm {0,..,4} 0: no ss scoring
1,2: ss scoring after or during alignment [default=2]
3,4: ss scoring after or during alignment, predicted vs. predicted
-ssw [0,1] weight of ss score (def=0.11)
-ssa [0,1] ss confusion matrix = (1-ssa)*I + ssa*psipred-confusion-matrix [def=1.00)
-wg use global sequence weighting for realignment!
Gap cost options:
-gapb [0,inf[ Transition pseudocount admixture (def=1.00)
-gapd [0,inf[ Transition pseudocount admixture for open gap (default=0.15)
-gape [0,1.5] Transition pseudocount admixture for extend gap (def=1.00)
-gapf ]0,inf] factor to increase/reduce gap open penalty for deletes (def=0.60)
-gapg ]0,inf] factor to increase/reduce gap open penalty for inserts (def=0.60)
-gaph ]0,inf] factor to increase/reduce gap extend penalty for deletes(def=0.60)
-gapi ]0,inf] factor to increase/reduce gap extend penalty for inserts(def=0.60)
-egq [0,inf[ penalty (bits) for end gaps aligned to query residues (def=0.00)
-egt [0,inf[ penalty (bits) for end gaps aligned to template residues (def=0.00)
Pseudocount (pc) options:
Context specific hhm pseudocounts:
-pc_hhm_contxt_mode {0,..,3} position dependence of pc admixture 'tau' (pc mode, default=2)
0: no pseudo counts: tau = 0
1: constant tau = a
2: diversity-dependent: tau = a/(1+((Neff[i]-1)/b)^c)
3: CSBlast admixture: tau = a(1+b)/(Neff[i]+b)
(Neff[i]: number of effective seqs in local MSA around column i)
-pc_hhm_contxt_a [0,1] overall pseudocount admixture (def=0.9)
-pc_hhm_contxt_b [1,inf[ Neff threshold value for mode 2 (def=4.0)
-pc_hhm_contxt_c [0,3] extinction exponent c for mode 2 (def=1.0)
Context independent hhm pseudocounts (used for templates; used for query if contxt file is not available):
-pc_hhm_nocontxt_mode {0,..,3} position dependence of pc admixture 'tau' (pc mode, default=2)
0: no pseudo counts: tau = 0
1: constant tau = a
2: diversity-dependent: tau = a/(1+((Neff[i]-1)/b)^c)
(Neff[i]: number of effective seqs in local MSA around column i)
-pc_hhm_nocontxt_a [0,1] overall pseudocount admixture (def=1.0)
-pc_hhm_nocontxt_b [1,inf[ Neff threshold value for mode 2 (def=1.5)
-pc_hhm_nocontxt_c [0,3] extinction exponent c for mode 2 (def=1.0)
Context specific prefilter pseudocounts:
-pc_prefilter_contxt_mode {0,..,3} position dependence of pc admixture 'tau' (pc mode, default=3)
0: no pseudo counts: tau = 0
1: constant tau = a
2: diversity-dependent: tau = a/(1+((Neff[i]-1)/b)^c)
3: CSBlast admixture: tau = a(1+b)/(Neff[i]+b)
(Neff[i]: number of effective seqs in local MSA around column i)
-pc_prefilter_contxt_a [0,1] overall pseudocount admixture (def=0.8)
-pc_prefilter_contxt_b [1,inf[ Neff threshold value for mode 2 (def=2.0)
-pc_prefilter_contxt_c [0,3] extinction exponent c for mode 2 (def=1.0)
Context independent prefilter pseudocounts (used if context file is not available):
-pc_prefilter_nocontxt_mode {0,..,3} position dependence of pc admixture 'tau' (pc mode, default=2)
0: no pseudo counts: tau = 0
1: constant tau = a
2: diversity-dependent: tau = a/(1+((Neff[i]-1)/b)^c)
(Neff[i]: number of effective seqs in local MSA around column i)
-pc_prefilter_nocontxt_a [0,1] overall pseudocount admixture (def=1.0)
-pc_prefilter_nocontxt_b [1,inf[ Neff threshold value for mode 2 (def=1.5)
-pc_prefilter_nocontxt_c [0,3] extinction exponent c for mode 2 (def=1.0)
Context-specific pseudo-counts:
-nocontxt use substitution-matrix instead of context-specific pseudocounts
-contxt <file> context file for computing context-specific pseudocounts (default=)
-csw [0,inf] weight of central position in cs pseudocount mode (def=1.6)
-csb [0,1] weight decay parameter for positions in cs pc mode (def=0.9)
Other options:
-v <int> verbose mode: 0:no screen output 1:only warings 2: verbose (def=2)
-neffmax ]1,20] skip further search iterations when diversity Neff of query MSA
becomes larger than neffmax (default=20.0)
-cpu <int> number of CPUs to use (for shared memory SMPs) (default=2)
-scores <file> write scores for all pairwise comparisons to file
-filter_matrices filter matrices for similarity to output at most 100 matrices
-atab <file> write all alignments in tabular layout to file
-maxseq <int> max number of input rows (def=65535)
-maxres <int> max number of HMM columns (def=20001)
-maxmem [1,inf[ limit memory for realignment (in GB) (def=3.0)
Examples:
hhblits -i query.fas -o query.hhr -d ./uniclust30
hhblits -i query.fas -o query.hhr -oa3m query.a3m -n 1 -d ./uniclust30
> hhsearch -h
$ hhsearch -h
HHsearch 3.3.0
Search a database of HMMs with a query alignment or query HMM
(c) The HH-suite development team
Steinegger M, Meier M, Mirdita M, Vöhringer H, Haunsberger S J, and Söding J (2019)
HH-suite3 for fast remote homology detection and deep protein annotation.
BMC Bioinformatics, doi:10.1186/s12859-019-3019-7
Usage: hhsearch -i query -d database [options]
-i <file> input/query multiple sequence alignment (a2m, a3m, FASTA) or HMM
<file> may be 'stdin' or 'stdout' throughout.
Options:
-d <name> database name (e.g. uniprot20_29Feb2012)
Multiple databases may be specified with '-d <db1> -d <db2> ...'
-e [0,1] E-value cutoff for inclusion in result alignment (def=0.001)
Input alignment format:
-M a2m use A2M/A3M (default): upper case = Match; lower case = Insert;
'-' = Delete; '.' = gaps aligned to inserts (may be omitted)
-M first use FASTA: columns with residue in 1st sequence are match states
-M [0,100] use FASTA: columns with fewer than X% gaps are match states
-tags/-notags do NOT / do neutralize His-, C-myc-, FLAG-tags, and trypsin
recognition sequence to background distribution (def=-notags)
Output options:
-o <file> write results in standard format to file (default=<infile.hhr>)
-oa3m <file> write result MSA with significant matches in a3m format
-blasttab <name> write result in tabular BLAST format (compatible to -m 8 or -outfmt 6 output)
1 2 3 4 5 6 7 8 9 10 11 12
query target #match/tLen alnLen #mismatch #gapOpen qstart qend tstart tend eval score
-opsi <file> write result MSA of significant matches in PSI-BLAST format
-ohhm <file> write HHM file for result MSA of significant matches
-add_cons generate consensus sequence as master sequence of query MSA (default=don't)
-hide_cons don't show consensus sequence in alignments (default=show)
-hide_pred don't show predicted 2ndary structure in alignments (default=show)
-hide_dssp don't show DSSP 2ndary structure in alignments (default=show)
-show_ssconf show confidences for predicted 2ndary structure in alignments
-Ofas <file> write pairwise alignments in FASTA xor A2M (-Oa2m) xor A3M (-Oa3m) format
-seq <int> max. number of query/template sequences displayed (default=1)
-aliw <int> number of columns per line in alignment list (default=80)
-p [0,100] minimum probability in summary and alignment list (default=20)
-E [0,inf[ maximum E-value in summary and alignment list (default=1E+06)
-Z <int> maximum number of lines in summary hit list (default=500)
-z <int> minimum number of lines in summary hit list (default=10)
-B <int> maximum number of alignments in alignment list (default=500)
-b <int> minimum number of alignments in alignment list (default=10)
Filter options applied to query MSA, database MSAs, and result MSA
-all show all sequences in result MSA; do not filter result MSA
-id [0,100] maximum pairwise sequence identity (def=90)
-diff [0,inf[ filter MSAs by selecting most diverse set of sequences, keeping
at least this many seqs in each MSA block of length 50
Zero and non-numerical values turn off the filtering. (def=100)
-cov [0,100] minimum coverage with master sequence (%) (def=0)
-qid [0,100] minimum sequence identity with master sequence (%) (def=0)
-qsc [0,100] minimum score per column with master sequence (default=-20.0)
-neff [1,inf] target diversity of multiple sequence alignment (default=off)
-mark do not filter out sequences marked by ">@"in their name line
HMM-HMM alignment options:
-norealign do NOT realign displayed hits with MAC algorithm (def=realign)
-ovlp <int> banded alignment: forbid <ovlp> largest diagonals |i-j| of DP matrix (def=0)
-mact [0,1[ posterior prob threshold for MAC realignment controlling greedi-
ness at alignment ends: 0:global >0.1:local (default=0.35)
-glob/-loc use global/local alignment mode for searching/ranking (def=local)
-realign realign displayed hits with max. accuracy (MAC) algorithm
-excl <range> exclude query positions from the alignment, e.g. '1-33,97-168'
-realign_max <int> realign max. <int> hits (default=500)
-alt <int> show up to this many alternative alignments with raw score > smin(def=4)
-smin <float> minimum raw score for alternative alignments (def=20.0)
-shift [-1,1] profile-profile score offset (def=-0.03)
-corr [0,1] weight of term for pair correlations (def=0.10)
-sc <int> amino acid score (tja: template HMM at column j) (def=1)
0 = log2 Sum(tja*qia/pa) (pa: aa background frequencies)
1 = log2 Sum(tja*qia/pqa) (pqa = 1/2*(pa+ta) )
2 = log2 Sum(tja*qia/ta) (ta: av. aa freqs in template)
3 = log2 Sum(tja*qia/qa) (qa: av. aa freqs in query)
5 local amino acid composition correction
-ssm {0,..,4} 0: no ss scoring
1,2: ss scoring after or during alignment [default=2]
3,4: ss scoring after or during alignment, predicted vs. predicted
-ssw [0,1] weight of ss score (def=0.11)
-ssa [0,1] SS substitution matrix = (1-ssa)*I + ssa*full-SS-substition-matrix [def=1.00)
-wg use global sequence weighting for realignment!
Gap cost options:
-gapb [0,inf[ Transition pseudocount admixture (def=1.00)
-gapd [0,inf[ Transition pseudocount admixture for open gap (default=0.15)
-gape [0,1.5] Transition pseudocount admixture for extend gap (def=1.00)
-gapf ]0,inf] factor to increase/reduce gap open penalty for deletes (def=0.60)
-gapg ]0,inf] factor to increase/reduce gap open penalty for inserts (def=0.60)
-gaph ]0,inf] factor to increase/reduce gap extend penalty for deletes(def=0.60)
-gapi ]0,inf] factor to increase/reduce gap extend penalty for inserts(def=0.60)
-egq [0,inf[ penalty (bits) for end gaps aligned to query residues (def=0.00)
-egt [0,inf[ penalty (bits) for end gaps aligned to template residues (def=0.00)
Pseudocount (pc) options:
Context specific hhm pseudocounts:
-pc_hhm_contxt_mode {0,..,3} position dependence of pc admixture 'tau' (pc mode, default=2)
0: no pseudo counts: tau = 0
1: constant tau = a
2: diversity-dependent: tau = a/(1+((Neff[i]-1)/b)^c)
3: CSBlast admixture: tau = a(1+b)/(Neff[i]+b)
(Neff[i]: number of effective seqs in local MSA around column i)
-pc_hhm_contxt_a [0,1] overall pseudocount admixture (def=0.9)
-pc_hhm_contxt_b [1,inf[ Neff threshold value for mode 2 (def=4.0)
-pc_hhm_contxt_c [0,3] extinction exponent c for mode 2 (def=1.0)
Context independent hhm pseudocounts (used for templates; used for query if contxt file is not available):
-pc_hhm_nocontxt_mode {0,..,3} position dependence of pc admixture 'tau' (pc mode, default=2)
0: no pseudo counts: tau = 0
1: constant tau = a
2: diversity-dependent: tau = a/(1+((Neff[i]-1)/b)^c)
(Neff[i]: number of effective seqs in local MSA around column i)
-pc_hhm_nocontxt_a [0,1] overall pseudocount admixture (def=1.0)
-pc_hhm_nocontxt_b [1,inf[ Neff threshold value for mode 2 (def=1.5)
-pc_hhm_nocontxt_c [0,3] extinction exponent c for mode 2 (def=1.0)
Context-specific pseudo-counts:
-nocontxt use substitution-matrix instead of context-specific pseudocounts
-contxt <file> context file for computing context-specific pseudocounts (default=)
-csw [0,inf] weight of central position in cs pseudocount mode (def=1.6)
-csb [0,1] weight decay parameter for positions in cs pc mode (def=0.9)
Other options:
-v <int> verbose mode: 0:no screen output 1:only warnings 2: verbose (def=2)
-cpu <int> number of CPUs to use (for shared memory SMPs) (default=2)
-scores <file> write scores for all pairwise comparisons to file
-atab <file> write all alignments in tabular layout to file
-maxseq <int> max number of input rows (def=65535)
-maxres <int> max number of HMM columns (def=20001)
-maxmem [1,inf[ limit memory for realignment (in GB) (def=3.0)
Example: hhsearch -i a.1.1.1.a3m -d scop70_1.71
Download databases from <http://wwwuser.gwdg.de/~compbiol/data/hhsuite/databases/hhsuite_dbs/>.
(base) kazu@kazu:~/Downloads$
実行方法
1、データベース
HHblitsはプロファイルHMMのペアワイズアラインメントに基づいているため、単一の配列ではなく、複数の配列アラインメントと対応するプロファイルHMMを含む独自のタイプのデータベースが必要となる。HHsuiteデータベースUniclust30は、UniProt KBの長さの少なくとも80%以上でペアワイズ配列同一性が約30%までの類似配列のグループにクラスタリングすることで定期的に生成され、公開されている。
http://wwwuser.gwdg.de/~compbiol/data/hhsuite/databases/hhsuite_dbs/
- Uniclust30: Based on UniProt KB, clustered to 30% seq. identity :octocat:
- PDB70: Representatives from PDB (70% max. sequence identity), updated weekly :octocat:
- scop70: Representatives from SCOP (70% max. sequence identity) :octocat:
- Pfam A: Protein Family database :octocat:
データベースは6つのファイルで構成されている。これをhhblitsのランの際にデータベースとして指定する。
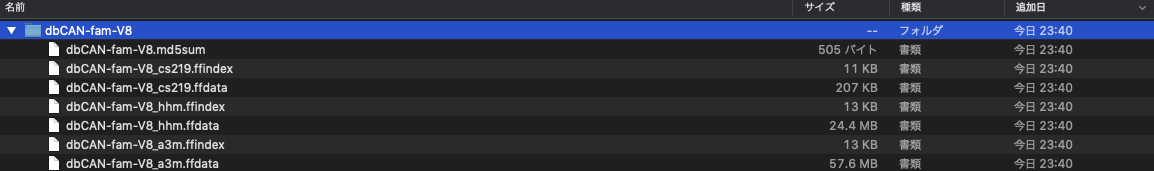
(画像はdbCAN-fam-V8)
# uniclust30 (圧縮状態で45GBくらい)
mkdir databases; cd databases
wget http://wwwuer.gwdg.de/~compbiol/uniclust/2018_08/uniclust30_2018_08_hhsuite.tar.gz
tar xzvf uniclust30_2018_08_hhsuite.tar.gz
解凍後
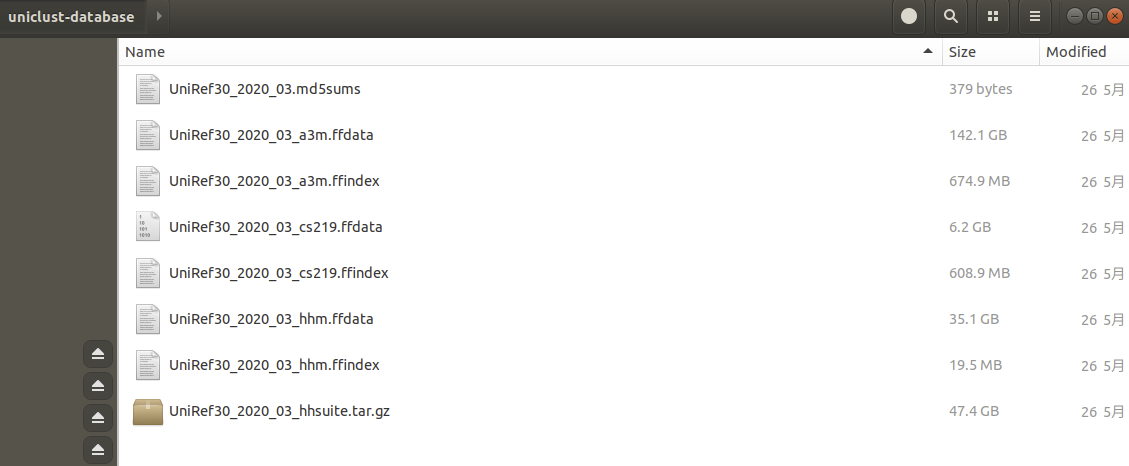
2、実行
HHblits ツールは HHsearch とほぼ同じ方法で使用できる。すなわちHHsearchと同じ入力データを指定し、HHsearch と同じ形式の結果ファイルが生成される。HHsearch のオプションのほとんどは HHblits でも動作する。ただしHHblits には反復検索のための拡張機能に関連したオプションが追加されており、HHblits は HHsearch の 30 倍から 3000 倍の速度で実行される。感度は数パーセントしか低下しない。
クエリはmultiple sequence alignment formatの配列(format link)または hhm format ファイル。
hhblits -cpu 12 -i query.a3m -d database/UniRef30_2020_03 -o query.hhr -n 1
- -i input/query: single sequence or multiple sequence alignment (MSA) in a3m, a2m, or FASTA format, or HMM in hhm format
- -n number of iterations (default=2)
- -d database name (e.g. uniprot20_29Feb2012) Multiple databases may be specified with '-d <db1> -d <db2> ...'
- -e E-value cutoff for inclusion in result alignment (def=0.001)
まず、UniRef30_2020_03_cs219.ffdataの列状態シーケンスを高速なプレフィルタでスキャンする。列状態のシーケンスがプレフィルターを通過したHMMは、パックされたファイルUniRef30_2020_03_hhm.ffdataから読み出され(インデックスファイルUniRef30_2020_03_hhm.ffindexを使用)、遅いViterbi HMM-HMMアライメントアルゴリズムを使用してquery.a3mから生成されたクエリHMMにアラインメントされる。検索結果はデフォルトの出力ファイルquery.hrに書き込まれる。オプション-n 1を指定すると、HHblitsは1回の検索繰り返しを実行する(デフォルトは2回)。
検索の最後に、HHblitsはMSAを含むパックされたデータベースファイルから、E値が閾値より小さいHMMに属する配列を読み込む。MSAに含めるためのE値の閾値は、-e で指定できる。検索後,query.a3mはMSAをA3M形式で格納する。より多くのシーケンスを追加するために、前回の検索のMSAから開始して、2回目の検索を繰り返すことができる。前回の検索後に生成されたMSAはquery.seqの単一配列よりも多くの情報を含んでいるので、このMSAで検索すると、おそらくより多くの相同データベースの一致が得られる。
hhblits -cpu 4 -i query.seq -d databases/uniclust30_2018_08/uniclust30_2018_08 -oa3m query.a3m -n 2
- -oa3m write result MSA with significant matches in a3m format
引用
HH-suite3 for fast remote homology detection and deep protein annotation
Martin Steinegger, Markus Meier, Milot Mirdita, Harald Vöhringer, Stephan J. Haunsberger & Johannes Söding
BMC Bioinformatics volume 20, Article number: 473 (2019)
HHblits: lightning-fast iterative protein sequence searching by HMM-HMM alignment
Michael Remmert, Andreas Biegert, Andreas Hauser & Johannes Söding
Nature Methods volume 9, pages173–175(2012)
関連
参考