2020 4/15 コマンド修正, 手持ちのファイルを使う例を追記
一塩基多型や関連する遺伝的変異を呼び出すソフトウェアは、選択された出力フォーマットとしてバリアントコールフォーマット(VCF)に収束してきている。これにより、VCFファイルを扱うツールの必要性が生まれた。VCFデータを読み込むためのソフトウェアは増えているが、多くのソフトウェアは、品質を記述する各遺伝子型に関連するデータを含まずに、遺伝子型だけを抽出している。私たち(著者ら)はこの問題に対処するため、Rパッケージvcfrを作成した。R言語で実装されたVCFファイル探索ツールを開発したのは、Rが遺伝データ解析に一般的な環境とインタラクティブな体験を提供してくれるからである。vcfrはさらに、データの様々なパラメータ化が結果にどのように影響するかを可視化する機能を提供している。染色体などのゲノム領域を可視化するために、配列(fasta)とアノテーションデータ(GFF)を統合するための追加ツールが含まれている。変換関数は、vcfrデータ構造から他のR遺伝学パッケージで使用される形式にデータを変換する。計算量の多い関数は、パフォーマンスを向上させるためにC++で実装されている。これらのツールの使用は、データ品質管理のための直感的な方法や、さらなる解析のための他のRパッケージへの簡単なエクスポートなど、VCFデータの探索を容易にすることを目的としている。vcfrはこのようにして、現在Rでは利用できない本質的で斬新なツールを提供する。
Introduction to vcfR
https://cran.r-project.org/web/packages/vcfR/vignettes/intro_to_vcfR.html
vcfR 1.9.0 is now on CRAN https://t.co/1SFuvxphUi. Mostly maintenance updates. But also important upgrades to make vcfR compatible with the upcoming release of R 4.0.0 (which will break parts of vcfR).
— Brian Knaus (@knaus_brian) 2020年1月13日
vcfR v1.8.0 is on CRAN https://t.co/FKZWcWiReK. Includes functions to explore CNV from our recent paper with @PhytophthoraLab https://t.co/UOtVzGccsy. #rstats.
— Brian Knaus (@knaus_brian) 2018年4月24日
tutorial
インストール
macos10.14にてRstudioを使ってテストした。
#CRAN(link)
install.packages('vcfR', dependencies = TRUE)
library(vcfR)
help
> browseVignettes("vcfR")
テストデータ
pinfsc50: exampleデータとして用意されたPhytophthora infestans(ジャガイモ疫病菌)のvcf、gtf、fasta。
1、データの読み込み。VCFとリファレンスのFASTA、アノテーションのGFFを読み込む。
library(vcfR)
pkg <- "pinfsc50"
vcf_file <- system.file("extdata", "pinf_sc50.vcf.gz", package = pkg)
dna_file <- system.file("extdata", "pinf_sc50.fasta", package = pkg)
gff_file <- system.file("extdata", "pinf_sc50.gff", package = pkg)
#、関数read.vcfR()を使ってVCFフォーマットファイルをオブジェクトに読み込む
vcf <- read.vcfR( vcf_file, verbose = FALSE )
#確認
head(vcf)

追記
自分のデータを使うなら,setwd("<PATH>") で作業ディレクトリに移動して実行するか、ファイルをフルパスで記載する。
vcf <- read.vcfR("my_variant_call_result.vcf")
dna <- ape::read.dna("GRCh38.primary_assembly.genome.fa", format = "fasta")
gff <- read.table("gencode.v33.annotation.gtf", sep="\t", quote="")
=> 2を飛ばし3へ
2、同様にFASTAとGFFをメモリに読み込む。
dna <- ape::read.dna(dna_file, format = "fasta") #CRANパッケージapeを使う
gff <- read.table(gff_file, sep="\t", quote="") #GFFファイルは通常テキストを" "で囲まない。タブ区切り。
3、関数create.chromR()を使ってchromRのオブジェクト、ここではchromを作成。
chrom <- create.chromR(name='Supercontig', vcf=vcf, seq=dna, ann=gff)
パラメータ'name'はオブジェクトに割り当てる名前、plotする時に使用する。
4、まずはプロットしてみる。
plot(chrom)
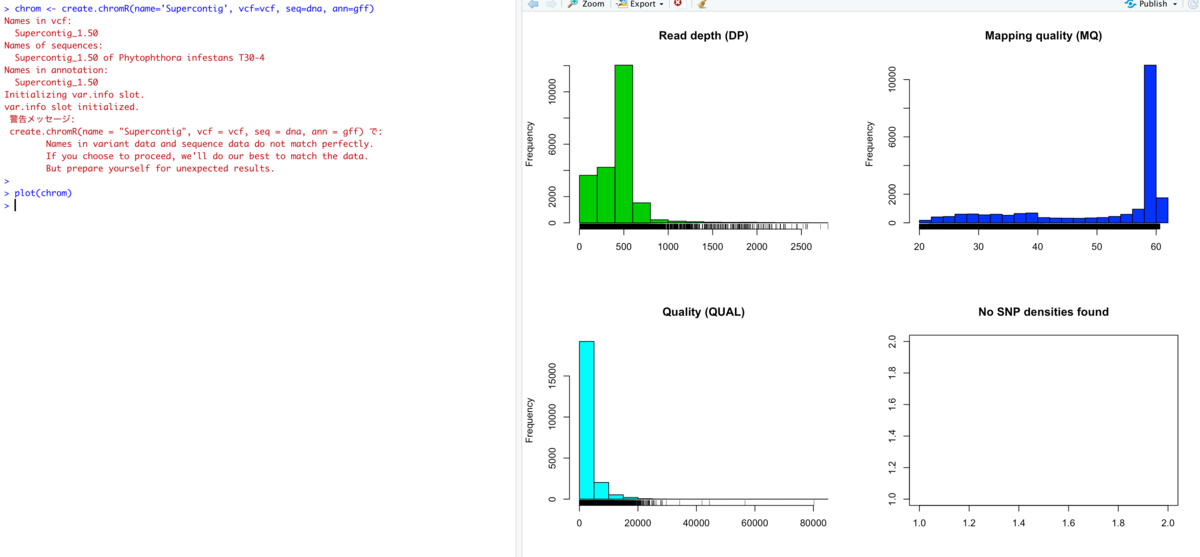
Read depthが際立っており、右の方にロングテールがある。
バリアントコーラーによっては、VCFにこれらを描画するためのフィールドを持っていない可能性がある事に注意する。また、コーラーによって定義が異なり、値の範囲が異なる可能性もある。そのため、可視化が重要な意味を持つ(マニュアルより)。
5、視覚化した結果を元に、関数masker()による信頼性の低いデータのフィルタリングを行う。chromRオブジェクト(chrom)とクオリティ(QUAL)の下限、read depthの範囲、map qualityの範囲を指定する。結果をオブジェクトchromqtに保存。
chrom <- masker(chrom, min_QUAL = 1, min_DP = 300, max_DP = 700, min_MQ = 59.9, max_MQ = 60.1)
6、前処理が終わったら、関数proc.chromR()を使ってバリアント、配列、アノテーションデータを分析する。
chrom <- proc.chromR(chrom, verbose=TRUE)
plot(chrom)
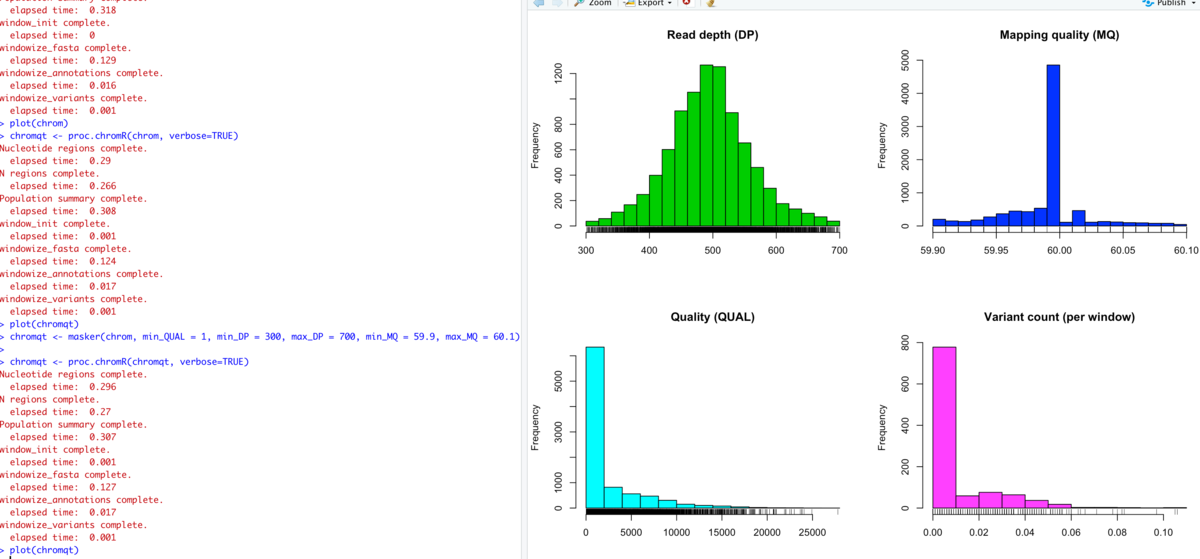
ウィンドウごとのバリアントカウントが得られた。
7、関数 chromoqc() を使ってデータの合成プロットであるクロモプロットを出力。
chromoqc(chrom, dp.alpha=20)
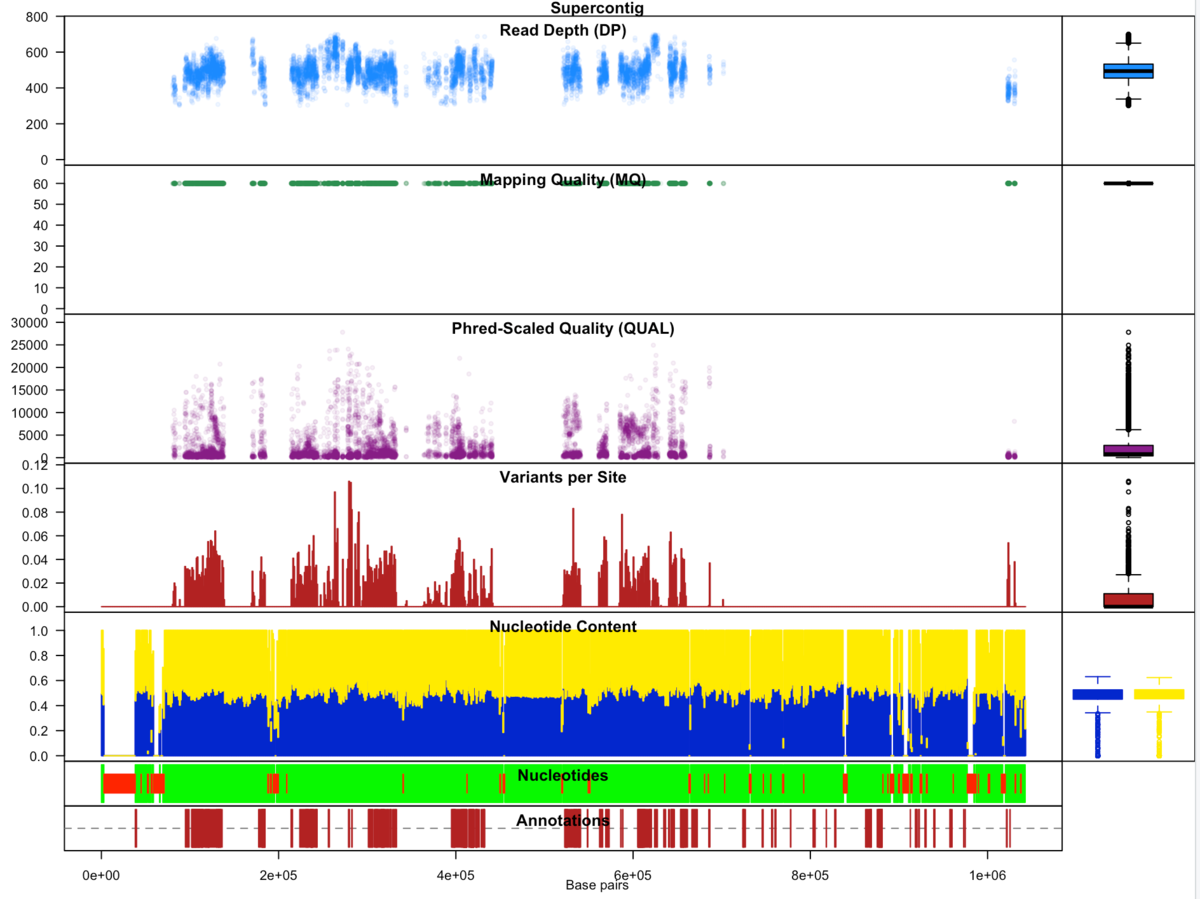
クロモプロットでは、3つのデータソースからのデータをまとめられる。一番下のプロットがアノテーションデータで、例えば、遺伝子のエクソンなどは暗赤色の長方形で表される。アノテーショントラックの上にはシーケンストラックがある。この中で、コールされたヌクレオチドは緑で、コールされていないヌクレオチド(Ns)は赤で表示される。ゲノムの質によっては、コールされていないヌクレオチドが含まれている場合がある。アノテーショントラックの上には、G/C含有量のトラックがある。その上のトラックは、ウィンドウごとのバリアントの数を要約している。その上には、phead scaleのクオリティ、マッピングクオリティ、リードデプスのドットプロットが表示される。
特定の領域だけ描画。
chromoqc(chrom, xlim=c(5.2e+05, 5.4e+05))

8、関数 write.vcf() を使ってフィルタリングしたVCFファイルを出力する。
#元のVCF
write.vcf(vcf, "output.vcf.gz")
unlink("output.vcf.gz")
#QTしたVCF
write.vcf(chrom, file="good_variants.vcf.gz", mask=TRUE)
unlink("good_variants.vcf.gz")
引用
vcfr: a package to manipulate and visualize variant call format data in R.
Knaus BJ, Grünwald NJ.
Mol Ecol Resour. 2017 Jan;17(1):44-53.
関連