biological sequences内の特徴の視覚化は、シーケンシングデータの統合分析および解釈に不可欠である。豊富なfeature annotations(例:遺伝子、プロモーター)および実験的証拠(例:RNA-Seq読み取り)のコンテキストでゲノムDNAなどの核酸配列を視覚化するソフトウェアソリューションは、インタラクティブなデータ分析および仮説生成のための不可欠な研究ツールになった。顕著な例には、UCSCゲノムブラウザ(Meyer et al、2012)およびIGV(Robinson et al。、2011)が含まれる。対照的に、タンパク質のトポロジーのコンテキスト内での翻訳後修飾(PTM)やタンパク質ドメインなどのタンパク質機能の視覚化を上記の例のように、単一のアミノ酸残基の分解能までサポートするツールはほとんどない。これらには、RbDe(Skrabanek et al、2003)、TMRPres2D(Spyropoulos et al、2004)およびTOPO2 (http://www.sacs.ucsf.edu/TOPO2)が含まれる。重要なことに、これらのツールはいずれも、自動化された方法で実験プロテオームデータを統合してはいない(詳細な比較については、補足表S1および補足データを参照)。
実験的プロテオームデータが提供するタンパク質の構造と機能に関する独自の洞察を活用するには、統合された視覚化、インタラクティブな分析、仮説の生成を可能にするツールが必要である(Gehlenborg et al、2010)。多くの重要な細胞機能を実行し、現在のすべての薬物標的の半分以上を占める膜タンパク質の場合(von Heijne、2007)、トポロジーを考慮することが特に重要である。
Protterは、アノテーション付きおよび予測されたタンパク質シーケンス機能と、ペプチドおよびPTMの実験的プロテオームエビデンスの両方をタンパク質の膜貫通トポロジーに統合および視覚化できる、インタラクティブでカスタマイズ可能なWebベースのアプリケーションである。ユーザーは、多数のアノテーションソースから選択し、独自のプロテオミクスデータファイルを統合し、ターゲットの定量プロテオミクスアプリケーションに最適なペプチドを選択し、publication品質のイラストをエクスポートできる。
manual
http://wlab.ethz.ch/protter/help/
help
https://groups.google.com/forum/#!forum/protter
http://wlab.ethz.ch/protter/start/にアクセスする。
調べたいタンパク質のUniprotのaccesion IDを入力するか、アミノ酸配列をペーストしてsubmitする。
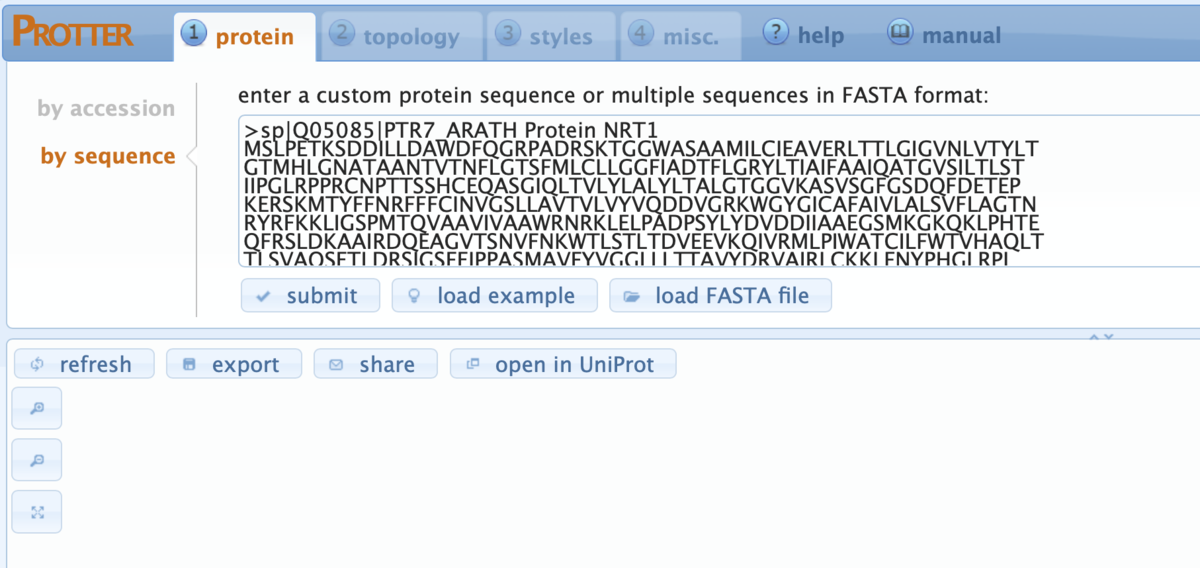
配列はリストとしても入力できる(図はonline manualより)。
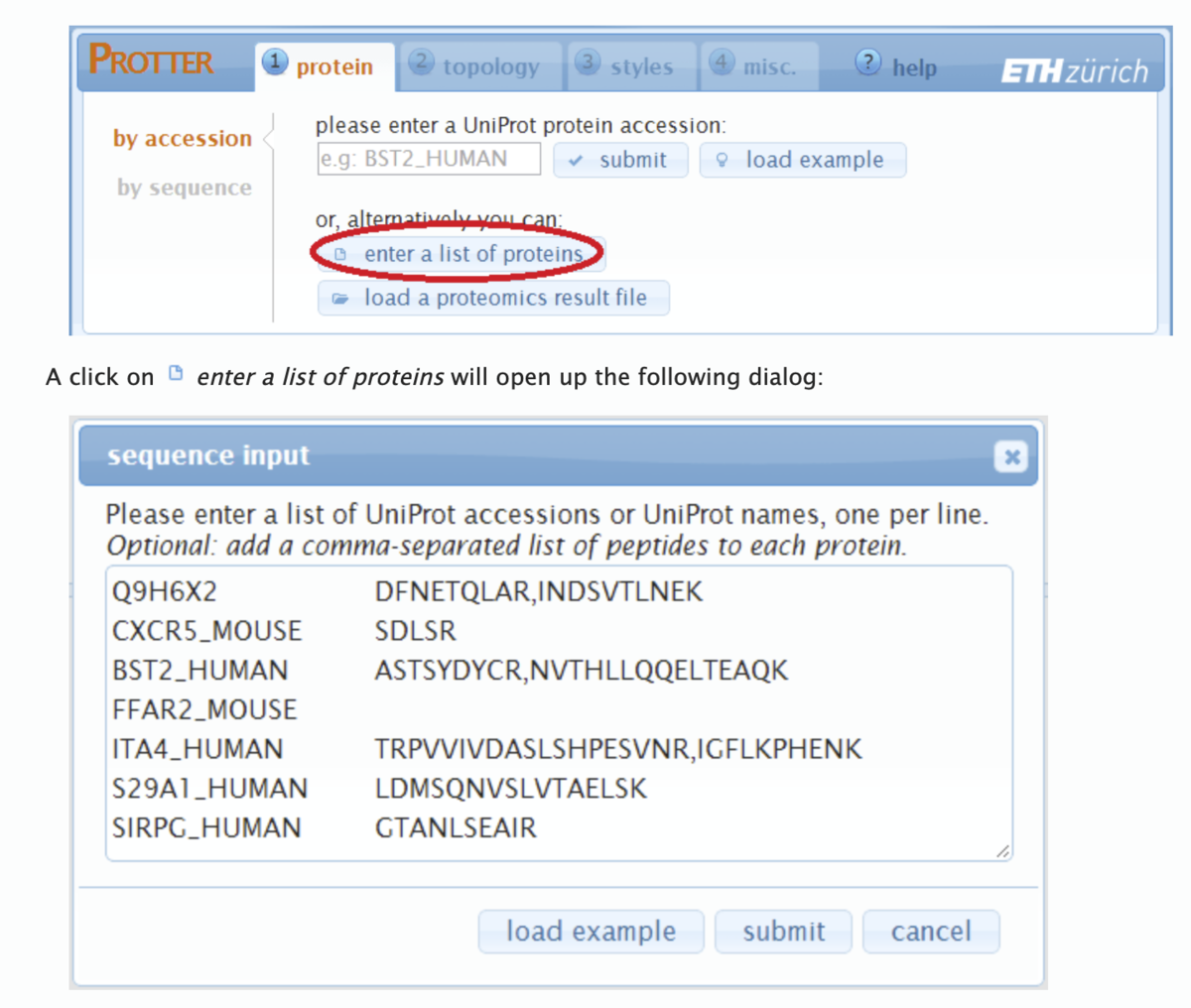
multi-fastaとして複数配列をペーストしてもO.K
結果が表示された。

上のメニューから色の指定、表記のフォーマットなど指定できる。ローマ数字に変更した。
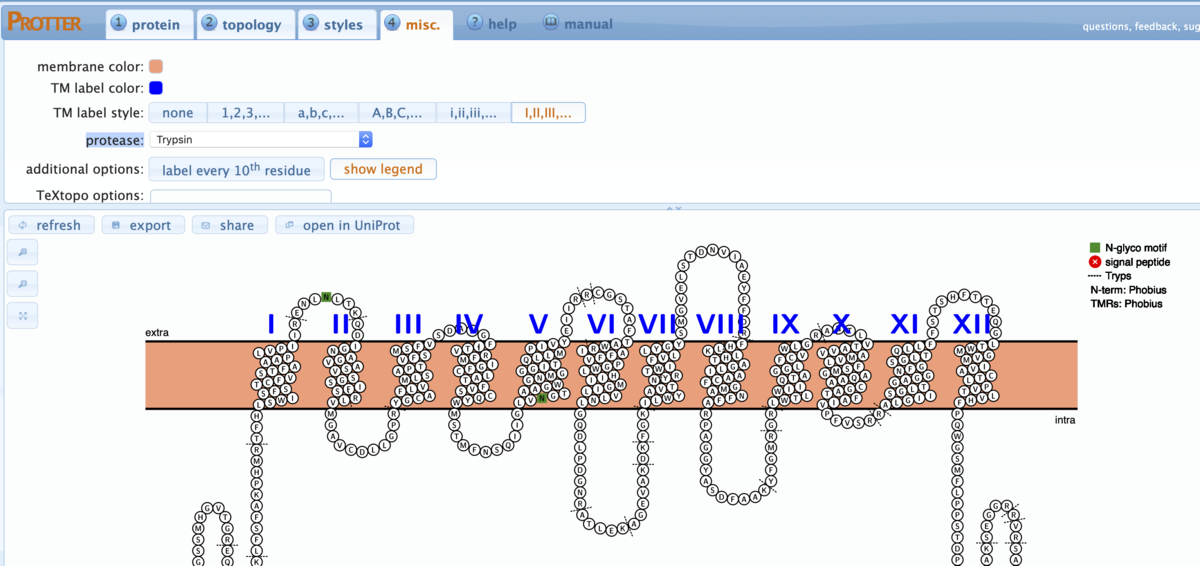
既知プロテアーゼの切断部位も追加できる。
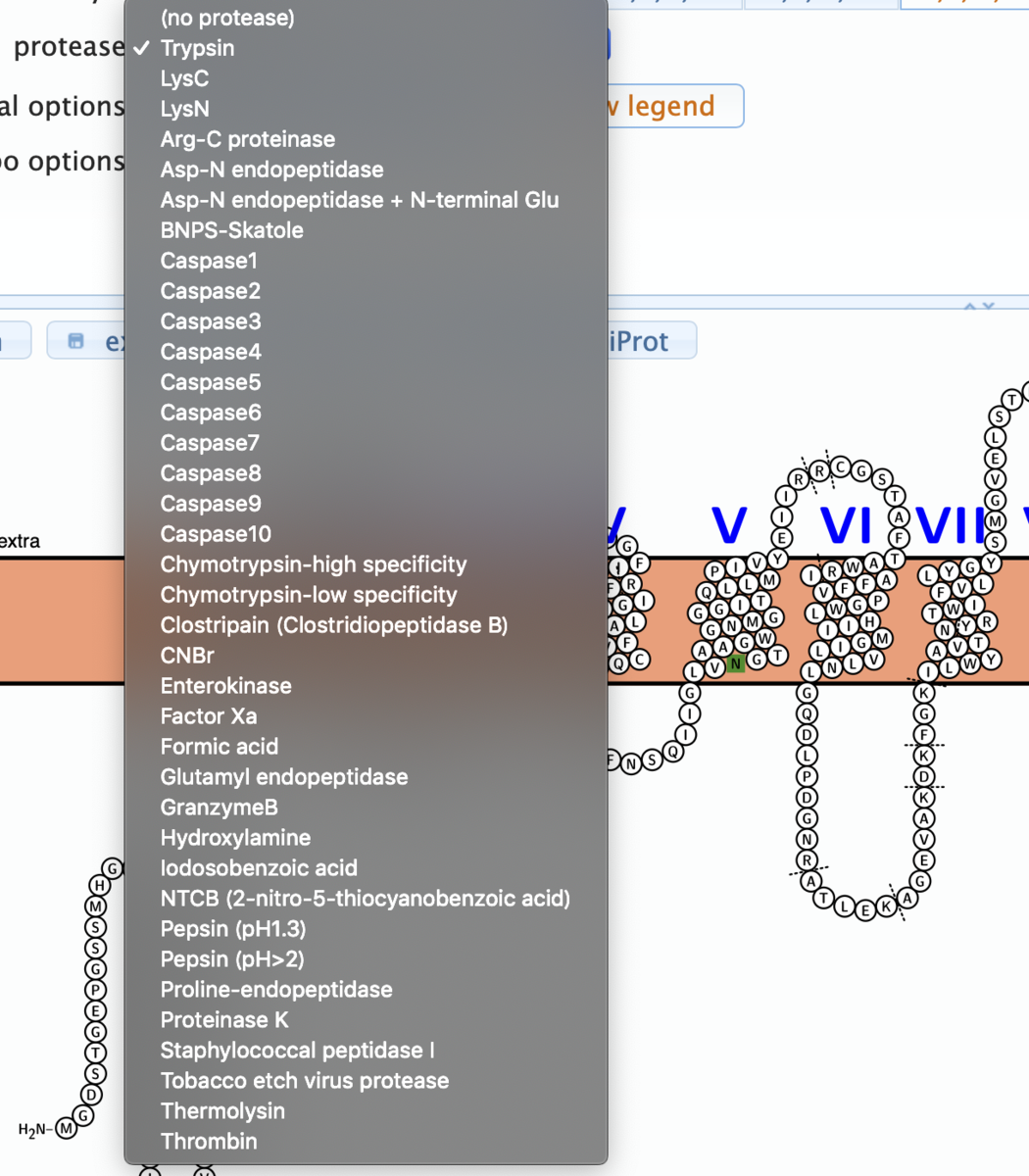
トリプシン切断部位を追加した。
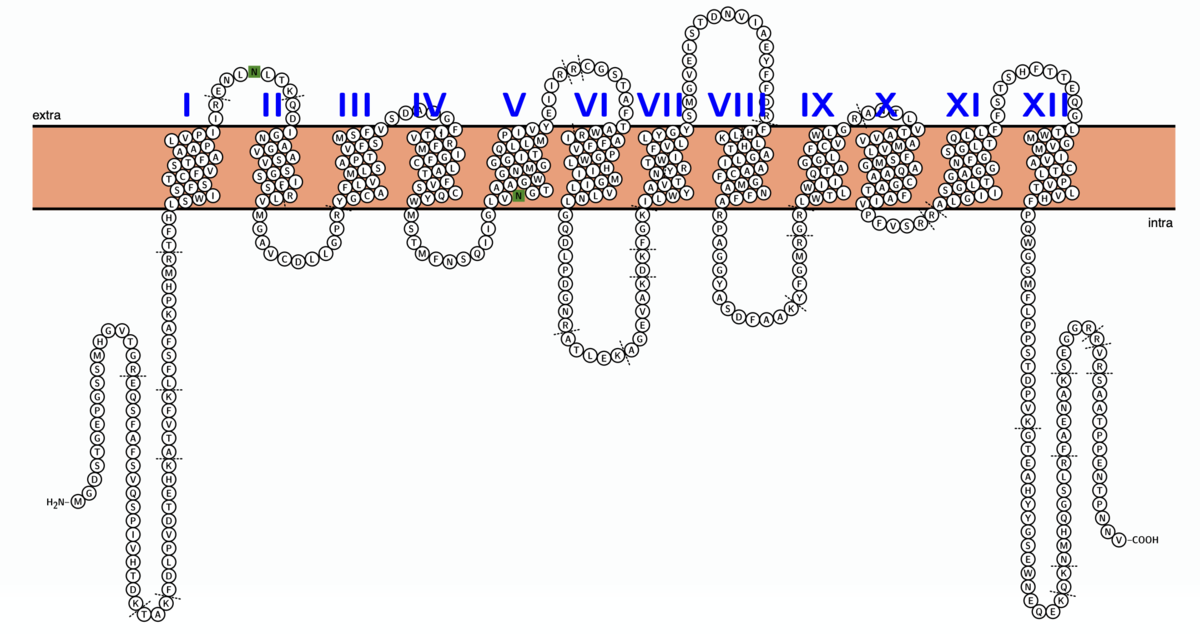
左上のボタンから図の拡大縮小、図の出力などが行える。open in UniProtをクリックすると、クエリの配列についてUNIPROTKBでblastサーチが行われる。

他の機能についてはmanualを読んでください。
引用
Protter: interactive protein feature visualization and integration with experimental proteomic data
Ulrich Omasits, Christian H. Ahrens, Sebastian Müller, Bernd Wollscheid
Bioinformatics, Volume 30, Issue 6, 15 March 2014, Pages 884–886
関連
