2020 3/2 タイトル修正。DNA解析ソフトと表記した。
2020 3/13 追記
高スループットシーケンス(HTS)テクノロジにより、多数のサンプルの低コストシーケンスが一般的になった。 ゲノムリシーケンシング、集団規模の変異検出、全トランスクリプトームシーケンシング、タンパク質結合核酸のゲノムワイド解析なども行われている。Artemisは、リファレンスゲノムとそれに対応するアノテーションのコンテキストで、さまざまなタイプのHTSデータセットの統合された視覚化と計算分析のためのツールである。
HP (Artemis、ACT、BamView、DNAPlotterの紹介)
http://sanger-pathogens.github.io/Artemis/Artemis/
poster
https://sanger-pathogens.github.io/Artemis/Artemis/artemis_genome_informatics_2010.pdf
manual
https://sanger-pathogens.github.io/Artemis/Artemis/artemis-manual.html
インストール
HPから、mac、windows、linux版をダウンロードできる。また、githubのリリースからダウンロードすることもできる。ここではgithubからmac版をダウンロードした。
依存
All recent releases from v18.0.0 onwards require a minimum of Java 9 and ideally Java 11. This must be installed first.
ダウンロードしたdmgファイルを実行、.appをApplicationにコピーする。

追記
またはcondaを使う。
conda create -n artemis
conda activate artemis
conda install -c bioconda -y artemis
#起動
art
実行方法
Artemis (CHADO)を起動する。
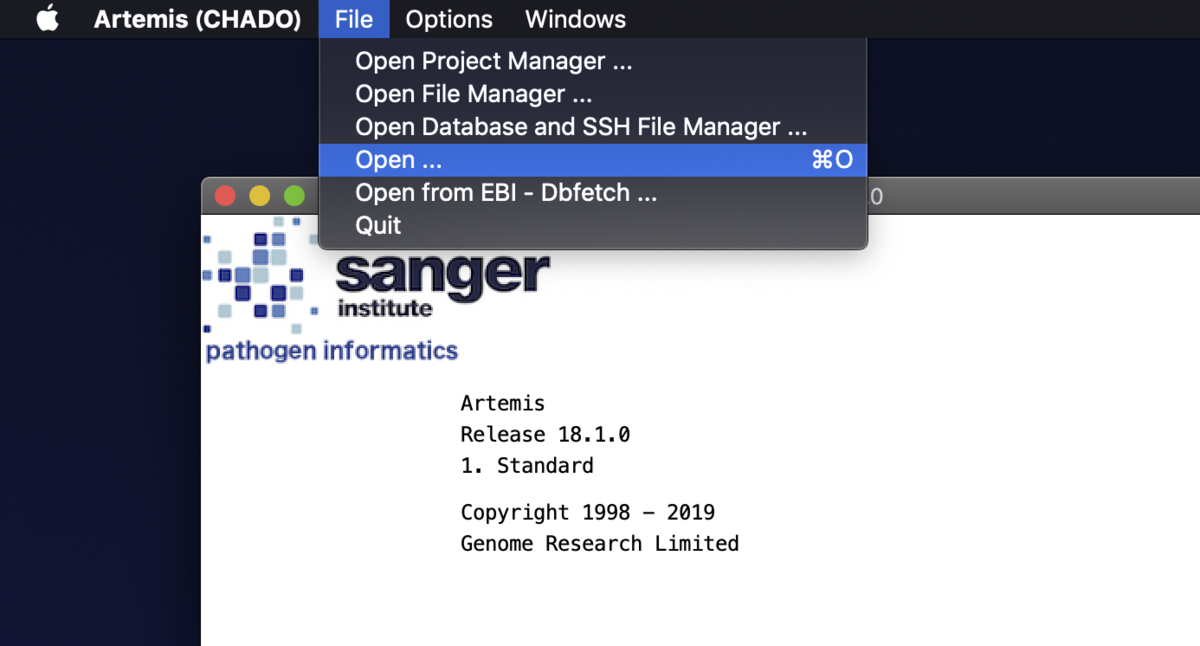
ファイルを開く。File => Open
リファレンスゲノムを読み込む。EMBL、GenBnak、FASTAなどに対応している。ここではbamファイルも読み込みたいので、そのリファレンスであるFASTAファイルを選択した。
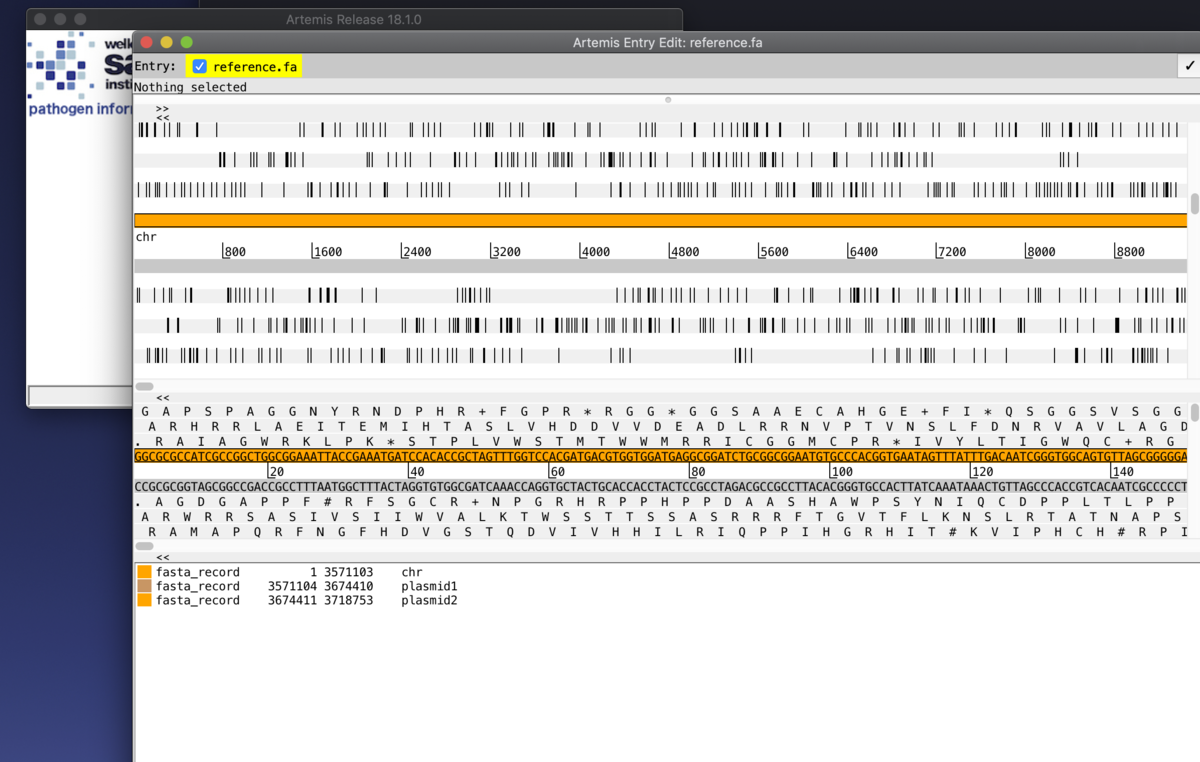
読み込まれた。
BAMを読み込む。File => Read BAM / CRAM / VCF
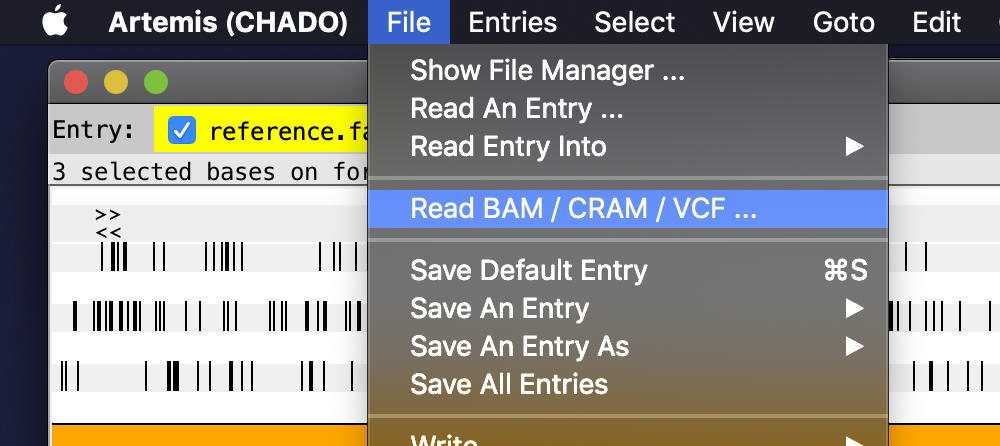
BAMファイルを指定する。
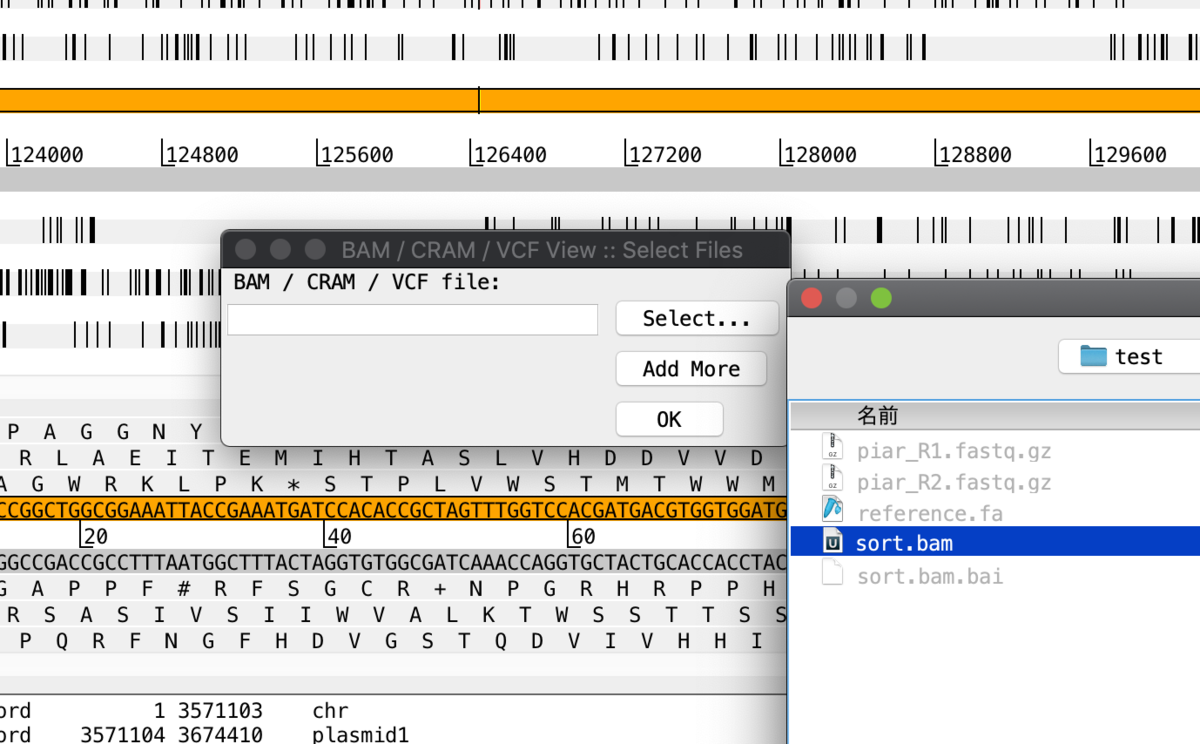
Add moreを押してVCFも同時に読み込メル。VCFはあらかじめ圧縮されてindexがつけられている必要がある。
bgzip file.vcf
tabix -p vcf file.vcf.gz
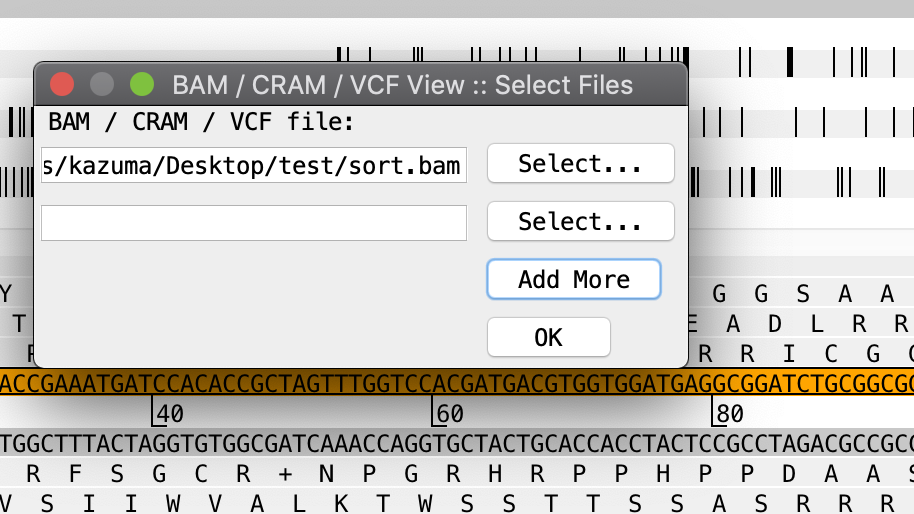
ここではbamファイルだけ読み込んだ。
reverse strandのstopコドンのラインを非表示にする。
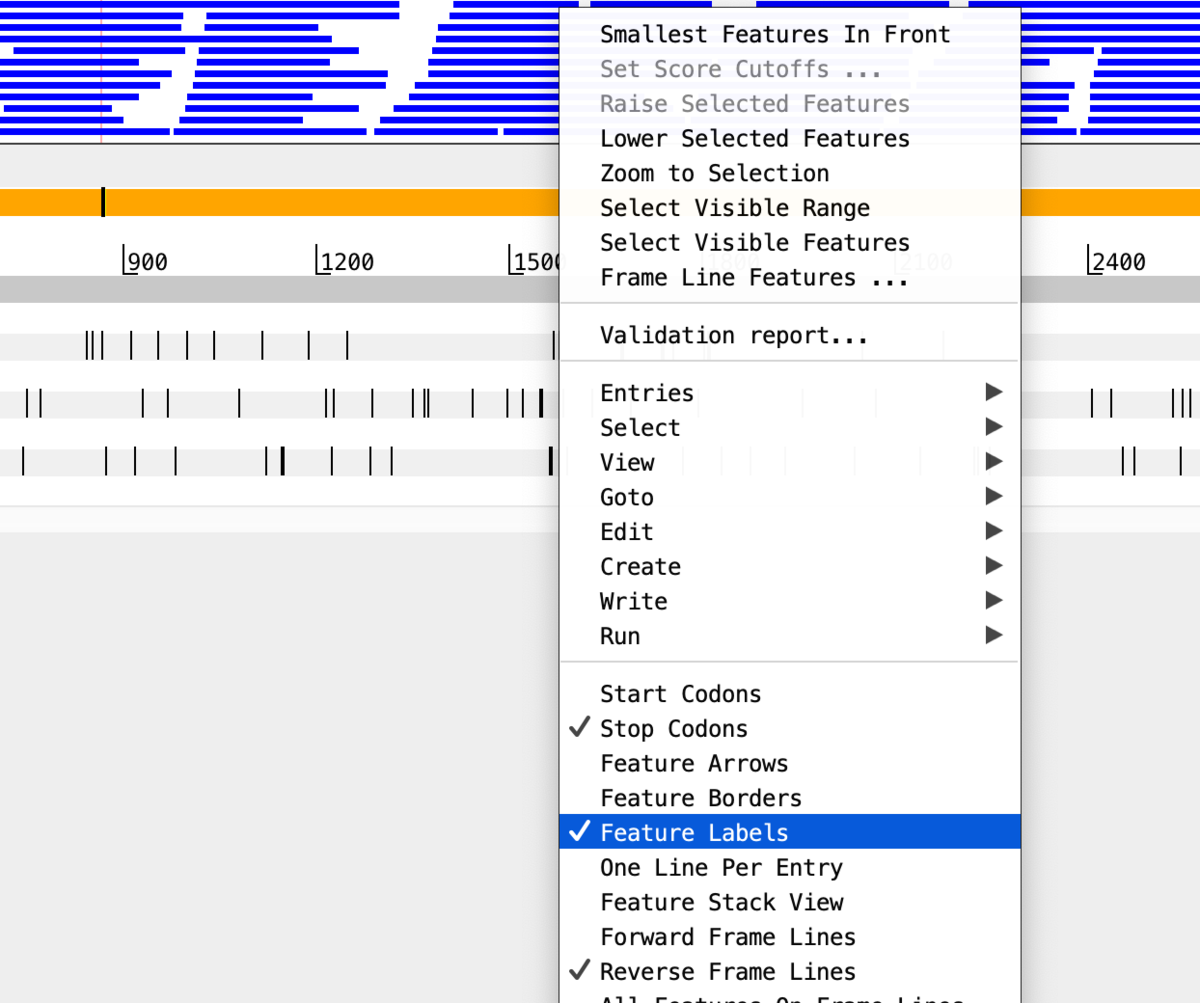
非表示になった。ここでは、左端の>>をクリックして不要なパネルも隠している。

右端の上下のスクロールバーを動かすことで拡大縮小できる。大きく縮小するとカバレッジ変化がラインで表現される。
拡大すると塩基が見えるようになる。
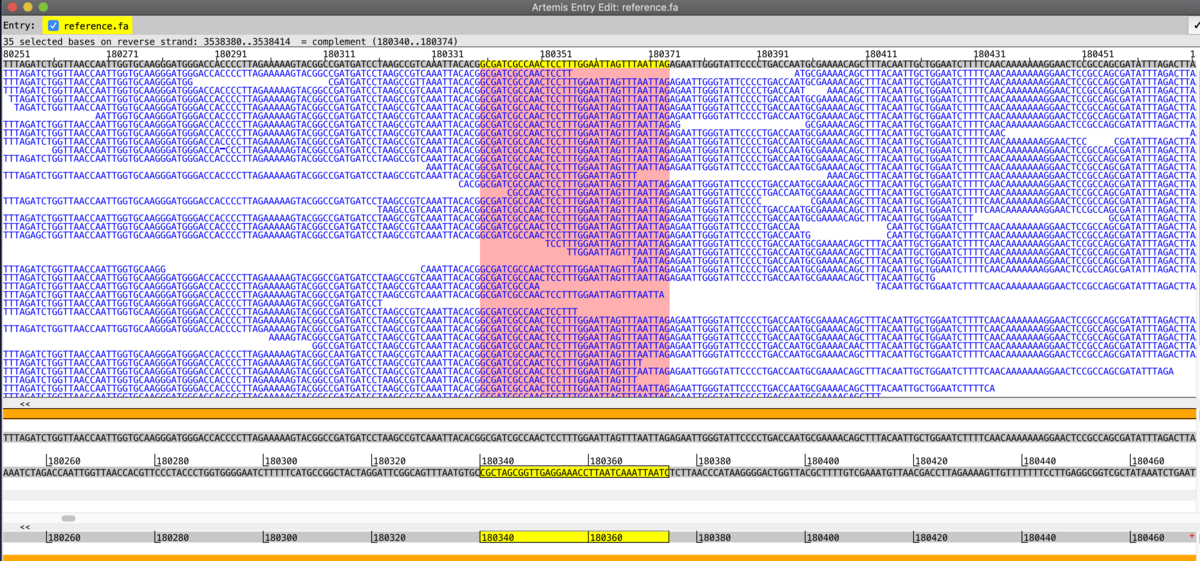
GenBankなどフィーチャー情報があるファイルを読み込んだ時は、豊富なツールを使用でき、単なるブラウザというより統合DNA解析ツールに近い(たとえばCLC genomicsやgenious)感じになる。
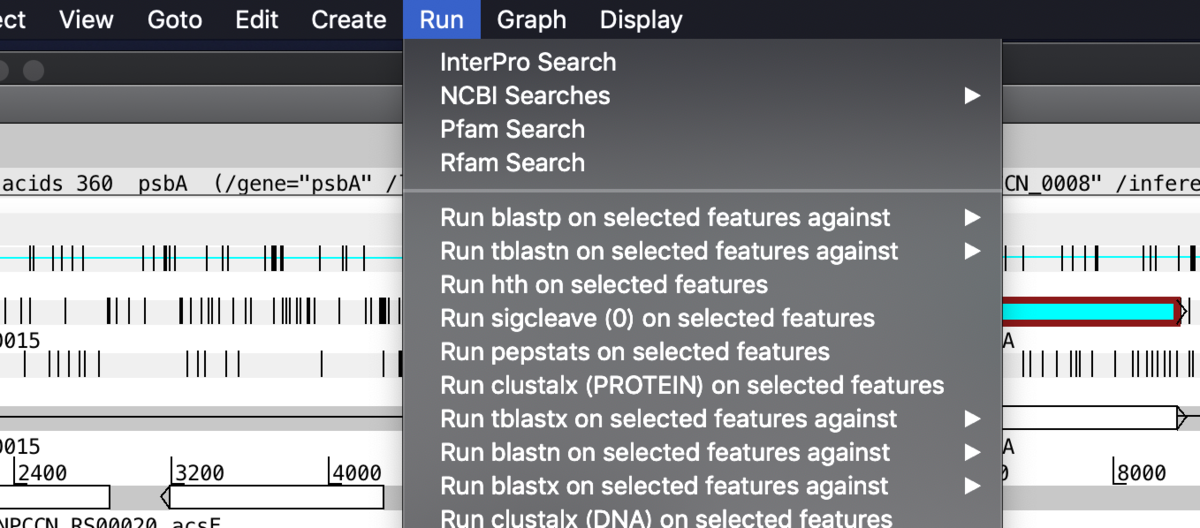
フィーチャーを選択してblastnを実行してみた。数秒待つとデフォルトのブラウザに結果が表示された(NCBIのオンラインのblast結果)。詳細な使い方はマニュアルを参照して下さい。
使用例
引用
Artemis: an integrated platform for visualization and analysis of high-throughput sequence-based experimental data
Carver T, Harris SR, Berriman M, Parkhill J and McQuillan JA
Bioinformatics (Oxford, England) 2012;28;4;464-9
Artemis and ACT: viewing, annotating and comparing sequences stored in a relational database
Carver T, Berriman M, Tivey A, Patel C, Böhme U et al.
Bioinformatics (Oxford, England) 2008;24;23;2672-6
Viewing and annotating sequence data with Artemis
Berriman M and Rutherford K
Briefings in bioinformatics 2003;4;2;124-32
Artemis: sequence visualization and annotation
Rutherford K, Parkhill J, Crook J, Horsnell T, Rice P, Rajandream MA and Barrell B
Bioinformatics (Oxford, England) 2000;16;10;944-5