2019 11/25 誤字修正
メタゲノムのde novoアセンブラについて少し誤解している人がいたので、注意喚起を兼ねて簡単にまとめておく。
メタゲノムのデータセットは特定の環境の生物の混ぜ物のシーケンシングリードに由来しているため、よく似ているがわずかに異なるclosely relatedなゲノム配列が様々なabundanceで存在している。メタゲノムのアセンブラは、この混合の(バルクの)シーケンシングリードセットから、複数の近縁な株のコンセンサスのバックボーン配列を構築するアセンブラである。この性質により、配列多少の多様性があっても、同種のゲノムはより長い領域に渡って再構成される。配列が高度に断片化していると、ゲノムを分類したり特徴を調べたりすることができなくなるため、このようなメタゲノムアセンブラの一般的性質は、下流解析を効率的に進めるためにも欠かせない特徴と言える。
しかしながら、この特徴により、異なる生物間で保存性の高い領域も1つの配列にまとめられる可能性がある。このことを理解していないと、ターゲットの配列が1生物からのみ由来すると判断して、解析に影響を及ぼす恐れがある。ここでは、ツールに振り回されないためにも、シミュレーションリードを使ってメタゲノムアセンブラの性質を実感できる簡単なテストを行ってみる。
異なるクラスタの生物間で長い領域に渡って保存性されている配列として、遺伝子クラスターが挙げられる。ここでは、よく研究されている遺伝子クラスターであるニトロゲナーゼのnifクラスターを例に説明する。下の図は、このnifクラスターの配列をNCBI のGenBnakで公開されている4生物からfetchして、blastnによるアラインメント比較した結果である。ペアワイズの配列比較と作図には、GenBankから自動でblast比較して簡単にゲノム比較の図を作れるEasyFig(紹介)を使っている。
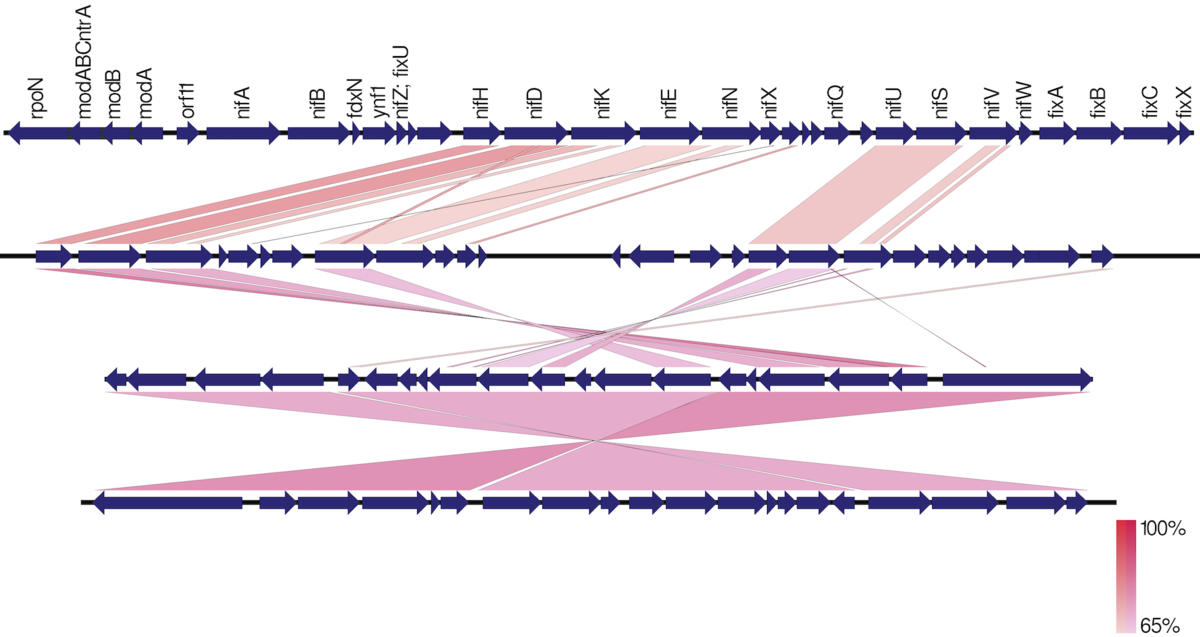
1、上では4つしかないが、以下の表の5生物のnif clusterをfetchした。これらのnif clusterの配列をリファレンスに、ARTのHiseq2500プロファイルでシミュレートして、ペアエンドのリード(150x2)を表のcoverageの深さで発生させた。

ディレクトリ構成

2、リードを結合して1つのファイルにまとめ、 de novoアセンブリを実行する。
cat */paired_1.fq > mix_1.fq
cat */paired_2.fq > mix_2.fq
#spades
spades -1 mix_1.fq -2 mix_2.fq -k auto -o spades
#metaspades
metaspades -1 mix_1.fq -2 mix_2.fq -k auto -o metaspades
3、spadesアセンブリのGFAファイルをBandageに読み込んで視覚化した。

カバレッジが低い生物のnif clusterは断片化しているが、長さとカバレッジを見る限り、カバレッジが十分な生物のnif clusterはほぼ全長が再構成できている。
次がmetaspadesのアセンブリ結果。

metaspadesでは再構成に成功している配列もあるものの、上の交差した配列群のように、共通した配列部分でパスを共有している領域がある。
結論
アンプリコンシーケンスで97% OTUなどの基準で比較を行うように、メタゲノムのアセンブラも、株レベルの細かな違いは無視して、近い距離にあるゲノムを1つのコンセンサス配列として表し、下流の様々な解析に適した配列を作成しようとする。そのため、メタゲノムのアセンブラを使うと、保存された遺伝子クラスターがパスを共有したコンセンサス配列として表現される場合があることを示した。では、仮に株レベルの違いまでアセンブラの感度を上げるとどうなるか? 実際のメタゲノムのシーケンシングリードをspadesアセンブラなどでアセンブリすれば分かるが、アセンブリグラフの様々な部位で分岐が生じるため、進化速度が遅い領域を除き、大半の領域では、アセンブリ後の配列は高度に断片化してしまう。これではそもそもアセンブルをする意味が薄くなってしまう。
メタゲノムのアセンブラは、まじめに考えれば解釈が困難な混合物のシーケンシングデータをなんとかして処理するため、また計算負荷を下げるため、アセンブリグラフをシンプルにし長い配列を出力しようと試みる。しかし、これに対するトレードオフの1つが、よく似た配列のある種誤ったコンセンサス表現である。Nif clusterのようなよく似た長い配列を分離するには、クラスターを構成するリードを回収して精度の高いspadesなどのアセンブラで再アセンブリするか(*1)、倍数体ゲノムからのハプロタイプ再構成のような追加のテクニックが必要になる。ということで、メタゲノムシーケンスの目的が保存された遺伝子クラスターの分析である場合、そのままwholeアセンブリするだけでは良い結果を出せない可能性があることを示した。
補足
実際に、これらのnifクラスターはどのくらい似ているのか、追加でall versus allのANI比較とmauveのゲノム比較を行った。
(ANI比較は4つのnifクラスターしか使用していない)。
ANI比較ではそれぞれのnifクラスターは70%以下の塩基配列相同性になった。
mauveによるゲノム比較。シンテシーブロック表現のeasyfigより細かい部分まで視覚化される。

追記
たとえcomplete MAGsが得られたとしても、それをpublic databaseに通常のゲノムとして登録することは避けるべきです。以下を読んでみてください。
*1
コミュニティの複雑性が低ければうまくいくかもしれない。