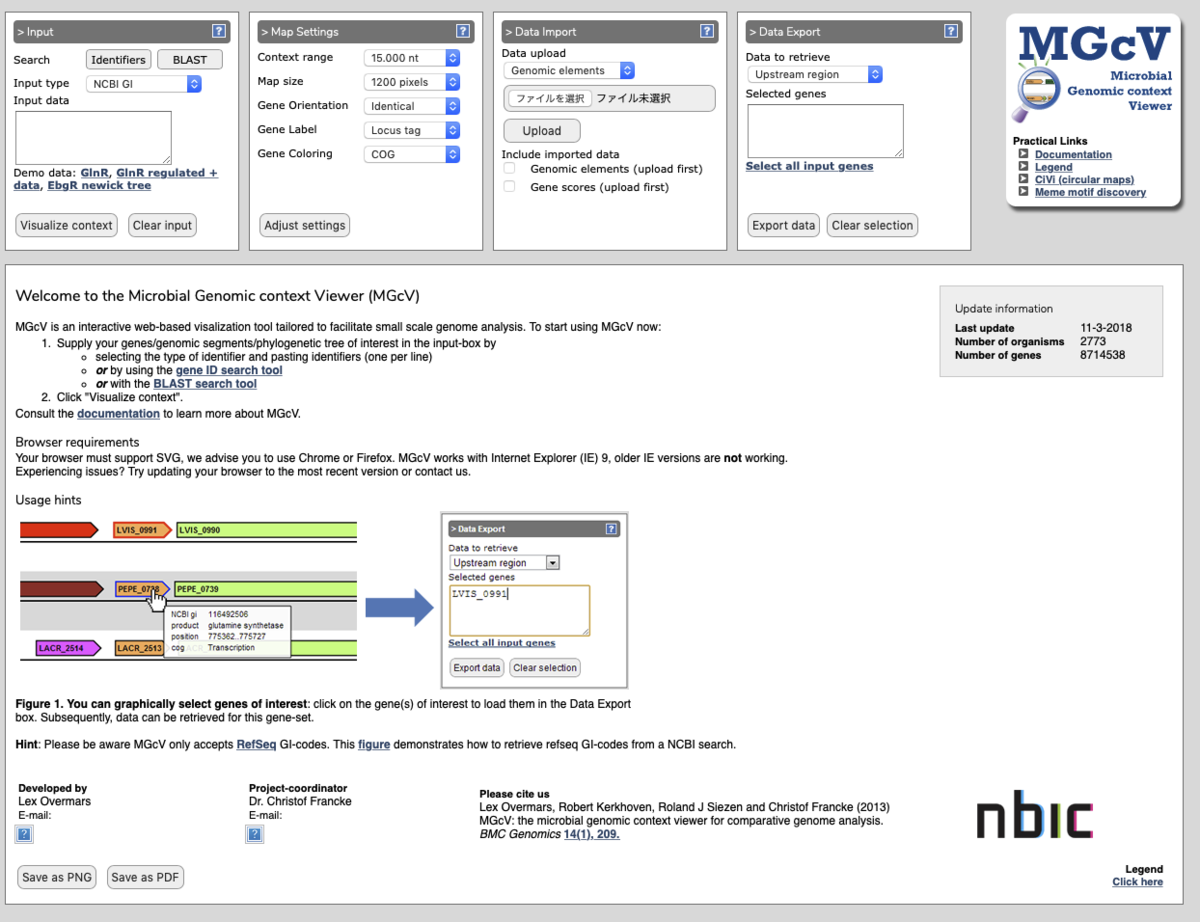
保存された遺伝子コンテキストは、多くのタイプの比較ゲノム解析で使用される。遺伝子機能のリードを提供し、調節配列の発見を導くだけでなく、代謝ネットワークの再構築を支援するために使用さされる。 Microbial Genomic context Viewer(MGcV)は、バクテリアの手動比較ゲノムコンテキスト分析の実践を強化するために調整された、インタラクティブなWebベースのアプリケーションである。
MGcVは、選択された任意の遺伝子セット、系統樹内の遺伝子、ゲノムセグメント、または調節エレメントのゲノムコンテキストを視覚化する、汎用性が高く使いやすいツールである。遺伝子機能のインタラクティブなアノテーション、調節エレメントの発見、または遺伝子調節ネットワークのシーケンスベースの再構築などの面倒なタスクを容易にするように調整されている。 MGcVは、NCBIからPfamドメイン、細胞内位置予測、GCコンテンツなどの遺伝子配列特性などの他のアノテーションデータと、原核遺伝子に関する情報を視覚的に統合する。また、転写調節の分析と再構築において、データの収集(上流領域、ID、アノテーションなど)を促進するための遺伝子のグラフィカルな選択を可能にするインタラクティブ機能の有用性を示す。さらに、推定調節因子とそれに対応するスコアまたはRNA-seqおよびマイクロアレイ実験からのデータをアップロードし、視覚化して、(ランク付けされた)比較コンテキストマップで解釈できる。ランク付けされたマップにより、予測される調節因子と実験データを相互に照らし合わせて解釈できる。
Documentation
https://mgcv.cmbi.umcn.nl/help.html
使い方
https://mgcv.cmbi.umcn.nl にアクセスする。
遺伝子/ゲノム領域の識別子(identifiers)を貼り付ける。
example dataを使う。
1、左上のウィンドウにNCBI GI(リンク)をペーストする。

視覚化するサイズや情報を指定する。

視覚化された。

NCBI GIはバージョンによって変化するのでややこしい。不明の時はMGcV組み込みのサーチツールを使うのが早い。左上のIdentifiersをクリックするとウィンドウが出現する。このウィンドウ内でIDを検索する。ゲノムが登録されている菌の株名、=> 酵素名の手順で絞り込める。

表示された候補を左上のウィンドウにペーストする。
多くの領域を一度に視覚化することもできる。

newick formatのファイルにも対応している。ただし各ラベルはNCBI-GIになっていないといけない。

関連する情報をexportしてMEMEに渡すことができる。

テスト時は機能しなかった(*1)。
引用
MGcV: the microbial genomic context viewer for comparative genome analysis
Overmars L, Kerkhoven R, Siezen RJ, Francke C
BMC Genomics. 2013 Apr 1;14:209
関連
*1 macos10.14にてchrome使用。