2019 11/17 補足説明追記
2019 12/28 インストールコマンド修正
2020 1/17 実行例追加
2020 10/19 help更新 (v.1.2.10)
2021 2/22 ツイート追記
本著者らはパブリックシーケンスデータベースへのゲノム送信をサポートする原核生物ゲノムアノテーションパイプラインDFASTを開発した。 DFASTは元々オンラインアノテーションサーバーとして開始され、現在までに(論文執筆時点)、2016年の最初の立ち上げ以来、7000以上のジョブが処理されている。ここでは、スタンドアロンコマンドとしても利用可能なDFAST用に新しく実装されたバックグラウンドアノテーションエンジンを紹介する。新しいエンジンは、代表的なサイズのバクテリアゲノムに、偽遺伝子、translation exceptions、特定のリファレンスゲノム間のオロソロガスな遺伝子の割り当てなどの豊富な情報を10分以内にアノテーション付けできる。さらに、DFASTのモジュラーフレームワークにより、ユーザーはアノテーションワークフローを簡単にカスタマイズできる。また、新しい機能の拡張と将来の新しいツールの組み込みも容易になっている。このソフトウェアはPython 3で実装され、Python 2.7と3.4の両方で実行できる(MacintoshおよびLinuxシステム上)。GPLv3ライセンスでhttps://github.com/nigyta/dfast_core/から無料で入手できる。オンライン版もhttps://dfast.nig.ac.jp/で利用できる。
FAQ
sample result
微生物ゲノムアノテーションパイプライン DFAST はサーバー移転を行いました。
— ytanizaw (@ytanizaw) 2021年2月22日
新しいURLは https://t.co/rnTOmxJe3f となります。
遺伝研スパコンを用いてジョブ実行を行うことで、同時実行数の増加(2→8)、実行時間の短縮を実現しました。
スタンドアローン版のファイルダウンロードツール dfast_file_downloader.py は最新版にアップデートしてください。https://t.co/8ek213cVuW
— ytanizaw (@ytanizaw) 2021年2月22日
スタンドアローン版の最新版はv1.2.13です。近日中にcondaからも利用可能になります。
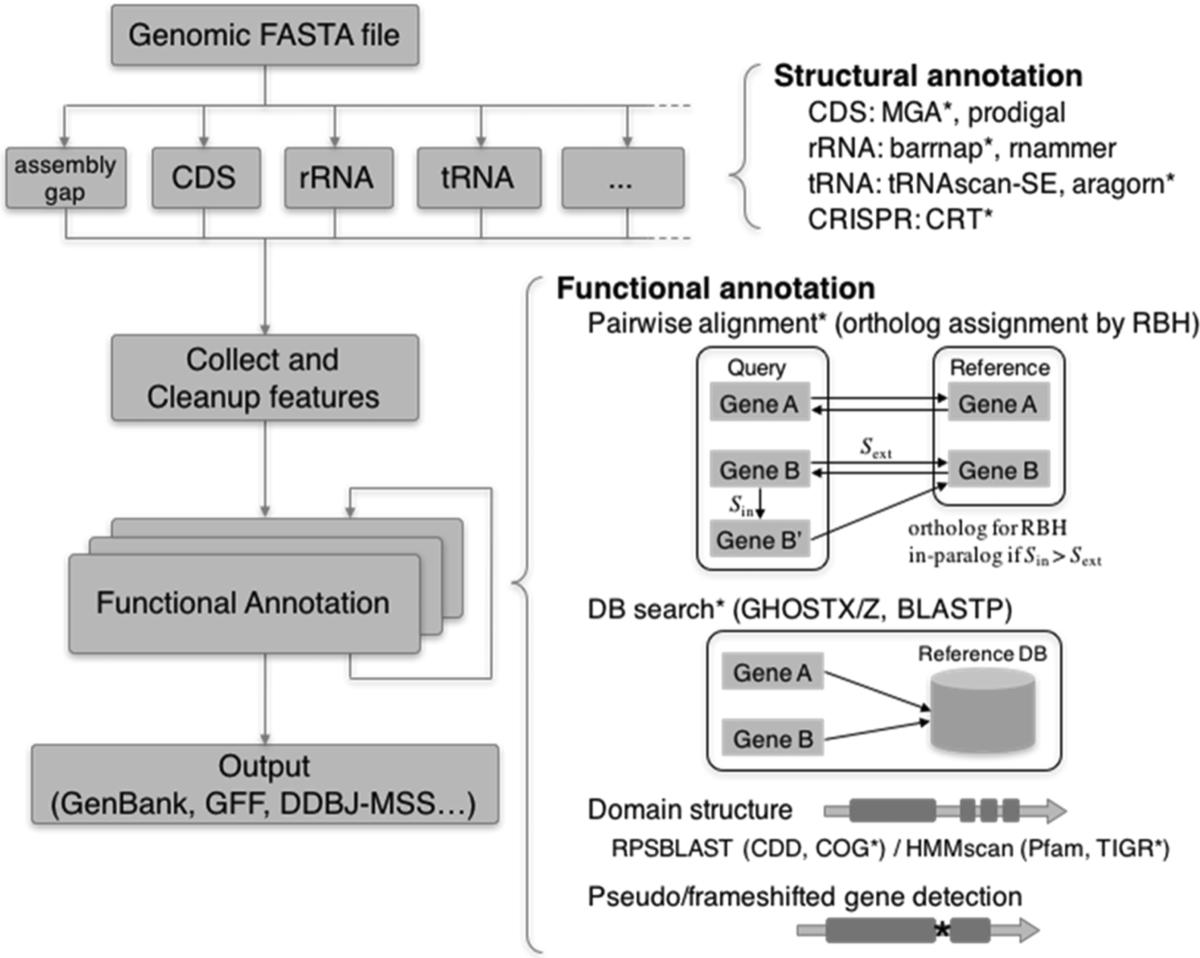
DFAST annotation workflow. 論文より転載
インストール(オフラインで使う場合)
Gihub
#bioconda (link)ここでは仮装環境に入れる
conda create -n dfast -y
conda activate dfast
conda install -c bioconda -y dfast
> dfast
$ dfast -h
usage: dfast -g your_genome.fna [options]
DFAST: DDBJ Fast Annotation and Submission Tool version 1.2.10.
Basic options:
-g PATH, --genome PATH
Genomic FASTA file
-o PATH, --out PATH Output directory (default:OUT)
-c PATH, --config PATH
Configuration file (default config will be used if not
specified)
--organism STR Organism name
--strain STR Strain name
Genome settings:
--complete BOOL Treat the query as a complete genome. Not required
unless you need INSDC submission files.
[t|f(=default)]
--use_original_name BOOL
Use original sequence names in a query FASTA file
[t|f(=default)]
--sort_sequence BOOL Sort sequences by length [t(=default)|f]
--minimum_length INT Minimum sequence length (default:200)
--fix_origin Rotate/flip the chromosome so that the dnaA gene comes
first. (ONLY FOR A FINISHED GENOME)
--offset INT Offset from the start codon of the dnaA gene. (for
--fix_origin option, default=0)
Locus_tag settings:
--locus_tag_prefix STR
Locus tag prefix (defaut:LOCUS)
--step INT Increment step of locus tag (default:10)
--use_separate_tags BOOL
Use separate tags according to feature types
[t(=default)|f]
Workflow options:
--threshold STR Thresholds for default database search (format:
"pident,q_cov,s_cov,e_value", default: "0,75,75,1e-6")
--database PATH Additional reference database to be searched against
prior to the default database. (format:
db_path[,db_name[,pident,q_cov,s_cov,e_value]])
--references PATH Reference file(s) for OrthoSearch. Use semicolons for
multiple files, e.g. 'genome1.faa;genome2.gbk'
--aligner STR Aligner to use [ghostx(=default)|blastp|diamond]
--use_prodigal Use Prodigal to predict CDS instead of MGA
--use_genemarks2 STR Use GeneMarkS2 to predict CDS instead of MGA.
[auto|bact|arch]
--use_trnascan STR Use tRNAscan-SE to predict tRNA instead of Aragorn.
[bact|arch]
--use_rnammer STR Use RNAmmer to predict rRNA instead of Barrnap.
[bact|arch]
--gcode INT Genetic code [11(=default),4(=Mycoplasma)]
--no_hmm Disable HMMscan
--no_cdd Disable CDDsearch
--no_cds Disable CDS prediction
--no_rrna Disable rRNA prediction
--no_trna Disable tRNA prediction
--no_crispr Disable CRISPR prediction
--gff GFF [Preliminary implementation] Read GFF to import
structural annotation. Ignores --use_original_name,
--sort_sequence, --fix_origin.
Genome source modifiers and metadata [advanced]:
These values are only used to create INSDC submission files and do not
affect the annotation result. See documents for more detail.
--seq_names STR Sequence names for each sequence (for complete genome)
--seq_types STR Sequence types for each sequence (chromosome/plasmid,
for complete genome)
--seq_topologies STR Sequence topologies for each sequence
(linear/circular, for complete genome)
--additional_modifiers STR
Additional modifiers for source features
--metadata_file PATH Path to a metadata file (optional for DDBJ submission
file)
--center_name STR Genome center name (optional for GenBank submission
file)
Run options:
--cpu INT Number of CPUs to use
--use_locustag_as_gene_id
Use locustag as gene ID for FASTA and GFF. (Useful
when providing DFAST results to other tools such as
Roary)
--dbroot PATH DB root directory (default:APP_ROOT/db
--force Force overwriting output
--debug Run in debug mode (Extra logging and retaining
temporary files)
--show_config Show pipeline configuration and exit
--version Show program version
-h, --help Show this help message
> dfast_file_downloader.py -h
$ dfast_file_downloader.py -h
usage: dfast_file_downloader.py [-h] [--protein STR [STR ...]]
[--cdd STR [STR ...]] [--hmm STR [STR ...]]
[--assembly [ACCESSION [ACCESSION ...]]]
[--assembly_fasta [ACCESSION [ACCESSION ...]]]
[--no_indexing] [-o PATH | -d PATH]
DFAST file downloader
--protein, --cdd, --hmm: For DFAST reference libraries.
Files will be downloaded to DB root directory by default.
DB root can be specified with "--dbroot" option.
--assembly, --assembly_fasta: For Reference genomes
Reference genome file will be downloaded from NCBI Assembly Database either in GenBank or Fasta format.
Files will be written to the current directory or the directory specified with "--out" option.
optional arguments:
-h, --help show this help message and exit
--protein STR [STR ...]
DFAST reference databases. [dfast|bifido|cyanobase|ecoli|lab]
--cdd STR [STR ...] Preformatted RPS-BLAST database. [Cdd|Cdd_NCBI|Cog|Kog|Pfam|Prk|Smart|Tigr]
--hmm STR [STR ...] Preformatted RPS-BLAST database. [Pfam|TIGR|dbCAN]
--assembly [ACCESSION [ACCESSION ...]]
Accession(s) for NCBI Assembly DB. eg. GCF_000091005.1 GCA_000008865.1
--assembly_fasta [ACCESSION [ACCESSION ...]]
Accession(s) for NCBI Assembly DB. eg. GCF_000091005.1 GCA_000008865.1
--no_indexing Do not perform database indexing
-o PATH, --out PATH Output directory (default: current directory.
For --assembly, --assembly_fasta. Not allowed with argument --dbroot)
-d PATH, --dbroot PATH
DB root directory (default: APP_ROOT/db.
For --protein, --cdd, --hmm. Not allowed with argument --out)
1、オンラインでの利用
https://dfast.nig.ac.jp にアクセスする。
ゲノムのFASTAファイルをアップロードする。任意でタイトルやメールアドレスも記載する。
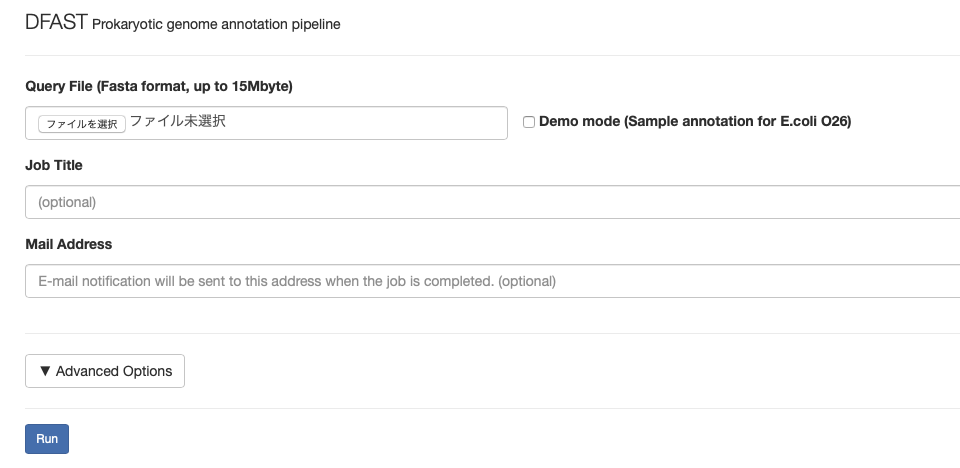
メールアドレスを記載しておくと、ジョブ終了後にメールが届く。
Advanced optionをクリックすると、サーチの感度設定、locus tag名の指定など、様々指定できる。ゲノム登録のことも考えて、この時点でふさわしい名前にしておくとよい。DnaAのオプションをつけておくと、環状ゲノムの想定でDnaAタンパク質をコードする遺伝子の上流を先頭にしてからアノテーションを行なってくれる(DnaA遺伝子上流には一般的に遺伝子がコードされていないため、ORFがゲノム配列の先頭と最後でsplitするリスクがなくなる(*1)。また、ゲノムの比較もしやすくなる。)。デフォルトではオフセット100bpになっている。

OptionでNCBI COGデータベースとTIGRAMsを使ったサーチも実行できる。
より近い生物のdatabaseからアノテーションをかけた方が一般的に結果は良くなる。下記から選択可能。

Runを押して実行する。すぐに終わるので、このままタブに残しておくとよい。

早ければ数分で結果が返ってくる(放置しておくと自動更新で結果が表示される)。annotation.zipをクリックすると、全アノテーションデータをzipでダウンロードできる。

DDBJのMSSに登録する時のファイルも入っている。
2、オフラインでの利用 (*2)
データベースのダウンロード(詳しくはdfast GithubのREADME参照)
dfast_file_downloader.py --protein dfast
例えばDnaA遺伝子の上流100bpが先頭(5'末端 、つまり1bp目)に来るようにフラグをつける(Finishしたゲノムのみで正常動作する)。 CPUスレッド8指定、出力はout。
dfast -g input_genome.fasta --fix_origin --offset 100 -o OUT --cpu 8
引用
DFAST: a flexible prokaryotic genome annotation pipeline for faster genome publication
Yasuhiro Tanizawa, Takatomo Fujisawa, Yasukazu Nakamura
Bioinformatics, Volume 34, Issue 6, 15 March 2018, Pages 1037–1039
DFAST and DAGA: web-based integrated genome annotation tools and resources.
Tanizawa Y, Fujisawa T, Kaminuma E, Nakamura Y, Arita M
Author information
Biosci Microbiota Food Health. 2016;35(4):173-184. Epub 2016 Jul 14
*1
初期に登録されたゲノムには、そうゆうものがあったりして面倒だった。
*2
メモリがそれなりにないと、スワップが連続して起きてターミナルがハングアップするので注意する。物理メモリ24GBのマシンは1度ハングアップした。