鑑別診断 (differential diagnostics, wiki) およびトランスレーショナルリサーチの基盤となるのは、詳細な表現型の特定である: 詳細な個々の臨床異常のコンピューター分析である(ref.1,2)。 Human Phenotype Ontology(HPO, wiki)は、計算によるdeep phenotypeの最も包括的なリソースを提供し、計算可能な病気の定義、臨床異常の説明、またはゲノム診断を支援するために、希少疾患の分野におけるdeep phenotypeの事実上の標準になった。 Monarch Initiative(ref.3,4)の基本的かつ統合的なコンポーネントであるHPOは、学術的および商業的な多数の組織によって国際的に採用されている。これらには、100,000ゲノムプロジェクト、NIH未診断疾患プログラムおよびネットワーク(UDPおよびUDN)、未診断疾患ネットワークインターナショナル(UDNI)、RD-CONNECT、SOLVE-RDおよびその他多く(ref.5–9)が含まれる。 HPOは最近、国際希少疾病研究コンソーシアム(IRDiRC)認定リソースとしての地位を獲得し、ゲノミクスと健康のためのグローバルアライアンス(ref.10)および関連する仲人取引所(ref.3,11)で使用されている。ここでは、このシリーズの以前の論文(ref.12,13)から改訂、拡張、または発明した統合HPOリソースについて説明する。
本著者らは、以前、表現型駆動のゲノム診断をサポートするため、著者らのグループや他の研究者によって開発された一連のアルゴリズムについて報告した(ref.12)。それ以来、HPOはますます多くのユースケースに適用されている。現在、HPOの使用は、臨床全エクソームおよびゲノムシーケンス(WES / WGS)データの分析(ref.14〜25)、およびトランスレーショナルリサーチとバイオインフォマティクスのデータ統合(ref.16、26〜39)で一般的である。electronic health-record (EHR)由来の請求コードのHPO termsへのマッピングに基づく表現型リスクスコアにより、EHR表現型の高スループットの確認が可能になり、メンデル遺伝病のケースとコントロールが区別でき、病原性のメンデル変異と関係があるバリアントを確認して特徴付けられる(ref.40)。別の設定では、自然言語処理によりHPO termsを抽出するためにEHRが調査され、結果として得られたtermは、小児患者のメンデル遺伝病の原因遺伝子の優先順位付けに成功した(ref.41)。さらに、ますます多くの商用アプリケーションがHPO termsを使用している。たとえば、SimulConsult Genome-Phenome AnalyzerはHPO termsを使用して検出結果にタグを付ける。これは現在、エクスポートされたレポートのコードを使用してユーザーが入力した調査結果を文書化するために使用されており、コードは診断で考慮される入力として電子健康記録の調査結果を識別するためにも使用される(ref.42)。 HPOの主要な機能は、哺乳類表現型オントロジー(MP)(ref.43)、Uberon(ref.44)、Cell Ontology(ref.45)などの基本的な研究オントロジーとの論理的な相互運用性である。この相互運用性は、Exomiserツール内で活用される(以下で説明)。国際マウス表現型コンソーシアム(IMPC)は、最近、ヒトのメンデル遺伝病の360の新しい候補分子の原因を特定した(ref.46)。これらには、心筋に影響を及ぼす遺伝性心疾患「不整脈誘発性右室異形成」、および筋力低下とぎこちない歩行をもたらす神経損傷を特徴とする「シャルコー・マリー・トゥース病, wiki」が含まれていた。この発見は、(i)ヒトの疾患がその成分HPO表現型の観点から定義されていたために可能になった。 (ii)マウスの表現型はMPにマッピングされた。 (iii)Monarchの表現型比較アルゴリズム(ref.47)は、HPとMPを簡単に横断するように設計されている。同様に、ラットゲノムデータベース(RGD)は、哺乳類表現型(MP)オントロジーの表現型termを使用して、表現型の遺伝子、QTL、および系統にアノテーションを付ける(ref.43)。最近では、RGDはヒトの表現型のアノテーションをMPからHPOに変換した(ref.48)。
(複数段落省略)
さらに、HPOプロジェクトは、フレームワークHIPBI-RD(希少疾患分野での相互運用性を高めるためにフェノミクス情報を調和させる)でOrphanetとの長期的な共同作業を行っており、SOLVE-RDプロジェクトのフレームワーク、および希少疾患のための欧州共同資金プログラム(EJP-RD)で継続される。このプロジェクトにより、Orphanetデータベース内の疾患に関する60 000以上のHPOアノテーションと、1,000を超える新しいtermのリクエストおよび既存のHPO termsの他の改善がもたらされた。表現型疾患のアノテーションには、表現型が確立された診断基準の一部であるか、または病態徴候であるという事実だけでなく、疾患における表現型の発生頻度が含まれる(論文表2)。これらのアノテーションはダウンロードして利用でき、Orphanet Webサイトで参照できる。さらに、このコラボレーションによりHPO-ORDOオントロジーモジュール(HPOとOrphanet Rare Diseasesオントロジーを併用できるHOOM)が作成された。
東北メディカルのHPOのアップデートに関するアナウンス(update in 2017)
ヒトの表現型のオントロジーに関する論文がNucleic Acids Research誌 に掲載されました | 東北メディカル・メガバンク機構
統合TV
2015-12-05 Phenotype Ontologies and their role in understanding rare disorders
External Tools
https://hpo.jax.org/app/tools/external
Exomiserについて(ref.2)
https://hpo.jax.org/app/tools/exomiser
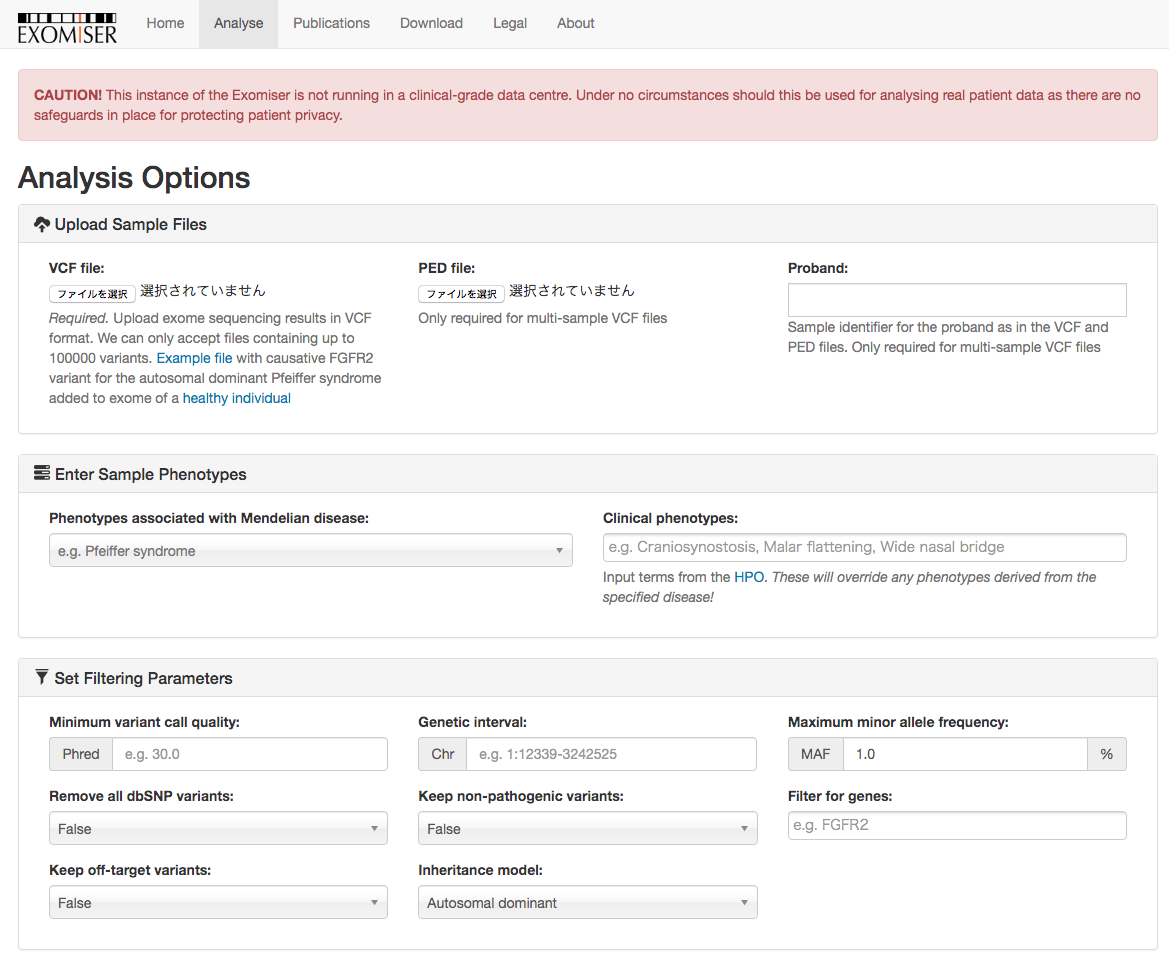
WESは新規疾患遺伝子発見の研究に革命をもたらしたが、低品質および人々に一般的なバリアントや病原性ではないと思われるバリアントを除去する一般的なフィルタリング戦略の後に残っている候補バリアントの数が非常に多いため、多くのケースが未解決のままである。 Exomiserは、オフターゲット、共通、および同義のバリアントをさらに検討することにより、全エクソームデータセットをフィルタリングし、予測される病原性とマイナーアレル頻度(バリアントスコア)に基づいて残りのバリアントを評価する。最終ランキングは、個々のスコアの合計として計算され、PHIVEスコアが生成される。
使い方
https://hpo.jax.org/app/にアクセスする。
termの階層構造確認、termの詳細の閲覧、疾患名からのterm検索など様々な機能があるが、ここではExomiserの流れについて簡単に紹介する。
オンラインで使う場合、下記にアクセスする(ローカル)。
https://monarch-exomiser-web-dev.monarchinitiative.org/exomiser/submit?
作成中
引用
Expansion of the Human Phenotype Ontology (HPO) knowledge base and resources
Sebastian Köhler Leigh Carmody, Nicole Vasilevsky, Julius O B Jacobsen, Daniel Danis, Jean-Philippe Gourdine, Michael Gargano, Nomi L Harris, Nicolas Matentzoglu, Julie A McMurry, David Osumi-Sutherland, Valentina Cipriani, James P Balhoff, Tom Conlin, Hannah Blau, Gareth Baynam, Richard Palmer, Dylan Gratian, Hugh Dawkins, Michael Segal, Anna C Jansen Ahmed Muaz, Willie H Chang, Jenna Bergerson, Stanley J F Laulederkind, Zafer Yüksel, Sergi Beltran Alexandra F Freeman, Panagiotis I Sergouniotis, Daniel Durkin, Andrea L Storm Marc Hanauer Michael Brudno, Susan M Bello Murat Sincan Kayli Rageth Matthew T Wheeler Renske Oegema Halima Lourghi Maria G Della Rocca Rachel Thompson Francisco Castellanos, James Priest Charlotte Cunningham-Rundles Ayushi Hegde, Ruth C Lovering, Catherine Hajek Annie Olry Luigi Notarangelo, Morgan Similuk, Xingmin A Zhang, David Gómez-Andrés, Hanns Lochmüller,, Hélène Dollfus, Sergio Rosenzweig, Shruti Marwaha Ana Rath Kathleen Sullivan, Cynthia Smith Joshua D Milner, Dorothée Leroux, Cornelius F Boerkoel Amy Klion, Melody C Carter, Tudor Groza, Damian Smedley, Melissa A Haendel Chris Mungall, and Peter N Robinson
Nucleic Acids Res. 2019 Jan 8; 47(Database issue): D1018–D1027
ref.2
Improved exome prioritization of disease genes through cross-species phenotype comparison
Robinson PN, Köhler S, Oellrich A; Sanger Mouse Genetics Project, Wang K, Mungall CJ, Lewis SE, Washington N, Bauer S, Seelow D, Krawi
tz P, Gilissen C, Haendel M, Smedley D
Genome Res. 2014 Feb;24(2):340-8. Epub 2013 Oct 25.
参考
https://www.megabank.tohoku.ac.jp/news/18436