MicroRNA(miRNA)は、18〜25ヌクレオチドの小さな非コーディングRNAであり、動物と植物の両方で転写後レベルの中心的な調節因子である。 miRNAとそのターゲットmRNAの完全またはほぼ完全な相補的結合は、mRNAの分解を促進したり、mRNAの翻訳を抑制したりして、遺伝子発現を負に制御する(ref.1)。 miRNAは、細胞周期(ref.2)、細胞分化(ref.3)、アポトーシス(ref.4)などの多くの生物学的プロセス、およびガンを含むさまざまな疾患(ref.5)に関与している。 miRNAとその標的相互作用の研究は、疾患の発症との因果関係により研究者の間で関心を集めている。重要なことに、miRNAの調節不全の発現パターンは、疾患の診断と予後の潜在的な バイオマーカーとして役立つ可能性がある(ref.6)。
miRNA-ターゲット相互作用(MTI)を分析および識別するための多くのWebベースのmiRNA関連データベースが確立されている。これらのうち、miRBase(ref.5)は、包括的なmiRNAの命名法、配列、およびアノテーションデータの検索を容易にする主要なmiRNA配列リポジトリである。 DIANA-TarBase(ref.6)は、詳細なメタデータ、実験方法論、および条件を備えた、手動でキュレーションされた実験的に検証されたmiRNA-遺伝子相互作用を含むデータベースである。 HMDD(ref.7)およびmiR2Disease(ref.8)には、実験的に検証されたヒトmiRNAおよび疾患関連MTIが含まれる。 MTIデータベースは広く利用可能だが、MTI関連の研究は近年多く発表されている。したがって、実験的に検証されたmiRNA-ターゲット相互作用の集中情報リポジトリは、簡単にアクセスでき、長期にわたって更新される必要がある。
miRNAとそのターゲットの塩基対に依存する計算アプローチは、miRNAとそのターゲット結合部位の識別に役立つが(ref.9)、完全なシードペアリングは、必ずしもmiRNAとそのターゲット間の実際の相互作用を表すとは限らない(ref.10)。したがって、miRNAとその標的相互作用を確立して、特定のmiRNAの機能を解明するために、実験的検証を実施する必要がある。 MTIはいくつかの方法で実験的に検証できる(ref.11)。 mRNAの発現レベルはqRT-PCRなどの手法を使用して検出でき、タンパク質産物はELISA、免疫組織化学、ウエスタンブロットで測定できる。レポーターアッセイは、活性の低下やレポータータンパク質の発現を測定することにより、miRNAとそのターゲット間の直接的な相互作用を実証する最も信頼できる方法である(ref.12)。マイクロアレイやハイスループットシーケンシングなどの方法は、miRNAとそのターゲット間の間接的な関係を提供する(ref.13)。 CLIP(ref.14)、PAR-CLIP、(ref.15)、およびCLASH(ref.16)などの新しく開発された手法には、miRNAとそのターゲットを識別するためにも使用できるハイスループットシーケンスが組み込まれている。
最近、miRNA-ターゲット相互作用関連の研究が急速に増加している(論文図S1)。 miRNAターゲットの識別とさまざまな生物学的プロセスでの役割を強化するために、以前にmiRTarBaseを開発した(ref.17–19)。現在まで、ターゲット予測の正確なレートの改善、miRNA関連データベースまたはWebベースのツールの統合、さまざまな疾患のmiRNA遺伝子調節ネットワークの再構築、およびCancer Genome Atlas(TCGA)への組み込みなど広く使用されている。ここでは、生物学的調査、ネットワークベースの研究、計算機学習アプリケーションを促進するために、改良されたアノテーションと視覚化データを備えた実験的に検証されたMTIの最新のリポジトリを提供する。
Integbioデータベースカタログ
miRTarBase - Integbio データベースカタログ
help
http://mirtarbase.mbc.nctu.edu.tw/php/help.php
生物ごとのmiRNAターゲット相互作用情報数要約(ヒト、マウス、線虫が大半)
http://mirtarbase.mbc.nctu.edu.tw/php/statistics.php
使い方
http://mirtarbase.mbc.nctu.edu.tw/php/index.php にアクセスする。

Search
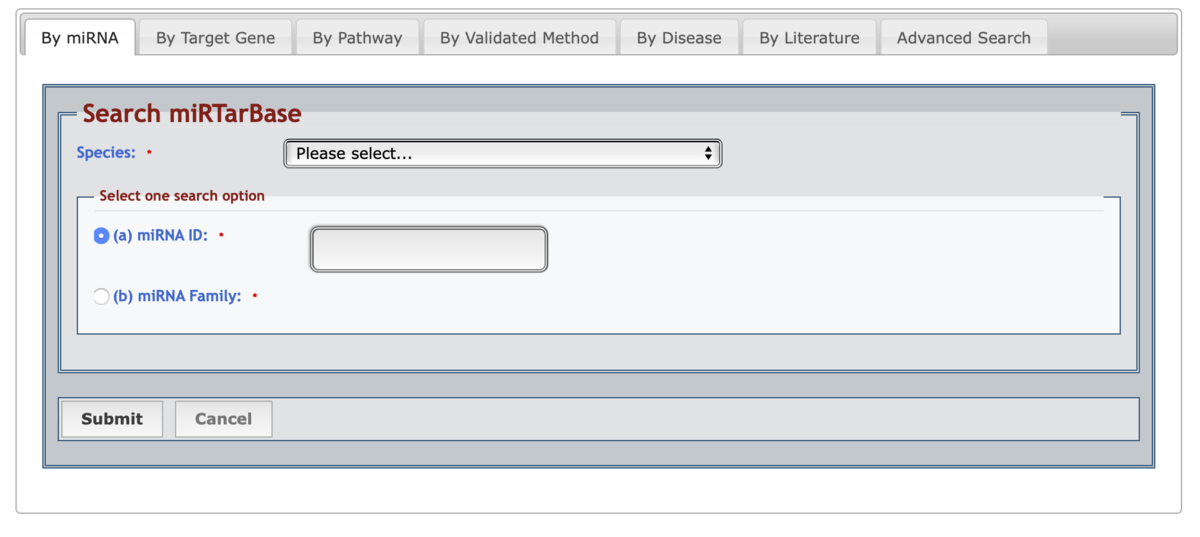
miRNAのID、miRNAのターゲット遺伝子名、パスウェイ名、ウエットの実験からのエビデンス、疾患、文献など様々な方法で検索できる。
http://mirtarbase.mbc.nctu.edu.tw/php/search.php#mirna
ターゲット遺伝子CDK6、データベースはヒトで検索した。
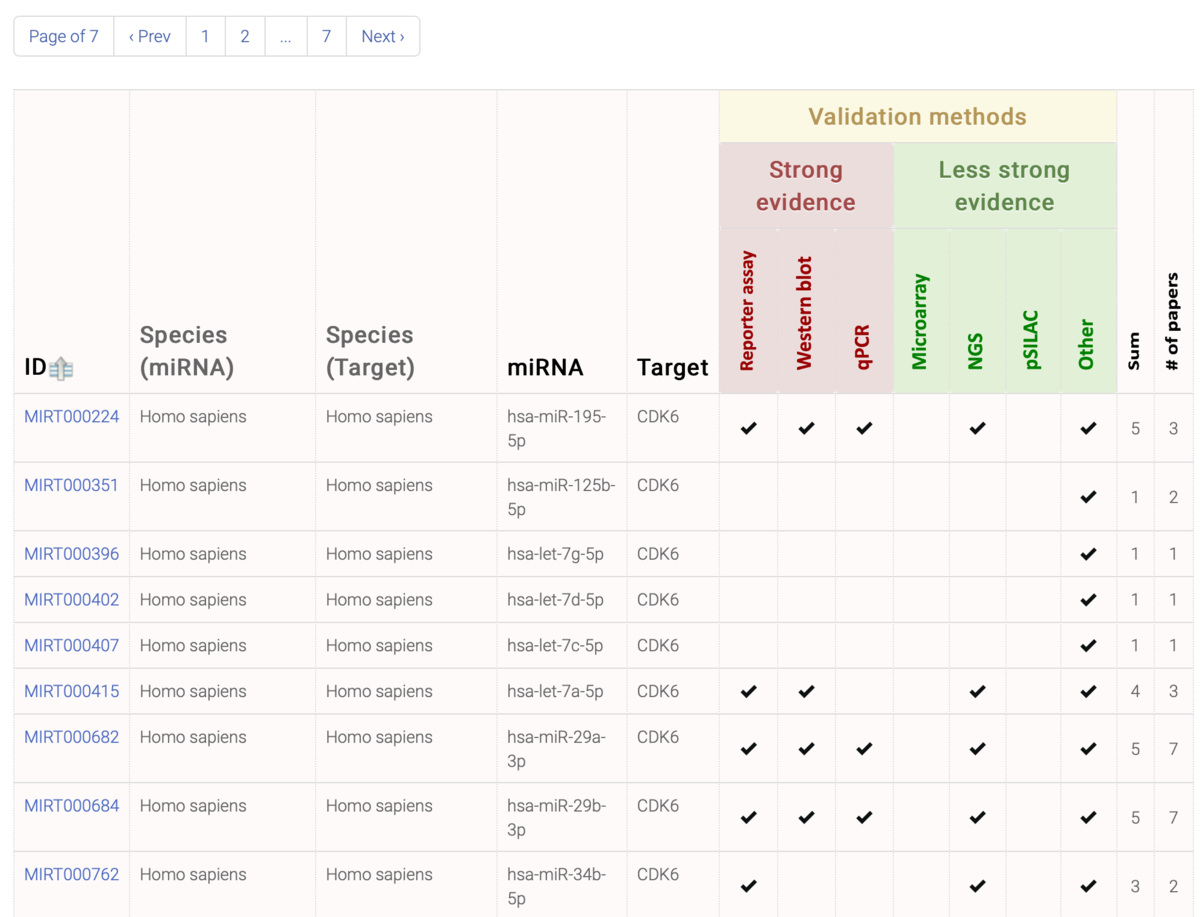
miRNAのID、miRNA とターゲットの種名、miRNA名、ターゲット遺伝子名、実験のエビデンスの有無、などが表形式で表示される。
id:MIRT000224を開いた。
左端のmiRNAタブ

miRNAの二次構造のほか、ワードクラウドで関係する疾患が視覚化されている。
Target geneタブ
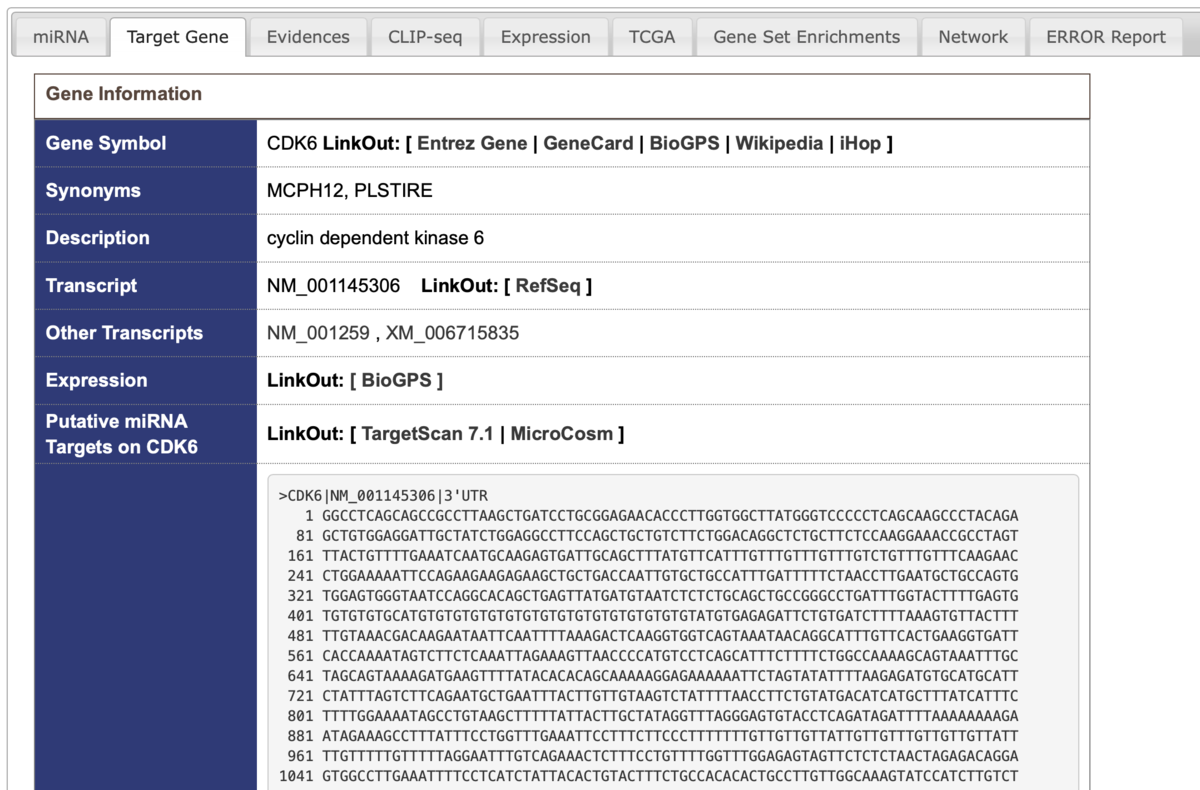
Evidencesタブ

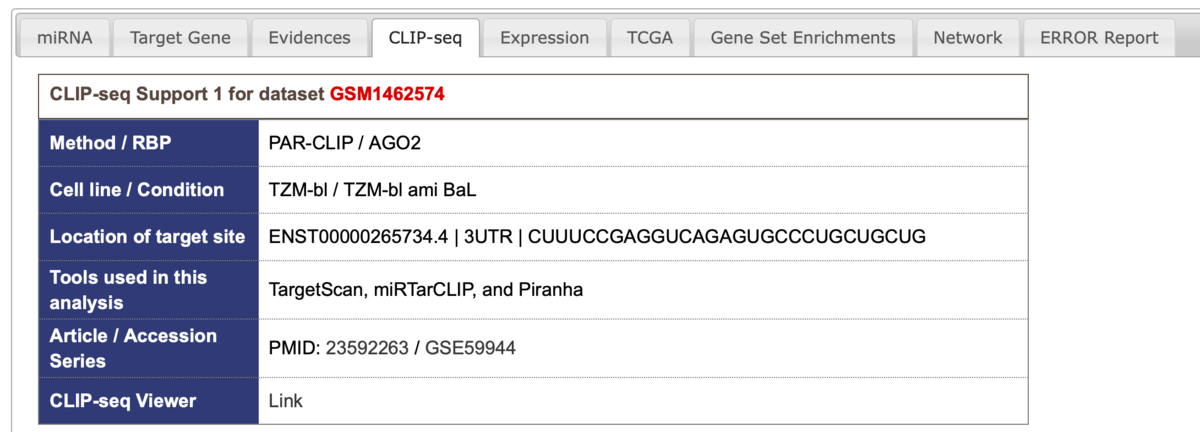
CLIP-seqタブ
Expressionタブ
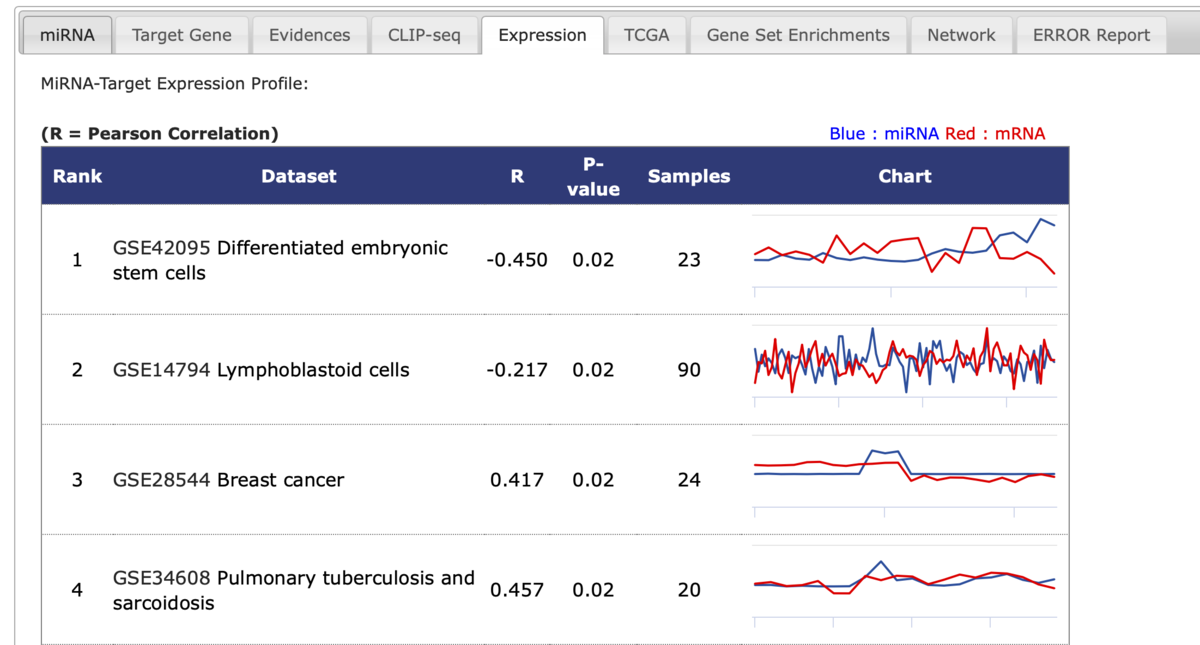
ピアソンの相関係数とP値付きでデータセットとの相関が示されている。
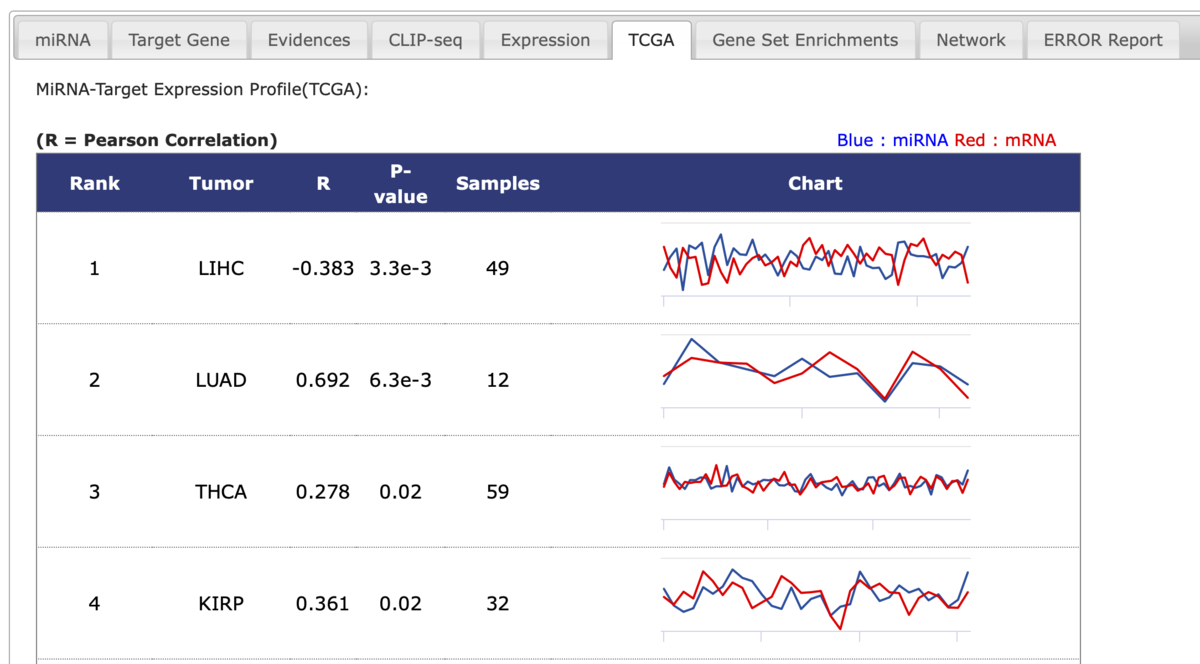
TCGA: MiRNA-Target Expression Profile
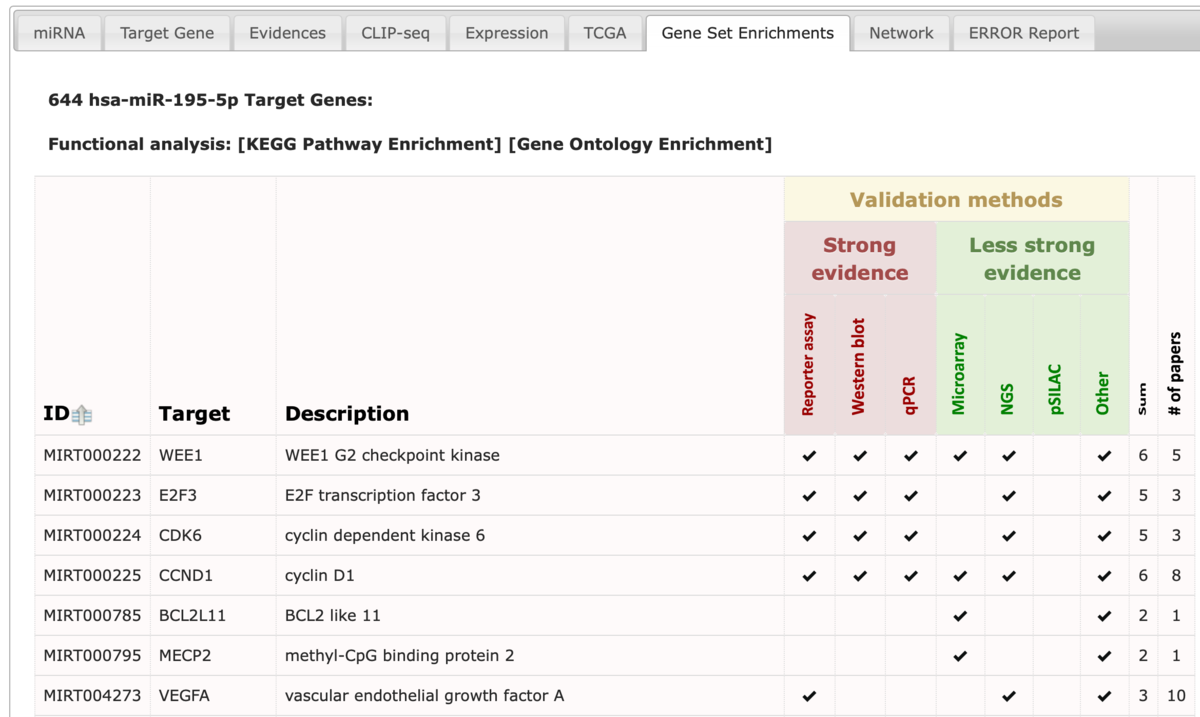
Gene Set Enrichments
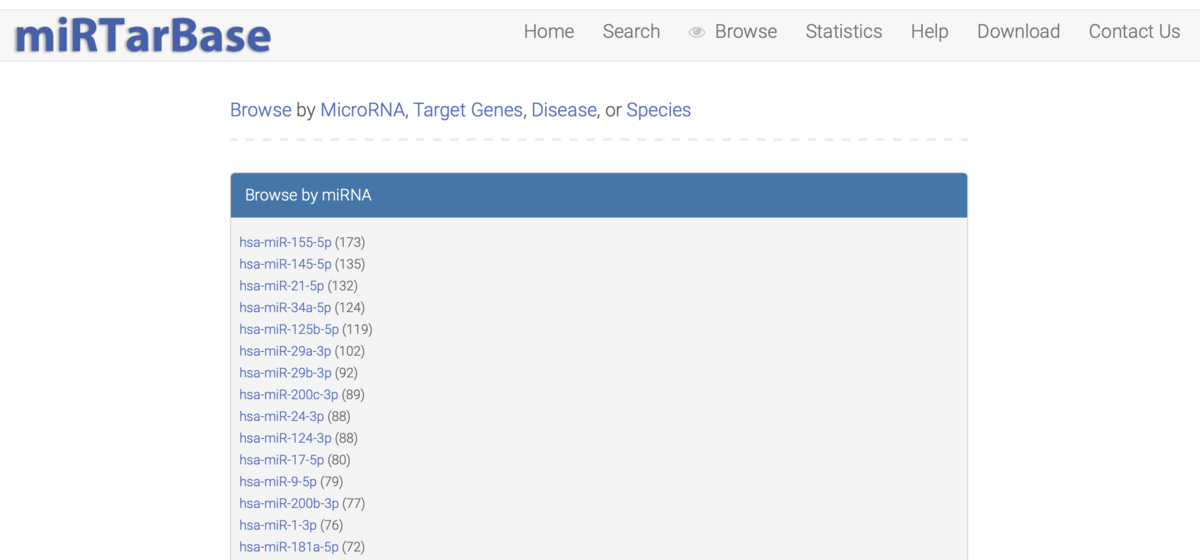
BrowseではmiRNA名や疾患などから検索できる。
引用
miRTarBase update 2018: a resource for experimentally validated microRNA-target interactions
Chou CH, Shrestha S, Yang CD, Chang NW, Lin YL, Liao KW, Huang WC, Sun TH, Tu SJ, Lee WH, Chiew MY, Tai CS, Wei TY, Tsai TR, Huang HT, Wang CY, Wu HY, Ho SY, Chen PR, Chuang CH, Hsieh PJ, Wu YS, Chen WL, Li MJ, Wu YC, Huang XY, Ng FL, Buddhakosai W, Huang PC, Lan KC, Huang CY, Weng SL, Cheng YN, Liang C, Hsu WL, Huang HD
Nucleic Acids Res. 2018 Jan 4;46(D1):D296-D302