2019 9/12 誤字修正、おかしな文章削除
ハイスループット研究からの遺伝子リストの解釈には、最新のデータに基づいた有能で便利なツールが必要である。 Enrichr(ref.1)、WebGestalt(ref.2)、Metascape(ref.3)、KOBAS(ref.4)、AgriGO(ref.5)などのfunctional enrichment analysisツールがいくつか存在する。これらのツールのランドスケープは、さまざまなデータソース、種、識別子の種類、および方法をカバーする多様なものである。サービスの大半は、最も広く使用されているナリッジリソースGene Ontology(GO)(ref.6)のマッピングを提供するが、他のデータソースの選択はツールによって異なる。たとえば、Human Phenotype Ontology(ref.7)はEnrichr、WebGestalt、Metascape、およびg:Profiler(ref.1–3,8)で使用できるが、mirTarBase miRNAターゲット情報はEnrichrおよびg:Profiler(ref.1,8)などのいくつかのツールにのみ含まれる。特定の種に焦点を当てたサービスもある。 AgriGOは主に植物に関するデータを提供する(ref.5)。
これらのツールは、さまざまな技術プラットフォームに実装されている。たとえば、WebGestaltには有名なWebサーバー(ref.2)があり、GSEAはスタンドアロンアプリケーション(ref.9)で知られる。Enrichrは、Webサービスに加えてRパッケージ(ref.1)もある。他のツールは、さまざまな技術プラットフォームにわたってユーザーにサービスを提供している。たとえば、g:Profilerは、WebクライアントとAPI、Python、Rパッケージを介してユーザーにサービスを提供し、Galaxyプラットフォームのツールとして利用できる(ref.10)。
Functional enrichmentツールの入力遺伝子リストは、幅広い実験プラットフォームに由来し、それぞれがデフォルトでサポートされる一意の識別子タイプを持っている。ほとんどのツールは、可能な識別子の限られたサブセットのみを受け入れるため、ユーザーが外部ツールを介して克服する必要がある障害が存在する。 g:Profilerはこのハードルを回避でき、同じクエリで混在する可能性のある100種類近い識別子タイプを自動的に検出して受け入れる。このネームマッピング機能は、いくつかのツールへの相互運用機能としてすでに組み込まれて、独立したg:Convertサービスとしても提供されている(ref.11–13)。
Functional enrichment analysisに使用される方法は、ツールによって異なる。EnrichrおよびWebGestalt(ref.1,2)と同様、g:Profilerは、超幾何テストを使用して入力遺伝子リストのFunctional termの有意性を測定する、最も広く使用されているover-representation解析アプローチを提供している。遺伝子リストの追加ランキング情報を考慮に入れる他の方法を提供するツールがある(WebGestalt、GSEA(ref.2,9))、または遺伝子調節ネットワークからの事前知識を使用する(WebGestalt(ref.2))。これらの方法にはすべて独自の制限があり、さまざまな方法を評価および比較するための優れたベンチマークデータがない(ref.14)。ユーザーに使いやすく高速なツールを提供するために、g:Profilerは1つのアプローチのみに焦点を合わせている。
Enrichment analysis専用のツールのうち、最初のリリース後に継続的かつ最新のサービスを提供しているのはごくわずかしかない。 g:Profilerは、2007 NAR Webサーバーの問題で最初に公開されて以来、非常に重要であり、2011年と2016年に更新論文を出している(ref.8、15、16)。さまざまな科学分野の研究者を継続的にサポートするために、サポートされる種と遺伝子識別子の種類を増やし、データ更新頻度、プログラム可能なアクセス、コアの高品質データソースを年間を通じて安定的に維持している(論文図1)。基礎となるデータの複雑さとサイズが増大するにつれて、g:Profilerの完全な技術的書き換えを導入した。これにより、最新のユーザーインターフェースとプログラミングインターフェースを介してユーザーに迅速かつ便利にサービスを提供できるだけでなく、機能を追加して安定したサービスを維持するための新しい道を開く。
FAQ
https://biit.cs.ut.ee/gprofiler/page/faq
List of organisms
https://biit.cs.ut.ee/gprofiler/page/organism-list
使い方
https://biit.cs.ut.ee/gprofiler/gost にアクセスする。
1、 g:GOSt - 入力遺伝子リストからfunctional enrichment analysisを実行
g:GOStは500近い生物をサポートし、数百の識別子タイプを受け入れる。遺伝子を既知の機能情報源にマッピングし、統計的に有意に豊富な用語を検出する。 Ensemblデータベースと菌類、Ensembl Genomesの植物または後生動物、およびWormBase ParaSiteの寄生虫固有のデータから定期的にデータを取得している。 Gene Ontologyに加えて、KEGG、 Reactome、WikiPathways、 miRTarBaseのmiRNAターゲットとTRANSFACの規制モチーフマッチからのパスウェイ、 Human Protein Atlasの組織特異性、CORUMからのタンパク質複合体およびHuman Phenotype Ontologyからのヒト疾患表現型、が含まれる。
デフォルトでは、g:GOStはアノテーション付きのすべてのタンパク質コーディング遺伝子のセットをバックグラウンドとして使用する。一部の実験では、遺伝子またはタンパク質のサブセットを使う。例えば、疾患特異的遺伝子のみのターゲットシーケンスではカスタムバックグラウンド情報を使用することが推奨される。
遺伝子リストをウィンドウ内にペーストするか、アップロードする。

ラン前に右の設定を確認する。まず生物種を選ぶ。

代表的なモデル生物の他、Ensemblの様々な生物をサポートしている。
様々なフォーマットをサポートしている。Gene OntologyのIDの他、遺伝子名、領域指定、dbSNPのIDも使える。

データソースを選択する。GOの他、KEGG 、Reactome、Wikipathway、Human Protein Atlasなど選択できる。

選べるデータソースは生物によって変わる。

上のリストにないカスタムアノテーションから絞り込んだリストを使う場合、pathway IDとGO termなどの関係を示したGMT formatのファイルをアップロードする。

結果はマンハッタンプロットで示される。

図の他、遺伝子とtermに関する詳細な情報を示したテーブルも出力される。
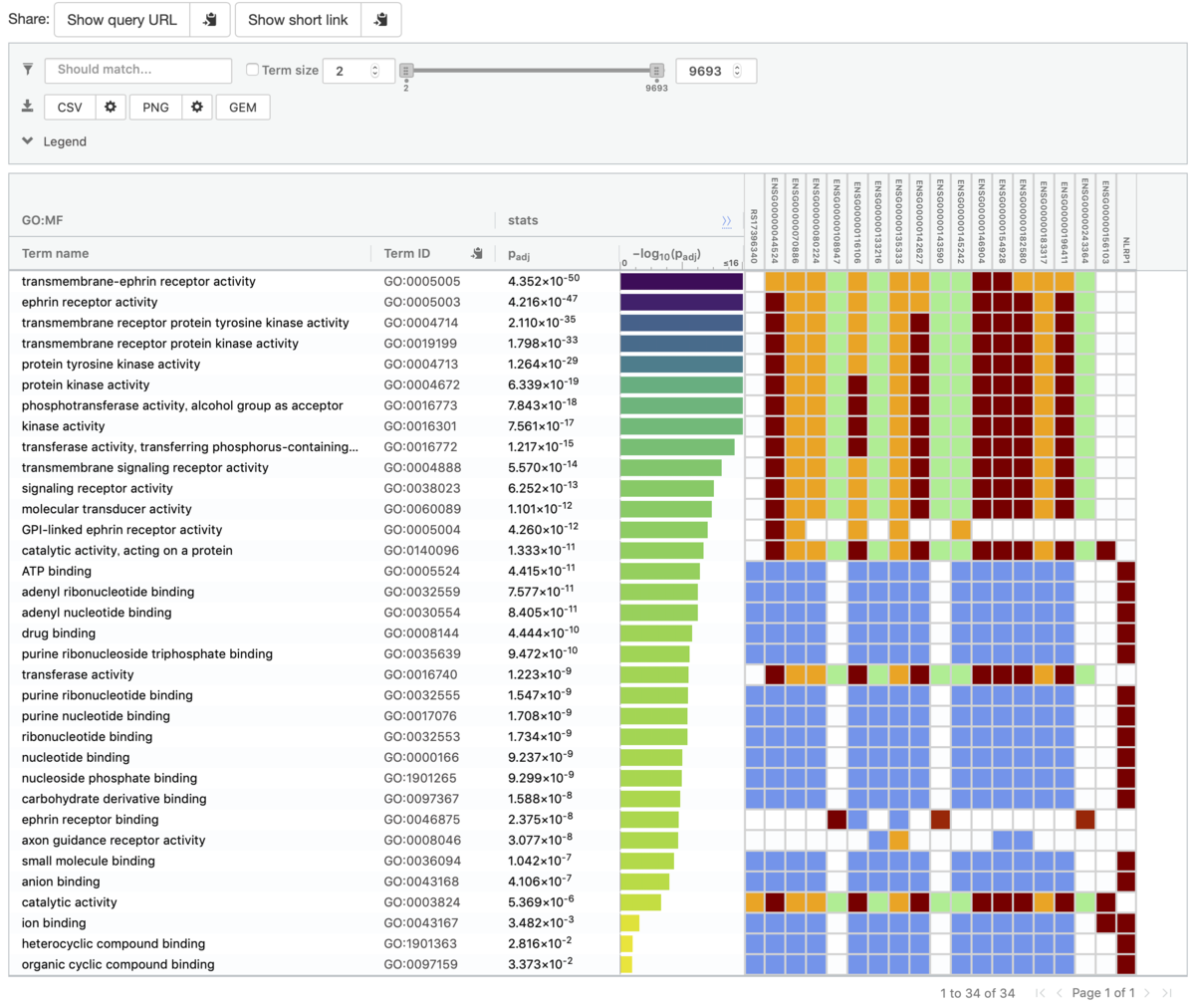

図と表はダウンロードできる。
2、 g:Convert – 遺伝子識別子の自動変換と詳細の表示
さまざまな遺伝子、タンパク質、マイクロアレイプローブ、および他の多くを変換できる。Ensembl Biomartを使う。ヒトでは98種類の識別子に対応している。

複数混合していても対応する。
結果はCSVとしてダウンロードできる。

3、g:Orth – 生物種間でオルソログ遺伝子をマッピング
入力遺伝子リストに対応するターゲット生物種のオルソログ遺伝子を自動的に取得する。マッピングは、ユーザーが入力した入力遺伝子IDをEnsembl ENSG識別子に最初に変換し、次に対象種の対応するオルソロガスな遺伝子情報を取得することにより、2段階で実行される。
クエリの生物種、ターゲットの生物種を指定する。

よく研究されたモデル生物について収集された知識を、あまり研究されていない種に転送するために使用できる。例えばオルソログマッピング後にエンリッチメント解析を実行すると、元の種のみを使用した場合よりもわかりやすい結果が得られる場合があるとされる。
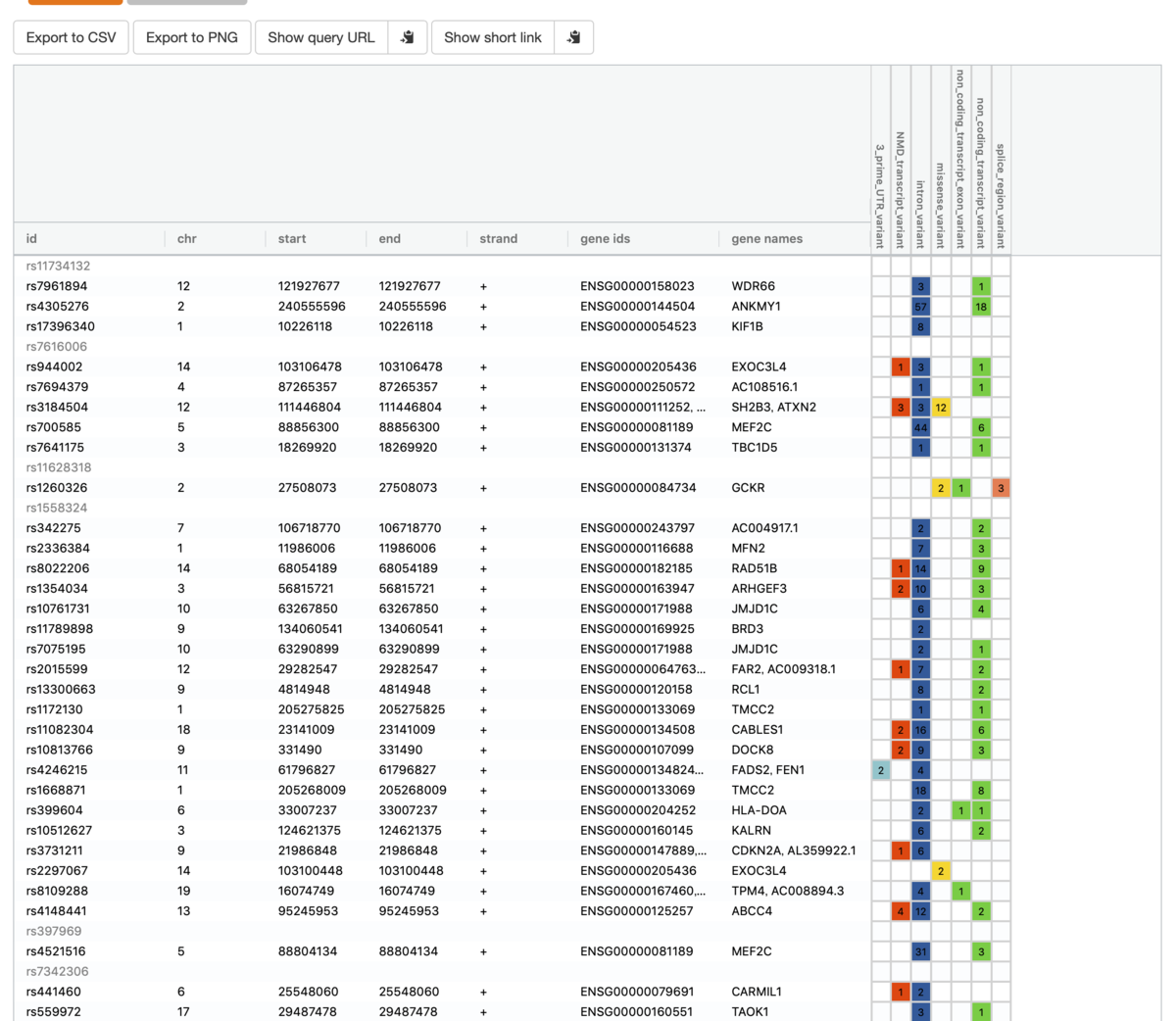
4、g:SNPense – SNP識別子マッピング
human SNP rsコード(rs7961894など)を遺伝子名に簡単にマップし、その染色体座標と予測される変異効果を取得できる。すべての基礎となるデータは、Ensembl Variation Dataから取得される。

バリアントエフェクトは、シーケンスオントロジーで定義されている、色分けされたバリアント結果のtermセットで説明される。

出力例
g:Profilerはユーザーがアップロードしたカスタム GMT ファイルもサポートしています。これにより、g:Profiler はあらゆる生物のデータを分析することができます。
2020 2/1追記
統合TVの解説
https://togotv.dbcls.jp/20200127.html
引用
g:Profiler: a web server for functional enrichment analysis and conversions of gene lists (2019 update)
Uku Raudvere,1 Liis Kolberg,1 Ivan Kuzmin,1 Tambet Arak,1 Priit Adler,1,2 Hedi Peterson,1,2and Jaak Vilo
Nucleic Acids Res. 2019 Jul 2; 47(W1): W191–W198
関連

