2019 10/15 誤字修正
2020 7/27 help更新
2020 8/14 コマンド追記
2020 12/25 誤字修正
多くのバイオインフォマティクスプログラムは、遺伝子および転写産物をGFF形式(General Feature Format)で表し、ゲノム上の遺伝子および転写産物の特徴(染色体またはscaffolds/contigs)の位置と属性を簡単に説明する。GFFには多くのバージョンがあるが、最も一般的なのはGTF2(Gene Transfer Format、ここで説明)とGFF3(ここで定義)である。これらの形式がStringTieおよびその他のバイオインフォマティクスプログラムによって解釈される方法に関する詳細を次に示す。
GTFファイル
GTF2仕様で見られるように、transcript_id属性は必須である。したがって、GFFパーサーもそれを想定している。一方、gene_id属性は、GFFパーサーでは厳密には必要ないが、遺伝子/遺伝子座識別子の下でオルタナティブな転写産物をグループ化するのに非常に役立つ。オプションのgene_name属性が見つかった場合は、シンボリック遺伝子名または短縮形の略語(HGNCまたはEntrez Geneの遺伝子記号など)として取得および表示される。一部のアノテーションソース(Ensemblなど)は、HUGOシンボルのようにgene_name属性に「人間が読み取れる」遺伝子名/シンボルを配置する(gene_idは、遺伝子に対して自動的に生成される単なる数値識別子の場合がある)。
通常、ほとんどのバイオインフォマティクスプログラムは、少なくとも転写構造全体を定義するのに十分なエクソン機能と、コーディングセグメントを指定するオプションのCDS機能を期待している。 GFFリーダーは、CDS機能全体が提供されている場合、start_codon、stop_codonなどの冗長機能を無視し、同様に、エクソン機能全体が提供されている場合、UTR機能は無視される。ただし、CDSおよび* UTR機能のみを提供することは依然として可能であり、GFFパーサーはそれに応じてエクソン構造を再構築する(これらのセグメントをエクソンセグメントに内部的に変換する)。StringTieやCufflinksなどのプログラムのGTF出力には、エクソンの親機能として機能する追加のトランスクリプト機能行と、トランスクリプト構造を定義し、同じtranscript_id属性を持つCDS機能もある。 これはGTF2仕様では必要ない。
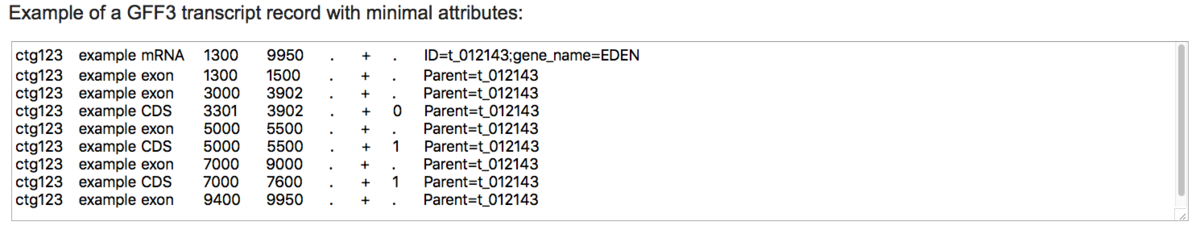
GFF3ファイル
GFF3仕様で定義されているように、親機能(通常は転写物、つまり「mRNA」機能)にはID属性が必要だが、ここでもオプションのgene_name属性を使用して、共通の遺伝子名の省略形を指定できる。 gene_nameが指定されていない場合、現在の親mRNAフィーチャの親遺伝子フィーチャのName属性またはID属性から推測することもできる(入力ファイルで指定されている場合)。 CDSおよびエクソン機能には、親トランスクリプト機能(通常は「mRNA」機能)のID属性の値と値が一致する親属性が必要である。
Example of a GTF2 transcript record with minimal attributes: (HPより転載)
プログラムgffreadを使用して、GFFファイルのさまざまな操作を検証、フィルター、変換、および実行できる。 プログラムはCufflinks、Stringtie、およびgffcompareと同じGFFパーサーコードを共有するため、特定のアノテーションソースからのGFFファイルがこれらのプログラムによって正しく「理解」されていることを確認するために使用できる。 したがって、gffreadユーティリティを使用して、ファイルからトランスクリプトを単純に読み取り、オプションでこれらのトランスクリプトをGFF3(デフォルト)またはGTF2形式(-Tオプション付き)でプリントし、必要でない属性を破棄し、オプションで修正することができる。
既にいくつか日本語の説明記事がありますが、このブログでも登場することが何度かあったので、簡単に使い方をまとめておきます。
HP
http://ccb.jhu.edu/software/stringtie/gff.shtml
インストール
依存
#bioconda (link)
mamba install -c bioconda -y gffread
> gffread -h
$ gffread -h
gffread v0.11.6. Usage:
gffread <input_gff> [-g <genomic_seqs_fasta> | <dir>][-s <seq_info.fsize>]
[-o <outfile>] [-t <trackname>] [-r [[<strand>]<chr>:]<start>..<end> [-R]]
[-CTVNJMKQAFPGUBHZWTOLE] [-w <exons.fa>] [-x <cds.fa>] [-y <tr_cds.fa>]
[-i <maxintron>] [--bed] [--table <attrlist>] [--sort-by <refseq_list.txt>]
Filter, convert or cluster GFF/GTF/BED records, extract the sequence of
transcripts (exon or CDS) and more.
By default (i.e. without -O) only transcripts are processed, discarding any
other non-transcript features. Default output is a simplified GFF3 with only
the basic attributes.
<input_gff> is a GFF file, use '-' for stdin
Options:
-i discard transcripts having an intron larger than <maxintron>
-l discard transcripts shorter than <minlen> bases
-r only show transcripts overlapping coordinate range <start>..<end>
(on chromosome/contig <chr>, strand <strand> if provided)
-R for -r option, discard all transcripts that are not fully
contained within the given range
-U discard single-exon transcripts
-C coding only: discard mRNAs that have no CDS features
--nc non-coding only: discard mRNAs that have CDS features
--ignore-locus : discard locus features and attributes found in the input
-A use the description field from <seq_info.fsize> and add it
as the value for a 'descr' attribute to the GFF record
-s <seq_info.fsize> is a tab-delimited file providing this info
for each of the mapped sequences:
<seq-name> <seq-length> <seq-description>
(useful for -A option with mRNA/EST/protein mappings)
Sorting: (by default, chromosomes are kept in the order they were found)
--sort-alpha : chromosomes (reference sequences) are sorted alphabetically
--sort-by : sort the reference sequences by the order in which their
names are given in the <refseq.lst> file
Misc options:
-F preserve all GFF attributes (for non-exon features)
--keep-exon-attrs : for -F option, do not attempt to reduce redundant
exon/CDS attributes
-G do not keep exon attributes, move them to the transcript feature
(for GFF3 output)
--keep-genes : in transcript-only mode (default), also preserve gene records
--keep-comments: for GFF3 input/output, try to preserve comments
-O process other non-transcript GFF records (by default non-transcript
records are ignored)
-V discard any mRNAs with CDS having in-frame stop codons (requires -g)
-H for -V option, check and adjust the starting CDS phase
if the original phase leads to a translation with an
in-frame stop codon
-B for -V option, single-exon transcripts are also checked on the
opposite strand (requires -g)
-P add transcript level GFF attributes about the coding status of each
transcript, including partialness or in-frame stop codons (requires -g)
--add-hasCDS : add a "hasCDS" attribute with value "true" for transcripts
that have CDS features
--adj-stop stop codon adjustment: enables -P and performs automatic
adjustment of the CDS stop coordinate if premature or downstream
-N discard multi-exon mRNAs that have any intron with a non-canonical
splice site consensus (i.e. not GT-AG, GC-AG or AT-AC)
-J discard any mRNAs that either lack initial START codon
or the terminal STOP codon, or have an in-frame stop codon
(i.e. only print mRNAs with a complete CDS)
--no-pseudo: filter out records matching the 'pseudo' keyword
--in-bed: input should be parsed as BED format (automatic if the input
filename ends with .bed*)
--in-tlf: input GFF-like one-line-per-transcript format without exon/CDS
features (see --tlf option below); automatic if the input
filename ends with .tlf)
Clustering:
-M/--merge : cluster the input transcripts into loci, discarding
"duplicated" transcripts (those with the same exact introns
and fully contained or equal boundaries)
-d <dupinfo> : for -M option, write duplication info to file <dupinfo>
--cluster-only: same as -M/--merge but without discarding any of the
"duplicate" transcripts, only create "locus" features
-K for -M option: also discard as redundant the shorter, fully contained
transcripts (intron chains matching a part of the container)
-Q for -M option, no longer require boundary containment when assessing
redundancy (can be combined with -K); only introns have to match for
multi-exon transcripts, and >=80% overlap for single-exon transcripts
-Y for -M option, enforce -Q but also discard overlapping single-exon
transcripts, even on the opposite strand (can be combined with -K)
Output options:
--force-exons: make sure that the lowest level GFF features are considered
"exon" features
--gene2exon: for single-line genes not parenting any transcripts, add an
exon feature spanning the entire gene (treat it as a transcript)
--t-adopt: try to find a parent gene overlapping/containing a transcript
that does not have any explicit gene Parent
-D decode url encoded characters within attributes
-Z merge very close exons into a single exon (when intron size<4)
-g full path to a multi-fasta file with the genomic sequences
for all input mappings, OR a directory with single-fasta files
(one per genomic sequence, with file names matching sequence names)
-w write a fasta file with spliced exons for each transcript
--w-add <N> for the -w option, extract additional <N> bases
both upstream and downstream of the transcript boundaries
-x write a fasta file with spliced CDS for each GFF transcript
-y write a protein fasta file with the translation of CDS for each record
-W for -w and -x options, write in the FASTA defline the exon
coordinates projected onto the spliced sequence;
for -y option, write transcript attributes in the FASTA defline
-S for -y option, use '*' instead of '.' as stop codon translation
-L Ensembl GTF to GFF3 conversion (implies -F; should be used with -m)
-m <chr_replace> is a name mapping table for converting reference
sequence names, having this 2-column format:
<original_ref_ID> <new_ref_ID>
-t use <trackname> in the 2nd column of each GFF/GTF output line
-o write the records into <outfile> instead of stdout
-T main output will be GTF instead of GFF3
--bed output records in BED format instead of default GFF3
--tlf output "transcript line format" which is like GFF
but exons, CDS features and related data are stored as GFF
attributes in the transcript feature line, like this:
exoncount=N;exons=<exons>;CDSphase=<N>;CDS=<CDScoords>
<exons> is a comma-delimited list of exon_start-exon_end coordinates;
<CDScoords> is CDS_start:CDS_end coordinates or a list like <exons>
--table output a simple tab delimited format instead of GFF, with columns
having the values of GFF attributes given in <attrlist>; special
pseudo-attributes (prefixed by @) are recognized:
@chr, @start, @end, @strand, @numexons, @exons, @cds, @covlen, @cdslen
-v,-E expose (warn about) duplicate transcript IDs and other potential
problems with the given GFF/GTF records
使い方
転写領域をGTFファイルで指定して(transcripts.gtf)、ゲノムのfasta (-g genome.fa)から転写領域の配列を取り出す(-w transcripts.fa)。
gffread transcripts.gtf -g genome.fa -w transcripts.fa
- -w write a fasta file with spliced exons for each GFF transcript
-
-g full path to a multi-fasta file with the genomic sequences for all input mappings, OR a directory with single-fasta files
- -o write the records into <outfile> instead of stdout
samtools faidx genome.faしてgenome.faのindexを作成し、次のラン時に-gフラグを立てると、上記のコマンドは高速化できる。
GTFからGFF3への変換。
gffread transcripts.gtf -g genome.fa -E -o transcripts.gff3
-
-E expose (warn about) duplicate transcript IDs and other potential problems with the given GFF/GTF record
-Eオプションは、gffreadに、入力ファイルの解析中に発生した潜在的な問題の警告を表示するために使う。
GFF3からGTFへの変換。
gffread transcripts.gff3 -g genome.fa -E -T -o transcripts.gtf
- -T main output will be GTF instead of GFF3
GTF/GFF3からBEDへの変換(バージョンによっては不可)。
gffread transcripts.gtf -g genome.fa -E --bed -o transcripts.bed
- --bed output records in BED format instead of default GFF3
出力でなく入力がBEDなら”--in-bed”をつける。
追記
exon配列(-w)、cDNA配列(-w)とタンパク質配列(-y)の抽出
gffread annotation.gff3 -g genome.fa -w transcripts.fa -x cds.fa -y protein.fa
- -w write a fasta file with spliced exons for each transcript
- -x write a fasta file with spliced CDS for each GFF transcript
- -y write a protein fasta file with the translation of CDS for each record
指定領域の抽出、ソート、duplicated idのフィルタリングなども行うことができる。
5kbより短いコンティグのアノテーションを捨てる。
#1 まずgenome(scaffolds.fa)を長さでソート、5Kb以上だけを保存
seqkit sort --quiet -r -l scaffolds.fa -L 5000 > scaffolds_sorted.fa
#2 indexingしてリード長を出力
samtools faidx scaffolds_sorted.fa
cut -f 2 scaffolds_sorted.fa > list
#3 gffreadでGFF3=>GTF変換、listファイル順にアノテーション出力。
gffread transcripts.gff3 -g genome.fa -E -T --sort-by list -o transcripts.gtf
#4 transcripts.gtfを開き、listの最後に出てくる配列より下のアノテーションを消す
引用
http://ccb.jhu.edu/software/stringtie/gff.shtml
参考
gffread を使った transcripts fasta 転写物の配列取得
http://bioinfo-dojo.net/2018/01/09/gffread_transcripts-fasta/
gffreadの使い方
https://home.hiroshima-u.ac.jp/~tigawa/?p=924
関連
paftools
GTF/GFF3をBED12フォーマットに変換したり、SAMをBEDに変換するなど。