2019 version2.4.1のヘルプに更新、論文追記、テストランのコマンドミス修正、リンク追加、コマンド修正、補足、リンク追加、You tube動画追加
2020 ツイート追加、help更新、例追記、コメント追記、ツイート追記
2021 5/8 動画リンク追加、6/16 subassembliesオプションのミス修正、8/24 ツイート追記
2022/04/15 バージョンアップに伴いコマンド修正
2023/02/12 追記
2024/04/13 インストール手順修正(conda-forge)
ゲノムアセンブリの問題は、最終的には、リピートキャラクタライゼーション問題、すなわちリピートグラフとしてのゲノム中のすべてのリピートファミリーをコンパクトに表現する(Pevzner et al。、2004)、ことに結びつく。 Long readの技術はリピートキャラクタライゼーション問題を無関係にしなかった。 代わりに、焦点が短いリピートから平均のSMSリードサイズに相当する長いリピートに移っただけだった:例えばKamath et al、2017は、多くのバクテリアゲノムを分析して、既存のSMSのアセンブラが単一コンティグにアセンブリできなかったことを示した。バクテリア(たとえば真核生物ではない)のゲノムも長いリピートを有するため、現在、SMSアセンブラは、ショートリードアセンブラが10年前に直面したのと同じ課題に直面している。このアセンブリの課題に取り組むために、SMS技術はcontact(Ghurye et al、2017 link)およびoptical(Weissensteiner et al、2017)マッピングデータによって補完されることが多い。
De Bruijnグラフはゲノムのリピートを全て表現できるエレガントな方法として登場し、ポピュラーなショートリードアセンブリアプローチとなっている。しかし、ほとんどのロングリードアセンブラは、リードのall-against-all の比較を必要とするoverlap-layout-consensus(OLC)アプローチを使用している。OLCアプローチはゲノム内のすべてのリピートの自然な表現を提供せず、最適なリピート解決を与えない (Koren et al., 2012, Chin et al., 2013, Berlin et al., 2015, Chin et al., 2016, Li, 2016, Koren et al., 2017)。
SMSアセンブリの最近の研究では、OLCのアプローチを別のde Bruijnグラフを構築して表現することを試みている(assembly graphと呼ばれる)。これらのアプローチはOLCのみのバクテリアのアセンブリを改善したが(Lin et al、2016、Kamath et al、2017)、エラーの多いSMSリードから構築されたde Bruijnグラフは非常にノイジーで、同じようなリピートをアセンブリグラフ内の単一のパスに折りたたむことを困難にしている。 OLCアプローチの開発者は、リピートキャラクタライゼーションの重要性を実証し、リピートを表現するためOLCベースのBogart グラフ(Koren et al、2017)を開発することにより、最良のオーバーラップグラフ(BOG)表現(Miller et al、2008)を改善しようとした。しかし、Bogartグラフはリピートグラフとは大きく異なり、Canuアセンブラ(Koren et al、2017)の開発者によって認められているように、それでもエラーが発生しやすい(Kamath et al、2017 de Bruijnベースの表現とOLCベースのリピート表現との間)。不必要な部分で詰まっているこれらのオーバーラップグラフの代わりに、stringグラフ(Meyers、2005)アプローチは(Miniasmアセンブラで実装された)、非分岐パスとしてゲノムのユニークなセグメントを表現しようとする。しかし、Kamath et al、2017で議論されているように、stringグラフも最適な repeat resolutionを達成しない。
エラーの多いロングリードからアセンブリグラフを作成する課題。ほとんどのショートリードアセンブラは、リードのすべてのk-mersに基づいてde Bruijnグラフを構成し、さらにさまざまなグラフの簡略化手順を使用してアセンブリグラフに変換する。このアプローチは、同じリピートの複数のインスタンスをアセンブリグラフの単一のパス(path)に折りたたみ、アセンブリグラフのedgeを回るゲノムツアーとしてゲノムを表す。しかし、SMSリードの場合、de Bruijn グラフアプローチの主要な前提(ゲノムのほとんどのk-merはたくさんのリードに存在している)は、長いk-merに対してはもちろん (e.g., k = 1000)、短いk-merでも成り立たない。結果として、ショートリードアセンブリで扱われた様々な問題(例えば、断片化されたde Bruijnグラフの処理方法、アセンブリグラフへの変換の仕方など)の多くは、SMSのde Bruijnグラフアプローチでは十分に議論されていない。
解決を繰り返すための積極的なアプローチは、しばしば誤ったアセンブリを生み出し、一方、保守的なアプローチは断片化されたアセンブリにつながる(Kamath et al、2017)。アセンブリグラフの構築は、積極的なアプローチと保守的なアプローチとの間のバランスを見出すための重要なステップである。これは、解決できないリピートと解決できるリピートを区別できうるからである。 SMSリードのためのアセンブリグラフを作成することは、様々なOLCアセンブラ(Kamath et al、2017)と比較して、繰り返しの分解能を向上させる。これは、その最適なバランスを見つけるのに適しているためである。また、アセンブリグラフは、ゲノム再構成研究(Lin et al、2014)に使用されるブレイクポイントグラフの特殊なケースを表すので、SMSリードを用いたゲノム変化の解析に適している(Chaisson et al、2015、Nattestad et al、 2017)。
ここでは、(HINGEのように)polishしたcontigからアセンブリグラフを作成するFlyeアルゴリズムについて説明する。これにより、グラフがより正確になる。 Flyeはまた、リピートコピー間の相違をなくすための新しいアルゴリズムを導入してHINGEを補完しており、リードがSMSリードがスパンしてブリッジすることができていないリピート間について、リピートのわずかな違いを見つけるアルゴリズムを導入している。FlyeはABruijnアセンブラ(Lin et al、2016 link)の上に構築されている。この方法は正確なオーバーラップありのcontigを構築するが、ゲノムのリピート構造を明らかにしない。 FlyeはABruijnとは対照的に、不正確なオーバーラッピングコンティグ(すなわち潜在的なアセンブリエラーを伴うコンティグ)を生成し、これらの初期コンティグを一致するすべての可能なアセンブリをコードする正確なアセンブリグラフに組み合わせる。 Flyeはさらに、アセンブリグラフの非ブリッジリピートを解決し、新しい、より絡み合っていないアセンブリグラフを構築し、最終的にこのグラフのパスによって形成される正確な最終コンティグを出力する。著者らは、様々なデータセットを使用していくつかの最先端のSMSアセンブラに対してFlyeをベンチマークし、正確なアセンブリを生成することを示すと同時に、アセンブリFinishに向けてどのような実験プランを取るべきか(e.g., using contact or optical maps)インサイトを提供する。
usage
https://github.com/fenderglass/Flye/blob/flye/docs/USAGE.md
Flyeに関するツイート
Flye 2.9 is out!
— Mikhail Kolmogorov (@fenderglass) 2021年8月21日
* Better support of short sequences (viruses or plasmids) - often missed by prev versions
* New mode for ONT Guppy5+ and Q20
* Polishing and structural accuracy improvements
* Optimized default parameters for HiFi and ONT datasetshttps://t.co/TnQ6rNmu1M
Flye 2.8 is out! Now with longer and faster HiFi assemblies. We also switched to a different k-mer indexing strategy (originally used in --meta). As a result, genome size estimate is no longer required at input, and large assemblies take 1.5-2x less RAM https://t.co/TnQ6rNmu1M
— Mikhail Kolmogorov (@fenderglass) 2020年8月5日
Flye 2.7 is out! Many improvements for assembly of real (and complex) metagenomes + new mode to reveal alternative haplotypes on the graph. Here is a bacterium that lives in sheep gut and supposedly is represented in many closely-related strains/species. https://t.co/gNFND8RKIS pic.twitter.com/1Ms77qdYrl
— Mikhail Kolmogorov (@fenderglass) 2020年3月4日
Presentation PDF (direct link)
インストール
mac os10.13のminiconda2-4.0.5環境でテストした。
Flye is available for Linux and MacOS platforms.
依存
- C++ compiler with C++11 support (GCC 4.8+ / Clang 3.3+ / Apple Clang 5.0+)
- GNU make
- Python 2.7
- Git
- Core OS development headers (zlib, etc)
Flye package includes some third-party software:
- libcuckoo
- intervaltree
- lemon
- minimap2
- Graphviz (optional)
本体 GIthub
#アセンブリグラフを可視化するならgraphvizをインストール
sudo apt install graphviz
or
mamba install -y -c bioconda graphviz
#本体のインストール(conda使用)
mamba install -c conda-forge -c bioconda -y flye
#レポジトリから
git clone https://github.com/fenderglass/Flye
cd Flye
python setup.py build
> flye -h
4$ flye -h
usage: flye (--pacbio-raw | --pacbio-corr | --pacbio-hifi | --nano-raw |
--nano-corr | --subassemblies) file1 [file_2 ...]
--genome-size SIZE --out-dir PATH
[--threads int] [--iterations int] [--min-overlap int]
[--meta] [--plasmids] [--trestle] [--polish-target]
[--keep-haplotypes] [--debug] [--version] [--help]
[--resume] [--resume-from] [--stop-after]
Assembly of long reads with repeat graphs
optional arguments:
-h, --help show this help message and exit
--pacbio-raw path [path ...]
PacBio raw reads
--pacbio-corr path [path ...]
PacBio corrected reads
--pacbio-hifi path [path ...]
PacBio HiFi reads
--nano-raw path [path ...]
ONT raw reads
--nano-corr path [path ...]
ONT corrected reads
--subassemblies path [path ...]
high-quality contigs input
-g size, --genome-size size
estimated genome size (for example, 5m or 2.6g)
-o path, --out-dir path
Output directory
-t int, --threads int
number of parallel threads [1]
-i int, --iterations int
number of polishing iterations [1]
-m int, --min-overlap int
minimum overlap between reads [auto]
--asm-coverage int reduced coverage for initial disjointig assembly [not
set]
--plasmids rescue short unassembled plasmids
--meta metagenome / uneven coverage mode
--keep-haplotypes do not collapse alternative haplotypes
--trestle enable Trestle [disabled]
--polish-target path run polisher on the target sequence
--resume resume from the last completed stage
--resume-from stage_name
resume from a custom stage
--stop-after stage_name
stop after the specified stage completed
--debug enable debug output
-v, --version show program's version number and exit
Input reads can be in FASTA or FASTQ format, uncompressed
or compressed with gz. Currently, PacBio (raw, corrected, HiFi)
and ONT reads (raw, corrected) are supported. Expected error rates are
<30% for raw, <3% for corrected, and <1% for HiFi. Note that Flye
was primarily developed to run on raw reads. Additionally, the
--subassemblies option performs a consensus assembly of multiple
sets of high-quality contigs. You may specify multiple
files with reads (separated by spaces). Mixing different read
types is not yet supported. The --meta option enables the mode
for metagenome/uneven coverage assembly.
You must provide an estimate of the genome size as input,
which is used for solid k-mers selection. Standard size
modifiers are supported (e.g. 5m or 2.6g). In the case
of metagenome assembly, the expected total assembly size
should be provided.
To reduce memory consumption for large genome assemblies,
you can use a subset of the longest reads for initial disjointig
assembly by specifying --asm-coverage option. Typically,
40x coverage is enough to produce good disjointigs.
You can run Flye polisher as a standalone tool using
--polish-target option.
#version2.9 help (v2.9-b1768)
# ./flye -h
usage: flye (--pacbio-raw | --pacbio-corr | --pacbio-hifi | --nano-raw |
--nano-corr | --nano-hq ) file1 [file_2 ...]
--out-dir PATH
[--genome-size SIZE] [--threads int] [--iterations int]
[--meta] [--polish-target] [--min-overlap SIZE]
[--keep-haplotypes] [--debug] [--version] [--help]
[--scaffold] [--resume] [--resume-from] [--stop-after]
[--read-error float] [--extra-params]
Assembly of long reads with repeat graphs
optional arguments:
-h, --help show this help message and exit
--pacbio-raw path [path ...]
PacBio regular CLR reads (<20% error)
--pacbio-corr path [path ...]
PacBio reads that were corrected with other methods
(<3% error)
--pacbio-hifi path [path ...]
PacBio HiFi reads (<1% error)
--nano-raw path [path ...]
ONT regular reads, pre-Guppy5 (<20% error)
--nano-corr path [path ...]
ONT reads that were corrected with other methods (<3%
error)
--nano-hq path [path ...]
ONT high-quality reads: Guppy5+ or Q20 (<5% error)
--subassemblies path [path ...]
[deprecated] high-quality contigs input
-g size, --genome-size size
estimated genome size (for example, 5m or 2.6g)
-o path, --out-dir path
Output directory
-t int, --threads int
number of parallel threads [1]
-i int, --iterations int
number of polishing iterations [1]
-m int, --min-overlap int
minimum overlap between reads [auto]
--asm-coverage int reduced coverage for initial disjointig assembly [not
set]
--hifi-error float [deprecated] same as --read-error
--read-error float adjust parameters for given read error rate (as
fraction e.g. 0.03)
--extra-params extra_params
extra configuration parameters list (comma-separated)
--plasmids unused (retained for backward compatibility)
--meta metagenome / uneven coverage mode
--keep-haplotypes do not collapse alternative haplotypes
--scaffold enable scaffolding using graph [disabled by default]
--trestle [deprecated] enable Trestle [disabled by default]
--polish-target path run polisher on the target sequence
--resume resume from the last completed stage
--resume-from stage_name
resume from a custom stage
--stop-after stage_name
stop after the specified stage completed
--debug enable debug output
-v, --version show program's version number and exit
Input reads can be in FASTA or FASTQ format, uncompressed
or compressed with gz. Currently, PacBio (CLR, HiFi, corrected)
and ONT reads (regular, HQ, corrected) are supported. Expected error rates are
<15% for PB CLR/regular ONT; <5% for ONT HQ, <3% for corrected, and <1% for HiFi. Note that Flye
was primarily developed to run on uncorrected reads. You may specify multiple
files with reads (separated by spaces). Mixing different read
types is not yet supported. The --meta option enables the mode
for metagenome/uneven coverage assembly.
To reduce memory consumption for large genome assemblies,
you can use a subset of the longest reads for initial disjointig
assembly by specifying --asm-coverage and --genome-size options. Typically,
40x coverage is enough to produce good disjointigs.
You can run Flye polisher as a standalone tool using
--polish-target option.
実行方法
* --genome-sizeは削除。v2.9では短い配列のアセンブリが改善されたため、plasmidオプションも廃止されている。
PacbioのCLR
1、テストデータのダウンロード
mkdir pacbio_ecoli_test && cd pacbio_ecoli_test/
wget https://zenodo.org/record/1172816/files/Loman_E.coli_MAP006-1_2D_50x.fasta
2、ラン
flye --pacbio-raw Loman_E.coli_MAP006-1_2D_50x.fasta --out-dir flye_output --threads 20
-
-t number of parallel threads [1]
-
-g estimated genome size (for example, 5m or 2.6g)
Pacbio Hifi (CCS)
flye --pacbio-hifi reads.fq.gz --out-dir flye_output --threads 20
Nanopore
flye --nano-raw ONT.fq.gz --out-dir flye_output --threads 20
#With scaffolding (v2.9よりScaffoldingは--scaffoldをつけてのオプション扱いになった. pacbioも同じ)
flye --nano-raw ONT.fq.gz --out-dir flye_output --threads 20 --scaffold
#エラーレート 3-5%の Q20 リード
flye --nano-hq ONT.fq.gz --out-dir flye_output --threads 20 --scaffold
Githubより
グラフの構造に基づいて、コンティグをさらにscaffoldsに並べることができる場合がある。--scaffoldをつけると、これらの順序付けされたコンティグは、scaffoldの一部としてアセンブリファイルに出力される(scaffold_という接頭辞付き)。assembly_info.txtファイルには、scaffoldがどのように形成されたかについての追加情報が含まれる。
Visualization
アセンブリグラフの可視化
dot -Tpng -O assembly_graph.gv
テストラン2
S.cerevisiaeのシミュレーションデータでもテストしてみる。
1、nanosim-h(紹介)を使い、R.9.4 2Dのデータを3万リード発生
nanosim-h -n 30000 -p ecoli_R9_2D Saccharomyces_cerevisiae.fa -o simulated
2、リード長チェック
seqkit stats simulated.fa
$ seqkit stats simulated.fa
file format type num_seqs sum_len min_len avg_len max_len
simulated.fa FASTA DNA 30,000 233,039,118 94 7,768 58,983
トータル2.3億bp (x20くらい)。
3、polishせず、Flyeでアセンブリ
flye --nano-raw simulated.fa --out-dir output --threads 8
macbook pro15インチ (2015)でテストすると、ランタイムは20分ほどだった。
出力

4、サイズチェック
seqkit stats output/scaffolds.fasta
$ seqkit stats output/scaffolds.fasta
file format type num_seqs sum_len min_len avg_len max_len
output/scaffolds.fasta FASTA DNA 52 12,311,247 3,809 236,754.8 885,399
56スキャホールドできている。
5、graphvizでグラフ可視化
dot -Tpng -O assembly_graph.gv

edgesが配列、ノード(頂点)は単なるジャンクション。全てのedgesはフォワード、リバースの両方が表現されている(+と-)。

ユニークなedgesは黒線だが、リピート配列のedgesは色線になる(repeatを完全にまたぐロングリードがなかったため、repeat resolutionできなかった配列)。隣接する場合、同じ色がアサインされる。
6、D-GENIES(紹介)を使ってdot plot解析

大きなミスアセンブリはない。
追記
2回polishing
flye --nano-raw nanopore.fq.gz --out-dir outdir --threads 40 -m 2
- -i number of polishing iterations [1]
trusted contigsを使う。
flye --nano-raw nanopore.fq.gz --out-dir outdir --genome-size 100M
--threads 40 --subassemblies contigs.fasta
flye --out-dir outdir --threads 40 --subassemblies contigs.fasta
- --subassemblies high-quality contigs input
結果が返って悪くなることもあるのでご注意ください。
追記2
途中から再開する。
flye --nano-raw ONT.fq.gz --out-dir flye_output --threads 20 --scaffold
- --resume resume from the last completed stage
- --resume-from stage_name resume from a custom stage
- --stop-after stage_name stop after the specified stage completed
追記3
v2.9からかは確認していませんが、アセンブリグラフ上では存在しても、depthが0か0に近いコンティグ(通常ジャンク)はassembly.fastaには書き出されないのを確認しました。そのようなコンティグを使いたい時はGFAを変換するなどの方法を取る必要があります。
追記4
Flyeでは部分的にcollapseしたハプロイドゲノムアセンブリが生成される。hapdupを使うことで倍数体resolivedのゲノムアセンブリを得ることができる。
引用
Assembly of Long Error-Prone Reads Using Repeat Graphs
Mikhail Kolmogorov, Jeffrey Yuan, Yu Lin, Pavel Pevzner
bioRxiv preprint Posted January 12, 2018.
doi: https://doi.org/10.1101/247148
Assembly of long error-prone reads using de Bruijn graphs
Lin Y, Yuan J, Kolmogorov M, Shen M, Chaisson M, Pevzner PA
Proc Natl Acad Sci U S A. 2016 Dec 27;113(52):E8396-E8405
追記
Assembly of long, error-prone reads using repeat graphs
Mikhail Kolmogorov, Jeffrey Yuan, Yu Lin, Pavel A. Pevzner
Nature Biotechnology, 01 April 2019
追記 2.4
Flye 2.4 with metagenome assembly mode is out! It is designed for non-uniform coverage distribution and scales to ultra-deep datasets. Find me on #PAGXXVII if you want to discuss in person (or if you are curious where the best tacos in San Diego are). https://t.co/gNFND8RKIS pic.twitter.com/u9tIAn9ImU
— Mikhail Kolmogorov (@fenderglass) January 15, 2019
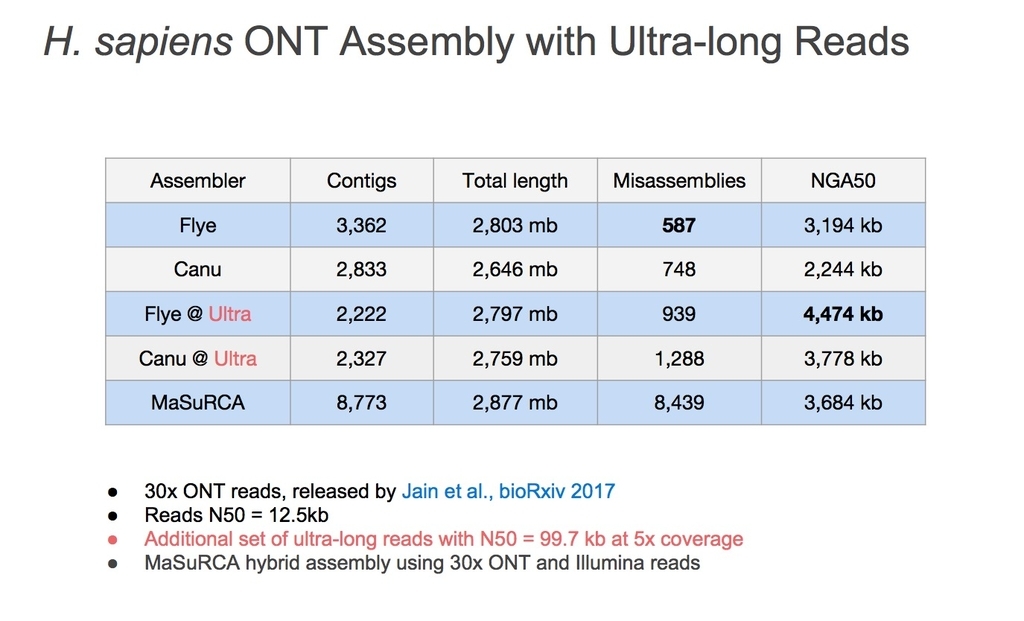
他のツールとの比較。
MaSuRCA4にも組み込まれるそうです。
関連
2019 9/7追加
勉強になります。
2021 2/19
Flye Genome Assembly--a How-To for all of us
2020 3/31
ゲノムサイズ350-Mbの生き物のディープなPromethIONシーケンシングデータ(raw 94GB)を使ってアセンブリタイムとピークメモリを調べてみた。ランタイムは64-hで、ピークメモリは191 GBだった(xeon platinum 8176 single, DDR4 2400MHz 64GBx8環境)。
> seqkit stats flye/assembly.fasta
file format type num_seqs sum_len min_len avg_len max_len
flye/assembly.fasta FASTA DNA 2,328 341,950,193 134 146,885.8 4,880,431