snp-distsは、Torsten Seemannさん (GIthub) がGithubで公開されている、配列間のSNP数を計算して、行列出力するツール。
If you also want a SNP distance matrix from the alignment, I wrote snp-dists to match: https://t.co/CAM04kDSBP
— Torsten Seemann (@torstenseemann) September 22, 2017
インストール
mac os10.14のminiconda3-4.3.30環境でテストした。
ビルド依存
- snp-dists is written in C to the C99 standard and only depends on zlib.
本体 Github
#Anaconda環境でcondaを使い導入
conda install -c bioconda -c conda-forge snp-dists
#またはhomebrewを使い導入
brew install brewsci/bio/snp-dists
> snp-dists -h
$ snp-dists -h
SYNOPSIS
Pairwise SNP distance matrix from a FASTA alignment
USAGE
snp-dists [options] alignment.fasta[.gz] > matrix.tsv
OPTIONS
-h Show this help
-v Print version and exit
-q Quiet mode; do not print progress information
-a Count all differences not just [AGTC]
-k Keep case, don't uppercase all letters
-c Output CSV instead of TSV
-b Blank top left corner cell instead of 'snp-dists 0.6'
URL
https://github.com/tseemann/snp-dists (Torsten Seemann)
——
テストラン
以下のような4つの配列のアライメントから、総当たりでペアワイズSNP distanceを計算し、matrixで出力する。
cat test/good.aln
$ cat test/good.aln
>seq1
AGTCAGTC
>seq2
AGGCAGTC
>seq3
AGTGAGTA
>seq4
TGTTAGAC
snp-distsを実行
snp-dists test/good.aln > distances.tab
出力のSNP distance matrixをexcelで開いてみる。
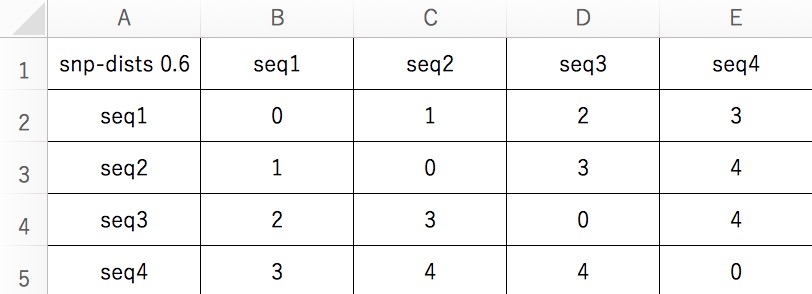
数値は塩基置換の数を表す。当然その配列自身で比較すると0になる。もっとも距離があるのはseq2とseq4、及びseq3とseq4。
本ツールはindelによるギャップ(-)は計算に入れません。
引用
https://github.com/tseemann/snp-dists