2018 11/12 リンクエラー修正
2019 3/9 分かりにくい部分を修正
過去10年にわたるショートリードシーケンシング、ロングリードシーケンシングおよびアセンブリの進歩(Salzberg et al、2011; Chin et al、2013; Hackl et al、2014)は、全ゲノムシーケンシングの扉を様々な分野の生物学者に開いた。公開されている配列データベースには既に数千ものドラフトゲノムと完全なゲノムが含まれており(Benson et al、2013)、さらに多くのものが進行中である(Pagani et al、2012)。特に、近年のアウトブレイク(Rasko et al、2011)に関連する病原菌株のハイスループットシークエンシングプロジェクト、およびメタゲノムおよびシングルセルシーケンシングアプローチを用いた微生物群集やパンゲノムを標的とする大規模生態学的研究がこのプロセスに寄与している(Turnbaugh et al、2007; Kashtan et al。、2014)。これらの豊富なデータセットは大規模な進化プロセスのために探索することができ、それにはゲノム組換え(Didelot、Méric&Falush、2012; Namouchi et al、2012; Yahara et al、2014)、アイランド伝達および水平遺伝子伝達(Fischer、2015; Touchon&Rocha、2007)、また、移動性または内在性のウイルスエレメントのしばしば関連するダイナミクス(Avrani et al、2011; Coleman et al、2006; Langille、Hsiao&Brinkman)が含まれる 。全ゲノム比較の他の応用には、paleopolyploidization events(Vanneste et al、2014)および腫瘍の異種性の定量的測定(Schwarz et al、2015)が含まれる。
しかし、得られた全ゲノム比較の適切な解釈を容易にするために、視覚化が重要である。アライメントしたゲノムのインタラクティブなグラフィカル表現を提供するための最初のツールの1つはMauve(Darling et al、2004)であった。 Mauveは、色と結合線で示される同種のシンテニーブロックを持つ同一直線上のレイアウトでゲノムを表す(Mauve紹介)。対話型スタンドアローンビューアーACT(Carver et al、2008)は、アライメントブロックに加えて、遺伝子などのゲノムアノテーション表現をサポートしている(ACT 紹介)。 RライブラリgenoPlotR(Guy、Kultima&Andersson、2010)とPythonベースのアプリケーションEasyFig(Sullivan、Petty&Beatson、2011)はいずれも共線形レイアウトとと機能 annotationsをサポートしているが、静止画を生成するように設計されており、インタラクティブな分析機能を欠いている。
線形レイアウトに加え、ゲノムの環状表現を行うツールが開発されている。BLASTatlas(Hallin、Binnewies&Ussery、2008)およびBRIG(Alikhan et al、2011)は、個々のゲノムのデータを表すために複数の同心円リングを使用し、BRIGはインタラクティブなグラフィカルインターフェースも提供する(BRIG紹介)。 GenomeRing(Herbig et al、2012)も同様に環状表現を用いるが、すべてのゲノムを同じリング上に置き、シンテニーブロックはリングの中心に伸びるアークで接続する。
(一部略)
既存のソフトウェアは、インタラクティブなツールはデータの探索には役立つが、図のエクスポートや品質の高いサポートは限られている。スクリプトベースのツールは、より高度なカスタマイズと図生成品質を提供するが、それぞれの言語に精通している必要があり、しばしば生成に時間がかかる。 SybilなどのWebベースおよびデータベースベースのスイートでは、アップロードとインポートの手順が利用を複雑にし、適用性を制限している。
ここでは、複数の全ゲノムアライメントのインタラクティブな視覚化のために設計されたスタンドアローンのアプリケーションAliTV(Alignment Toolbox and visualization)を紹介する。 AliTVは新しい全ゲノムアライメントを直接読んだり、自動的に生成したり、結果をすばやく探索したり、視覚化を操作したりカスタマイズしたりすることを目指している。 AliTVは、共通フォーマット(FASTA、GenBank、GFF、MAF、Newickなど)で配列とアノテーションまたはアライメントデータを読み取り、内部的にlastzを使用してアラインメントを計算する(Harris、2007)。ユーザーフレンドリーなインターフェイスは、最先端のD3.js JavaScriptフレームワーク上に構築され、一般的なWebブラウザでプラットフォームに依存しない方法で利用できる。ゲノムは、アノテーションと任意の系統樹を含む、高度にカスタマイズ可能な共直線レイアウトで表される。ツリーはAliTVによって計算されるのではなく、データ生成時に提供される必要がある。また、ゲノムの順序は、リアレンジメントを最小限に抑えるために自動的には最適化されない。カスタマイズは、ユーザーによる図の保存、再ロード、および高品質のSVGファイルへのエクスポートに対応している。
HP
https://alitvteam.github.io/AliTV/
Documentation
https://alitv.readthedocs.io/en/latest/index.html
AliTVに関するツイート
インストール
ubuntu16.04でテストした(docker使用。ホストOS: mac os10.12)。
依存
- perl
- cpanm
- lastz
#cpanmがないなら入れておく。
cd /usr/local/bin/
curl -LOk http://xrl.us/cpanm
chmod +x cpanm
#lastz
git clone https://github.com/lastz/lastz.git
cd lastz/
make
sudo make install
本体 GIthub
AliTV-Perl interface
git clone --recursive https://github.com/AliTVTeam/AliTV
cd AliTV/AliTV-perl-interface/
cpanm --installdeps .
# perl bin/alitv.pl -h
Usage:
# complex configuration via yml file
alitv.pl [OPTIONS] options.yml
# OR
# easy alternative including the generation of a yml file
alitv.pl [OPTIONS] *.fasta
Options:
--project Project name
The name of the project will be the given argument. If this
parameter was not provided, one project name will be auto generated.
This will be the base name for the log file, the yml file, and the
output file. If a YML file is provided, this value will be
overwritten by the basename of the YML file.
--output Output file
The name of the output file. If non is provided, the output file
name will be based on the project name. If STDOUT should be used,
please set the output filename to "-" via option "alitv.pl --output
-".
--logfile Log file
The name of the log file. If non is provided, the log file name will
be based on the project name.
--overwrite or --force Overwrite existing project.yml or output.json
files
Default behaviour is to keep existing project yml and json files. If
"--overwrite" or "--force" is specified, the files will be
overwritten. Overwriting can be expicitly disabled by
"--no-overwrite" or "--no-force" parameter.
すぐ使えるようにdockerイメージも上げておきます。
docker pull kazumax/alitv
#カレントと/dataを共有して立ち上げる
docker run --rm -itv $PWD:/data kazumax/alitv
cd /AliTV/AliTV-perl-interface/
#test run
perl bin/alitv.pl --project simple data/chloroset/*.fasta
#カレントにsimple~が出力されるので、これを下のdemoサイトでimportする。
テストラン
/data/chloroset/のfastaファイルを分析する。

cd /AliTV/AliTV-perl-interface/
perl bin/alitv.pl --project simple data/chloroset/*.fasta
fasta以外に、genbankファイルを使用できる(*1)。
デモサイトを使い可視化する。
https://alitvteam.github.io/AliTV/d3/AliTV.html
上のimportからjsonファイルを読み込む。
右端のimport => load JSONを選択、jsonファイルを読み込む。
結果が可視化された。
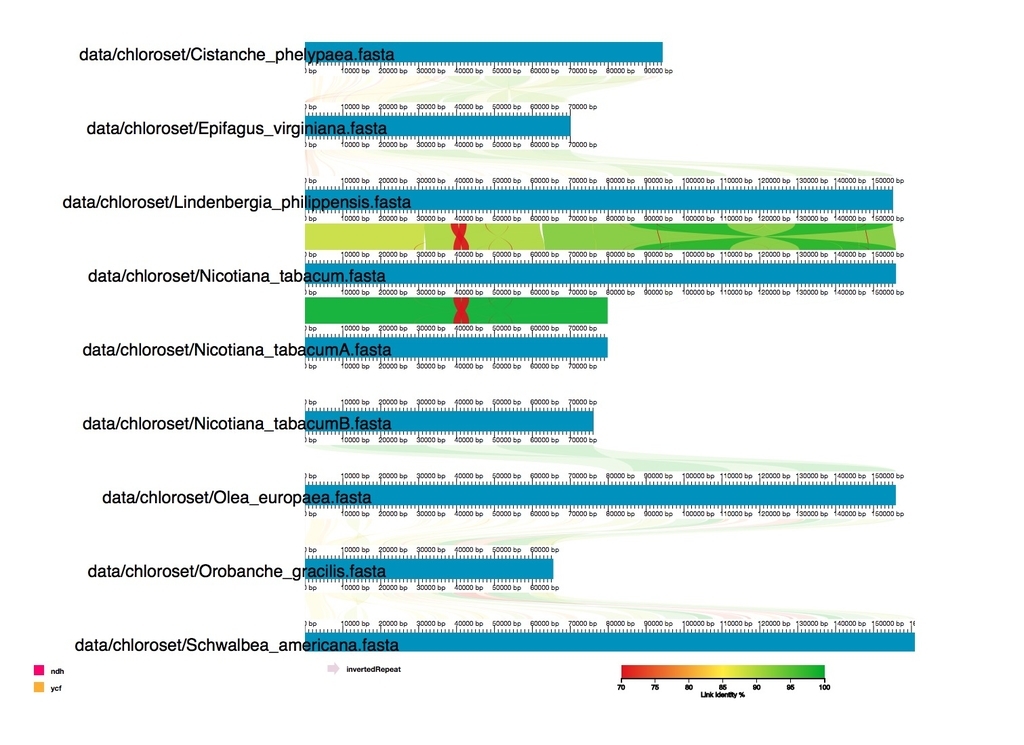 アライメント結果がカラーのリンクで可視化された。色は、右下のカラーグラデーションに従い、2配列間の高さを表している。上の図では、カーソルを配列の上に合わせて、その配列のアライメントだけハイライト表示している。
アライメント結果がカラーのリンクで可視化された。色は、右下のカラーグラデーションに従い、2配列間の高さを表している。上の図では、カーソルを配列の上に合わせて、その配列のアライメントだけハイライト表示している。
相同性の下限値を変更して、相同性が高い領域のみリンクを表示することもできる。上のメニューからFilterlingを選択。下限値を92%まで上げた。

赤~黄色~薄緑のリンクが表示されなくなった。
図はインタラクティブに操作可能。対象の配列の上で右クリック、change orientationを選択。

指定した配列の向きが逆になった。

move upとmove downボタンで上下の並びを順番を入れ替えられる。

自動ソートにも対応している。上のメニューのOrderからauto-orderを選択。

上に記載されているルールに従いソートされた。
結果はSVGで出力できる。

その他、ドラッグして拡大したり、カスタマイズした状態のjsonをexportすることもできます。著者らが準備した動画をみてください。
引用
AliTV—interactive visualization of whole genome comparisons
Markus J. Ankenbrand, Sonja Hohlfeld, Thomas Hackl, Frank Förster
June 12, 2017
*1
annotation trackがあれば遺伝子も表示できる。
上のメニュー → show hide annotations → geneの表示にチェックをつけてapply
関連