Antimicrobial resistance(AMR)(薬剤耐性。抗生物質耐性(AR or ABR)はAMRのサブクラス)は、ヒトの健康に対する主要な脅威の1つとなっており、世界中で年間700,000人の直接的な死因と推定されている[論文より ref.1]。この脅威に対処しなければ、この数字は2050年には1000万人に増加すると推定されている[ref.1]。薬剤耐性(AMR)に取り組むための戦略の重要な要素は、耐性のマーカーを同定するための迅速かつ正確な方法を有することである。 AMRとの闘いにおいて重要なツールの1つとなるように設定されたパーソナライズド・メディスンでのゲノムシーケンス解析使用によるゲノム配列データの利用可能性が増えたことによって、AMRのメカニズムおよび多様性に関する我々の理解が向上している。しかし、現在広く利用されているシーケンシング技術によって生成されたデータからAMR決定因子を直接同定することができるバイオインフォマティクスツールはほとんどない。現在利用可能な方法は、検出可能なAMRメカニズムのタイプに制限されているか、高スループット環境にスケーラブルできるものではない。
既存のツールの限界は、ハイスループットではないWebサービスを介してのみ利用可能であること、全ての微生物種についてのAMRの現在の知識を網羅的に表さないかもしれない特定のリファレンス配列に限定されている;入力としてアセンブリされたゲノム配列を必要とする。一塩基多型(SNP)に基づくAMR determinants を同定し解釈することができない;高い計算資源要件を有する。ほとんどのツールは、シーケンシングリードを一連のリファレンス遺伝子に合わせるものと、de novo アセンブリされた配列とリファレンス遺伝子の一致を検索するものの2つのカテゴリいずれかに分類される。広く使用されているSRST2 [ref.2]は、試料中の遺伝子の存在を予測するために、一連のリファレンス配列にリードをアライメントさせることに基づく方法の一例である。 KmerResistance [ref.3]も同様の手法を採用しているが、遺伝子の存在を特定するためにk-merマッチングを使用している(紹介)。 SRST2およびKmerResistanceは、カスタムリファレンス遺伝子セットとともに使用することができるが、耐性を付与するSNPなどの変異を直接同定または解釈することはできないため、遺伝子の存在によって耐性を同定することになる。 Mykrobe predictor [ref.4]は、k-mersでリファレンスグラフと照合する非常に高速なツールであり、変異型を特定することはできるが、そのデータを調べるためのAMR determinants のデータベースを作成する必要があり、現在は黄色ブドウ球菌および結核菌に限られている。
他の大部分のAMR検出ツールは、シーケンシングリードから生成するのに計算的に高価であるアセンブリされた配列を必要とし、完全なゲノムをデノボで組み立てる複雑さに起因するアセンブリエラーまたは失敗により、AMR determinants が見逃される可能性がある。これらの理由から、アライメントベースのアプローチは、以前にde novo assemblyの方法よりも優れていることが示されている[ref.2,3]。アセンブリされた配列を入力として使用するツールには、ResFinder [ref.5](v3紹介)、ARG-ANNOT [ref.6]、SSTAR [ref.7](紹介)、RAST [ref.8]などがある。これらの方法は、AMR遺伝子を同定するために、通常はblast [9]アルゴリズムを用いて、アセンブリされた配列をリファレンス遺伝子と一致させる。
ここでは、ARIBA(Assembly by Antimicrobial Resistance Identification by Assemblies)と呼ばれる新しいツールを紹介する。このツールは、マッピング/アラインメントとターゲットローカルアセンブリアプローチを組み合わせて、ペアのシーケンシングリードからAMR遺伝子と変異を効率的かつ正確に同定する。ローカルアセンブリを使用することは、アライメントされたデータの解釈のあいまいさを伴わずに連続した遺伝子またはヌクレオチド配列を提供しながら、アセンブリプロセスの複雑さをかなり低減する。 ARIBAには、ARG-ANNOT [ref.6]、CARD [ref.10]、MEGARes [ref.11]、ResFinder [ref.5]など、多数の公開データベースのサポートが含まれている。これは、コードまたは非コード配列を区別し、サンプル中に存在する各配列の詳細を提供する。同定された遺伝子がサンプル中で完全、切断または断片化されているかどうかを検証し、フレームシフト、非同義置換またはナンセンス変異などSNPおよびindelの結果として起きる効果も報告する。結果の解釈を容易にするため、ARIBAには複数のサンプルの結果を要約する関数が含まれている。これらの要約は、PhandangoインタラクティブVisualizationツール[ref.12 link]と互換性がある。最小阻止濃度(MIC wiki)データがサンプルに利用可能である場合、ARIBAは、統計解析および遺伝子型に対するMICのプロットを可能にする。 AMRを越え、ARIBAはより一般的に関心のある入力シーケンスを見つけるために使用でき、PlasmidFinder [ref.13](紹介)とVFDB [ref.14]データベースと、PubMLST [ref.15]からのデータを使用したmulti-locus sequence typing(MLST 日本臨床微生物学会2007総説)のための機能をサポートしている。
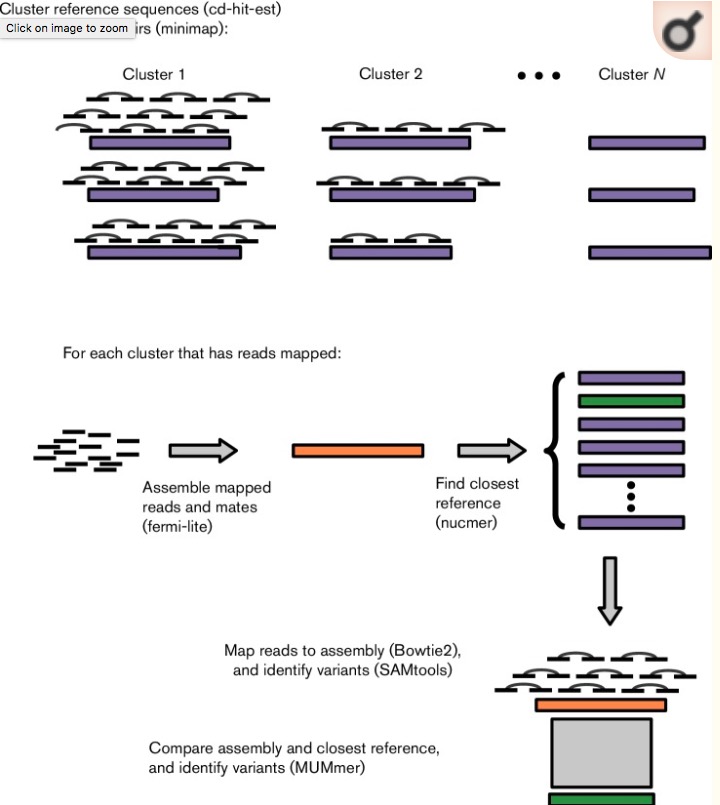
著者らはペアエンドシーケンスデータを入力としてpublicまたはカスタムデータベースからAMR determinantを識別するARIBAを開発した。簡単に言えば、AMRデータベース内のリファレンス配列は、CD-HIT [ref.16]を用いて類似性によってクラスタリングされる。リードは、各クラスタのリードセットを生成するために、minimap [ref.17]を使ってリファレンスシーケンスにマッピングされる。これらのリードは、そのクラスター内のシーケンスの少なくとも1つにマップされる。各クラスタのリードとそのシーケンスペアは、さまざまなパラメータの組み合わせでfermi-lite(Heng Li, github)を使用して独立してアセンブルされる。コンティグはMUMmerパッケージ[ref.18]のnucmerを使い最も似たリファレンスシーケンスが同定される。それからMUMmerのnucmerおよびshow-snpsプログラムを使いコンティグとリファレンス配列を比較して、配列間の完全性および任意の変異を同定する。クラスタのリードは、Bowtie2 [ref,19]を使用してアセンブリにマップされ、バリアントはSAMtools [ref.20]で呼び出される。最後に、AMRに重要であると予め定義された変異の存在または非存在を含むサンプルの同定されたすべての変異の詳細な報告がなされる。
ワークフロー。論文より転載。
ARIBAに関するツイート
インストール
依存
Python packages
- dendropy >= 4.2.0
- matplotlib (no minimum version required, but only tested on 2.0.0)
- pyfastaq >= 3.12.0
- pysam >= 0.9.1
- pymummer >= 0.10.1
本体 Github
Condaでインストールできる。またDockerイメージも提供されている。依存が多いので、Anaconda環境にてcondaで導入するか、他のツールとのバッティングを完全に避け仮想環境で使うのがラクと思われる。
#Anaconda環境ならcondaを使う(リンク)。
conda install -c bioconda ariba
#dockerイメージをpullする
docker pull sangerpathogens/ariba#Anacondaでないならpipで本体を導入し、依存も別に導入する。
pip install aliba
> docker run --rm -it -v /Users/user/docker_share/:/data sangerpathogens/ariba ariba -h
usage: ariba <command> <options>
ARIBA: Antibiotic Resistance Identification By Assembly
optional arguments:
-h, --help show this help message and exit
Available commands:
aln2meta Converts multi-aln fasta and SNPs to metadata
expandflag Expands flag column of report file
flag Translate the meaning of a flag
getref Download reference data
micplot Make violin/dot plots using MIC data
prepareref Prepare reference data for input to "run"
pubmlstget Download species from PubMLST and make db
pubmlstspecies
Get list of available species from PubMLST
refquery Get cluster or sequence info from prepareref output
run Run the local assembly pipeline
summary Summarise multiple reports made by "run"
test Run small built-in test dataset
version Get versions and exit
——
上記コマンドはホストの/Users/user/docker_shareと仮想環境のdata/を共有して立ち上げている。--rmで終了するとコンテナ破棄している(--rmをつけないと停止したコンテナが溜まってしまう)。
aliasつけたわけではないが、以後の説明でdocker run --rm -it -v /Users/user/docker_share:/data sangerpathogens/ariba aribaは省略してaribaとだけ表記する。
ラン
1、getref: Download reference data
例えばCARDデータベースをダウンロードする。
#usage: ariba getref [options] <db> <outprefix>
ariba getref card out.card
- <db> Database to download. Must be one of: argannot card megares plasmidfinder resfinder srst2_argannot vfdb_core vfdb_full virulencefinder
2、prepareref: Prepare reference data for input to "run"
ダウンロードしたリファレンス配列をデータベースに変換(準備)。
#usage: ariba prepareref [options] <outdir>
ariba prepareref -f out.card.fa -m out.card.tsv out.card.prepareref
- -f REQUIRED. Name of fasta file. Can be used more than once if your sequences are spread over more than on file
- -m Name of tsv file of metadata about the input sequences. Can be used more than once if your metadata is spread over more than one file. Incompatible with --all_coding.
out.card.preparerefディレクトリができる。
3、run: Run the local assembly pipeline
ローカルアセンブリ実行。
#SRAからNGSデータのダウンロード(*1)
fastq-dump --split-files SRR5132030 #ダウンロードとペアエンドfastq変換
#ローカルアセンブリ
#usage: ariba run [options] <prepareref_dir> <reads1.fq> <reads2.fq> <outdir>
ariba run out.card.prepareref pair1.fq pair2.fq output
- --threads Experimental. Number of threads. Will run clusters in
parallel, but not minimap (yet) [1] - --verbose Be verbose
- --min_scaff_depth Minimum number of read pairs needed as evidence for
scaffold link between two contigs [10] - --nucmer_min_id Minimum alignment identity for nucmer (delta-filter -i) [90]
- --nucmer_min_len Minimum alignment length (delta-filter -i) [20]
*1 ここではSRA explorerで"Salmonella Outbreak"で検索し、トップヒットしたデータを使った(https://www.ncbi.nlm.nih.gov/sra/?term=ERR1197641)
ジョブが終わるとoutputディレクトリができる。
output/の中身。
4、summary: Summarise multiple reports made by "run"
summary report作成。例えば3つのラン結果を統合する。
ariba summary out.summary output1/report1.tsv output2/report2.tsv output3/report3.tsv
out.summary.csvファイルが出力される。1データしか使っていないので、サンプルは1行のみ表示されている。

- pubmlstget: Download species from PubMLST and make db
Staphylococcus aureusのデータをPubMLST(リンク)からダウンロードし、データベースに変換(runに使用できる)。
ariba pubmlstget 'Staphylococcus aureus' out
- pubmlstspecies: Get list of available species from PubMLST
PubMLSTの利用可能な全データのダウンロード。
ariba pubmlstspecies
summary reportからMIC plotを出力するmicplotというコマンドもあります。
他にも作成したデータベースを検索する refquery 、調べたい遺伝子のマルチプルアライメント実行後のマルチFASTAとSNPS情報ファイル(TSSV形式)から、metadataに変換するaln2metaコマンドがある。aln2meta実行後は上記と同じようにpreparerefコマンドでデータベース化して、AMRの検索に利用することができる。
MLST callingのワークフローはチュートリアルを確認してください。
https://github.com/sanger-pathogens/ariba/wiki/MLST-calling-with-ARIBA
引用
ARIBA: rapid antimicrobial resistance genotyping directly from sequencing reads
Martin Hunt, Alison E Mather, Leonor Sánchez-Busó, Andrew J Page, Julian Parkhill, Jacqueline A Keane, Simon R Harris
Microb Genom. 2017 Sep 4;3(10):e000131.