ハイスループットシークエンシングは広範な技術となり、進化的研究を含む様々な研究分野でアクセス可能となっている。ゲノムが利用できない生物の転写産物をシーケンスし、注釈を付ける能力は、分子進化の分野における生物学者、特に非モデル生物を含むルーチンの仕事となっている。 RNA-Seqを介した非モデル生物の集団ゲノム解析の詳細なウォークスルーパイプラインは、De Wit [論文より ref.1 CrossRef]によって記述されている。このプロセスの第1ステップは、多数の配列のde novoアセンブリによる一連の転写セットの構築である。これは、Trinity [ref.2]、Trans-ABySS [ref.3]、Velvet-Oasis [ref.4]、MIRA [ref.5]、Newbler for 454[ref.6]などのさまざまなツールを使用して行うことができる。 Blast2Go [ref.7]、Trinotate [ref.8](紹介)、T-Ace [ref.9 pubmed]、annot8r [ref,10 pubmed]、FastAnnotator [ref.11 pubmed]などのようないくつかのソフトウェアスイートを使用して実行することができる。これらのツールは相同性検索のためのよく参照されてきた方法を使い、、既知配列データベースに対する相同性検索、タンパク質ドメイン同定、GO termおよびpathway解析などを行うが、全てBLASTライクな分析に大きく依存している。
BLAST(Basic Local Alignment Search Tool)[ref.12]は、未知ゲノム生物のトランスクリプトームシーケンシングからアセンブリされたESTのアノテーションを含む、配列間の局所的な相同性を示す領域を見出すため最も広く使用されるプログラムセットである。BLASTの結果はクエリ配列と個々のマッチ(被験体)の各々との間の類似性のレベルを示しているが、全トランスクリプトーム結果の洞察を達成する能力は乏しい。典型的には、種間の進化的保存の程度を決定し、研究対象の種において利用可能な経路の景観を推定し、あるいは種間発現パターンを比較することができる。
ESTデータベースの2つ以上のセット間のdifferential expressionプロファイルの確立は、アセンブリソフトウエアおよびDESeq [ref.13]およびedgeR [ref.14]のようなデジタル遺伝子発現統計ツールからなるパイプラインを用いて達成することができる。 TrinityとedgeRツールを使用するプロトコルは、Trinityウェブサイト[ref.2 CrossRef]に記載されている。これは、前提としてコンセンサストランスクリプトームを作成する必要がある。 T-Aceはトランスクリプトーム間の統計的な比較を提供する。しかしながら、いくつかの種からのraw発現データを統合してアセンブルされたコンセンサスのトランスクリプトームに依存する。
発現解析だけでなく、アセンブリソフトウェアやアノテーションソフトウェアの開発にも大きな努力が注がれているが、トランスクリプトーム全体の比較などの下流の調査ははるかに利用できない。ここでは、高速並列化BLASTフィルタリング・ユーティリティと全トランスクリプトのアラインメント比較を組み合わせた、ユーザーフレンドリーな統合ソフトウェア、VennBLASTを紹介する。 VennBLASTは直感的なベン図で結果を示し、BLAST比較での数関係を提供する。 VennBLASTは、全トランスクリプトームの進化的関連性の鳥瞰図を提供するが、遺伝子を有意義なサブグループに解剖し、様々なツールを使用してさらに解析することも可能である。例えばGene set enrichment analysisは、それらの背後にある生物学的意味を理解するために、VennBLAST選択遺伝子リストで実施することができる。これらのタスクを実行することは、バイオインフォマティクスにとっては直接的なことかもしれないが、VennBLASTの対象である非専門家にとっては時間がかかるものである。
VennBLASTは、対話型ユーザーインターフェイスを備えたデスクトップアプリケーションとして実行するよう実装されている。これはC#プログラミング言語で実装されており、ベン図を描画するために "Venn diagram plotter"コード[ref.15]を使用している。
VennBLASTはhttp://www.ariel.ac.il/research/fbl/softwareで非営利目的で自由に利用できる。
ダウンロード
mac os10.13のparallels 12-windows 10のエミュレーション環境でテストした。
公式ページ(プログラムとword形式のマニュアル)
ラン
下の図のようなワークフローで進める。
図1。論文より転載。
比較したい配列のblast解析を行う。マニュアルの通り、アミノ酸配列データベースに対してblastx解析する。例えば3つのデータベースA、B、 Cに対してコンティグ配列contigg.faをクエリとしてblastx解析を行う。
#Aのindexの作成
makeblastdb -in inputA.aa -dbtype prot
#Aのblastx
blastx -query contig.fa -db inputA.aa -out out.blastxA -outfmt "6 std qcovs" -max_target_seqs 1 -best_hit_overhang 0.1 -best_hit_score_edge 0.1 -num_threads 20
#Bのindexの作成
makeblastdb -in inputB.aa -dbtype prot
#Bのblastx
blastx -query contig.fa -db inputB.aa -out out.blastxB -outfmt "6 std qcovs" -max_target_seqs 1 -best_hit_overhang 0.1 -best_hit_score_edge 0.1 -num_threads 20
#Cのindexの作成
makeblastdb -in inputA.aa -dbtype prot
#Cのblastx
blastx -query contig.fa -db inputC.aa -out out.blastxC -outfmt "6 std qcovs" -max_target_seqs 1 -best_hit_overhang 0.1 -best_hit_score_edge 0.1 -num_threads 20
- -outfmt "6 std qcovs" getting the output in a tabular format which includes the default (standard)output parameters as well as the query coverage.
- -max_target_seqs control the number of matches recorded in the alignment (1 in this case)
- best_hit_overhang and best_hit_score_edge are used here in order to search for only the best matches for each query region reporting matches, and to avoid short hits
論文では2つの例で説明している。1つはone to manyの例で、もう1つはmany to oneの例である。one to manyの例では、非モデル生物Stylophora pistillata(ショウガサンゴ)のトランスクプトームデータからアセンブリして得たcontig配列をクエリとして、イソギンチャク目、イシサンゴ目、Hydrozoa目?の3つの目の様々なゲノムに対して、blastx解析をしている。それから、VennBLASTを使って同じ目の結果をマージしてe-valueでフィルタリングし、そのフィルタリング結果を比較してどの目に近いか等を議論している(論文を参照)。
dllとexeファイルが入っている。exeファイルをたたき起動する。

Uploadボタンからblast結果をアップロードし、 必要であれば中央のFilterボタンをクリックしフィルタリングする。例えばE valueが1e-10より高ければ除く。

save outputでフィルタリング後のblast結果は出力できる。
右下のMergeボタンを押すと下の画面が出現する。3つの群のblast結果を入力する。
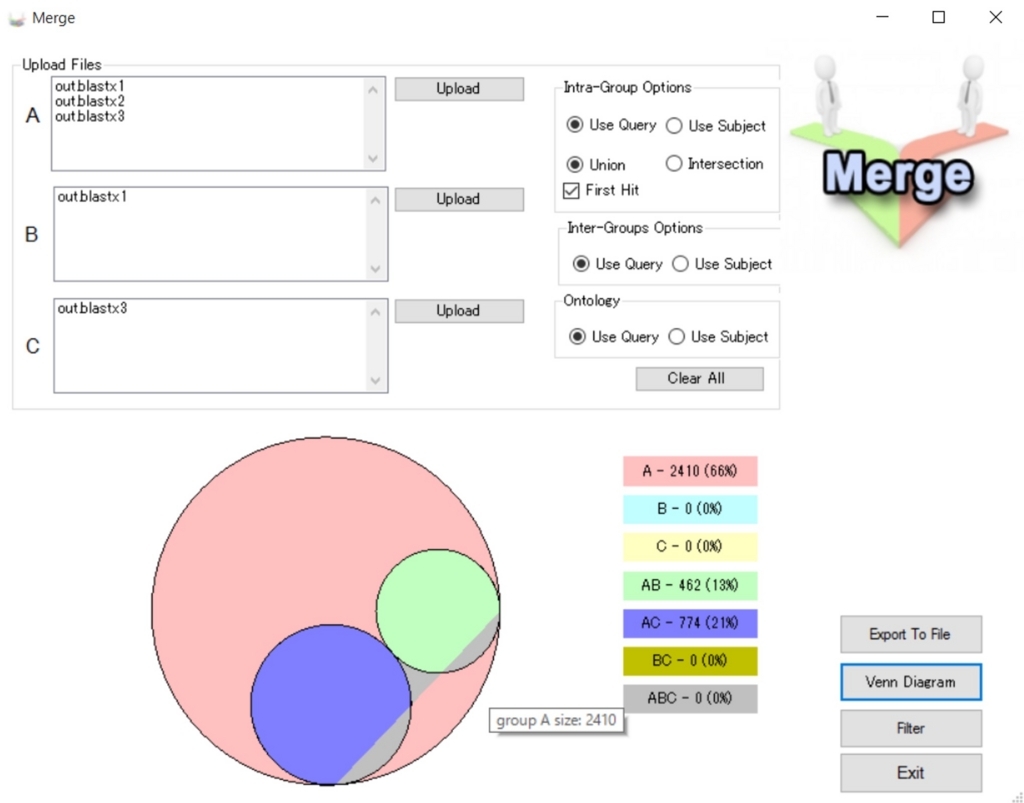
Venn Diagramボタンをクリックするとベン図が出力される(上は動作確認のテスト結果)。
引用
VennBLAST—whole transcriptome comparison and visualization tool
Zahavi T, Stelzer G, Strauss L, Salmon AY, Salmon-Divon M.
Genomics. 2015 Mar;105(3):131-6.
