保存されたシンテニーは、共通のゲノムを共有することによって支持される遺伝子間の推測された相同性関係を指し、生物のすべての領域にわたって広く使用される測定法である(論文より Moreno-Hagelsieb et al, 2001; Engstrom et al, 2007; Heger Ponting 2007; Poyatos and Hurst 2007; Tang、Bowers, et al 2008)。保存されたシンテニーは、大きなセットの遺伝子またはゲノム特徴が近接して(synteny)、しばしば同じ順序および向き(colinearity)で保存されている場合に明らかである(Tang、Bowers、et al。種間で保存されたシンテニーは、マップベースのクローニング、予測される遺伝子モデルの検証(Law et al。2015)、保存された非コード配列の同定(Haudry et al。2013)を含むゲノム研究のための不可欠な基盤となる。種内の保存されたシンテニーは、古代のpolyploidy events または他のタイプの大規模ゲノム重複を同定する(Wolfe 2001)。
シンテニーは、遺伝子相同性を確認するための追加情報を提供し、配列類似性のみに基づく推論よりはるかに信頼性が高い。典型的なBLAST(Basic Local Alignment Search Tool)分析の結果は、遺伝子の欠損または転位の有無を容易には示さない。ポピュラーなreciprocal best hitのアプローチは、先祖のゲノムを考慮したり、遺伝子または遺伝子ファミリーの進化の歴史についての多くの洞察を提供したりしない。より一般的には、OrthoMCL(Li et al、2003)やINPARANOID(Ostlund et al、2010)のようなタンパク質クラスタリングアルゴリズムは、進化速度が一定である場合に単一コピー遺伝子ファミリーには成功するかもしれないが、特に種間のパラロガス遺伝子の相反的な喪失の場合に、偽陽性の代謝産物を産生することがある。進化的な時間に渡って遺伝子の動きを追跡する位置研究は、より遺伝子中心のシンテニーツールを必要とする(Woodhouse et al、2011)。
キュレートされたシンテニー遺伝子セットは、ゲノム規模のパターンおよび進化の傾向を導き出すための重要なツールであり、広く普及している(Woodhouse et al、2011; Baxter et al、2012; Schnable et al、2012)。残念なことに、強固で正確な合成データセットを構築するには、現在、少数の研究グループに限定された専門的な比較ゲノムスキルが必要であり、今までのシンテニー情報を使用する主要な方法は、専門のグループによって公開された、手動でキュレーションされたシンテニー遺伝子セットによるものである。手動でキュレーションされた遺伝子セットは、公開サイクルによるラグがあり、所定のシンテニー遺伝子セットが公開されるまでに、新種のゲノムアセンブリが利用可能になることが多く、ゲノムアセンブリ、アノテーション、既存の公開されたゲノムの遺伝子情報が更新されることがよくある。ゲノムのアセンブリはますますハイペースでリリースされており、個々の研究者が種間のシンテニック領域を迅速に同定することができるツールが必要とされている。
シンテニーデータのコミュニティ利用の大部分は、一般的に、いくつかのユースケースの1つに分類される:1)1つ以上の追加の種においてその標的遺伝子のシンテニックオルソログを迅速に探したい特定の種からの特定の遺伝子に関心を持つ研究者、2)関連する種の集団全体にわたる単一の遺伝子または遺伝子ファミリーの変化を追跡したい研究者。シンテニー遺伝子リストを公開する際に導入されるまでの遅延に加えて、ほとんどの公開リストは保存されたシンテオックオーソログに関する情報を提供するだけであり、シンテニックオーソログが見つからない遺伝子について予測されるロケーションの情報は提供しない。これは、上記の#2の有用性、すなわちシンテシー分析の重要な利点の1つである「真のネガティブ」を特定する能力を大幅に制限している(“gray genome”として知られる) (Freeling et al、2008)。多くの進化的研究では、特定の遺伝子が実際にゲノム領域に欠けているか転移しているか(トランスポゼーション)の知識が必要である。これらの2つのシナリオでは遺伝子発現パターンの潜在的な変化が異なるため、遺伝子除去と転位を区別することは重要である。(一部略)
シンテニー検出ツールの新規実装を提供するにあたり、これまでのいくつかの著者によって設計および実装されたいくつかのツールを含む、一般的なシンティニー検出アルゴリズムの概要を示す。一般に、シンテニー探索アルゴリズムは、位置の共直線性または位置密度に基づいているか、どのタイプの統計的特徴を探索しているか(GhiurcutaおよびMoret 2014)、および「シンテニックブロック」の定義に基づいてグループ化することができる。最近のシンセシス検索ソフトウェアのリストには、iAdHore(Proost et al、2012)、mGSV(Revanna et al、2012)、SyMap(Soderlund et al、2011)、SynMap(Lyonsら2008)、Orthocluster(Vergara and Chen 2010) 、Synorth(Dong et al、2009)、MCScan(Tang、Wang、et al、2008)、MCScanX(Wang et al)などがある。これらのシンセシス検索ソフトウェアは、実行時間、計算資源要件、およびタイプI(偽陽性)またはタイプII(偽陰性)エラーのいずれかを最小限に抑えるという目標の点で、著者が受け入れたトレードオフで大きく異なる。さらに、実用的な観点からは、ツールはインターフェースタイプ(すなわち、コマンドライン、ウェブベース)および与えられたツールがグラフィック出力を提供するための組み込み機能を提供するかどうかによって区別され、結果の視覚的校正を可能にする(論文の表1)。
これらのアルゴリズムを慎重に評価した結果、より一般的な用途ではまだ満たされていない基本的な課題が示された。まず第一に、データのキュレーションはしばしば重要な課題である(Lohr 2014)。これは、ユーザーがゲノムのアノテーションファイルを、さまざまなアルゴリズムで必要なファイル形式に変換することを要求する。多くのツールがコマンドラインから実行され、与えられたツールから最も正確な結果を得るにはさまざまな設定を試す必要があり、複数の出力データセットを評価およびランク付けする方法を開発する必要があるユーザーにはさらに課題がある。興味のある生物の数が増えるにつれて、計算時間の要件は2次的に大きくなることが多く、これはオフラインアルゴリズムの課題を提示する。
著者らの新しいオンライン方法であるSynFindは、いくつかの最近のシステムに採用されていない新しい特徴を持つ。 SynFindは、予測ゲノム中の遺伝子と標的ゲノムとの間の複数のシンテニー領域を、予測された位置にシンテニックオルソログまたはパラログが存在するかどうかに全く無関係に同定する。 SynFindは、異なった進化距離および異なるゲノムアセンブリ品質を有する分類群における異なる構造的ゲノム変化に対処するためにシンテニー領域の密度および共線スコアの両方のオプションを提供する。 SynFindは、古代のpolyploidy events と他のゲノムに固有の遺伝子を明らかにするために、シンテニー深度表と遺伝子存在不在表を生成する。最も重要なのは、CoGeとの統合により、CoGeのツールを使用して、ユーザーが新しいゲノムを追加し、非公開にして、SynFindを使用してリリースされたものと同じように迅速に比較できるようにすることである。最新のゲノムデータとの緊密な統合により、コンピューティングリソースへのアクセス、下流のビジュアライゼーションおよび分析ツールが容易になり、進化的ゲノミクスおよび機能的ゲノミクスを橋渡しする多次元ゲノムデータセットの探索を容易にする自由なパイプラインが構築される。
チュートリアルビデオ
http://www.youtube.com/watch?v=2Agczny9Nyc.
CoGe (SynFindを選択)
https://genomevolution.org/coge/
動画で解説されているので、ここでは簡単に説明する。
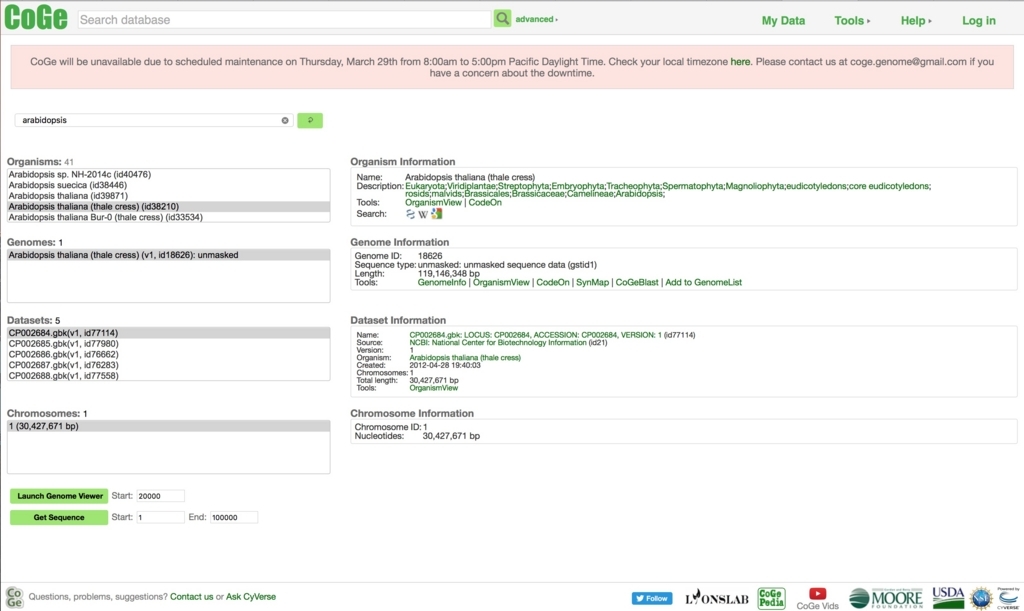
arabidopsisを検索。
Launch Genome Viewerを選択。
ビューアが立ちがる。右のメニューからgeneを追加。
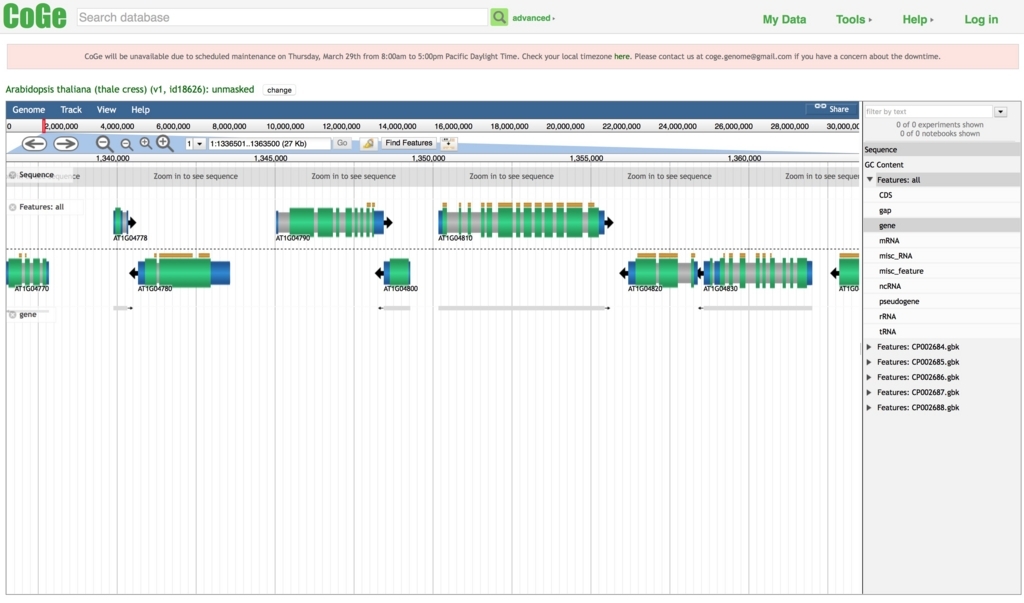
geneを選択してbox中のSynFindをクリック。
Oryza Sativaを選択。比較したいデータベースを1つまたは複数追加(データベースを選択してAddボタン)。

nitrateで検索。

興味あるアノテーションを選びページ下のRun SynFindを選択。
しばらく待つ。

対象遺伝子の近傍領域の比較結果が出る。
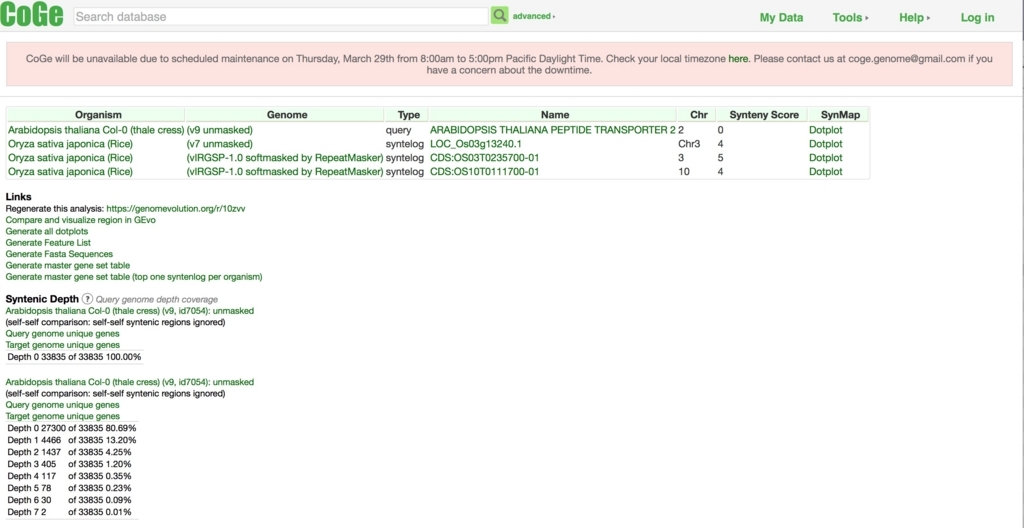
Dot plotを表示したり、リストをダウンロードできる。

引用
SynFind: Compiling Syntenic Regions across Any Set of Genomes on Demand.
Tang H, Bomhoff MD, Briones E, Zhang L, Schnable JC, Lyons E
Genome Biol Evol. 2015 Nov 11;7(12):3286-98.
Syntenic comparison of Arabidopsis thaliana and Arabidopsis lyrata