2018/12/21 anacondaとtwitterリンク追記 リンクミス修正
2019 3/6 minimap2に変更, 6/12 関連ツール追記, 6/13 関連ツール追記, 7/23 コードエラー修正、ショートリード使用例追記, 7/24 ループ用スクリプト追加、解析例追加、help更新, 7/29 追記
2022/04/25 GPUサポート(現在experimental)インストール追記
2022/06/05 構成変更
Pacific Biosciences(PacBio)およびOxford Nanopore Technologies(ONT)のロングリードシーケンシング技術の出現により、高いcontiguityを有するゲノムアセンブリを生産する能力は著しい進歩を遂げた。しかし、これらの技術の比較的高いエラー率(> 5%)に対処するために、アセンブル立パイプラインはリソース集約的なエラー訂正とコンセンサス生成(アセンブリ以後の)ステップに依存していた(Chin et al、 2013; Loman et al、2015)。 FALCON(Chin et al、2016)やCanu(Koren et al、2017)のような最近の方法では、この手法を改良してランタイムを大幅に改善しているが、大きなゲノムではまだ膨大な計算時間が要求されている(Sovićet et al)。最近、Li(2016)は、時間のかかるエラー訂正ステップを必要とせずにエラーのあるロングリードをアセンブルできることを示した。アセンブラminiasmは、他のlong-readアセンブラよりも1桁高速だが、他のメソッドの10倍以上のエラーを持つ可能性のあるシーケンスを生成する(Sovićet et al、2016a)。高速かつ正確なアセンブルは、構造変化検出、メタゲノム分類の改善、さらにはオンラインでの "read until"アセンブリ(Loose et al、2016)まで、幅広いアプリケーションを可能にする。そのため、迅速かつ正確なコンセンサスモジュールが不可欠である。これはLi(2016)によっても指摘されており、miniasmとminimapのスピードに合致するコンセンサスモジュールが開発された場合、高速なアセンブルが実現可能であることを強調している。
この論文では、Rapid ConsensusのためのRaconと呼ばれる非常に速いコンセンサスモジュールを提供することでこのニーズに取り組んでいる。Raconは、SIMDアクセラレーション、部分オーダーアライメントベースのスタンドアロンのコンセンサスモジュールで高品質のコンセンサスシーケンスを効率的に生成できる。 PacBioおよびOxford Nanoporeデータセットを用いたテストに基づいて、、Raconとmimiasmとを組み合わせることにより、最先端の方法と同等またはそれ以上の品質のコンセンサスゲノム生成を可能にする。
Raconはポリッシュツールではなく、コンセンサスコーラーであることが質問サイトで述べられている。
https://github.com/isovic/racon/issues/9
インストール
git clone --recursive https://github.com/isovic/racon.git racon
cd racon
mkdir build
cd build
cmake -DCMAKE_BUILD_TYPE=Release ..
make -j20
cd bin/
sudo make install
#bioconda(mac os, linux)
mamba install -c bioconda -y racon
#GPUサポート (CUDAが必要、認識してないならcmakeでエラーを起こす)
git clone --recursive https://github.com/lbcb-sci/racon.git racon
cd racon
mkdir build
cd build
cmake -DCMAKE_BUILD_TYPE=Release -Dracon_enable_cuda=ON ..
make -j20
> racon -h
# racon -h
usage: racon [options ...] <sequences> <overlaps> <target sequences>
<sequences>
input file in FASTA/FASTQ format (can be compressed with gzip)
containing sequences used for correction
<overlaps>
input file in MHAP/PAF/SAM format (can be compressed with gzip)
containing overlaps between sequences and target sequences
<target sequences>
input file in FASTA/FASTQ format (can be compressed with gzip)
containing sequences which will be corrected
options:
-u, --include-unpolished
output unpolished target sequences
-f, --fragment-correction
perform fragment correction instead of contig polishing
(overlaps file should contain dual/self overlaps!)
-w, --window-length <int>
default: 500
size of window on which POA is performed
-q, --quality-threshold <float>
default: 10.0
threshold for average base quality of windows used in POA
-e, --error-threshold <float>
default: 0.3
maximum allowed error rate used for filtering overlaps
-m, --match <int>
default: 5
score for matching bases
-x, --mismatch <int>
default: -4
score for mismatching bases
-g, --gap <int>
default: -8
gap penalty (must be negative)
-t, --threads <int>
default: 1
number of threads
--version
prints the version number
-h, --help
prints the usage
#GPU版は追加オプションがある
実行方法
RaconのランにはPAFファイル(Pairwise mApping Format) が必要で、これはminimap2で作ることができる(minimap紹介)。
1、PAFファイルの作成。リードの種類に合わせ、-x で一括指定パラメータをつける。
minimap2 -t 20 -x map-ont ONT_raw_assembly.fasta long_reads.fq > mapped.paf
- -t number of threads [3]
2、Raconでコンセンサス配列を出力。
racon -t 20 long_reads.fq mapped.paf ONT_raw_assembly.fasta > racon1.fasta
- --bq Threshold for the average base quality of the input reads. If a read has average BQ < specified, the read will be skipped. If value is < 0.0, filtering is disabled. [10.0]
- -e Maximum allowed error rate. Used for filtering faulty overlaps. [0.30]
- -t Number of threads to use. [4]
- --erc - Perform error-correction instead of contig consensus. The only difference is in the type of parallelization to achieve better performance. [false]
Alignment:
- -M Match score (positive value). [5]
- -X Mismatch penalty (negative value expected). [-4]
- -G Gap open penalty (negative value expected). [-8]
- -E Gap extend penalty (negative value expected). [-6]
- -v Verbose level. 0 off, 1 low, 2 medium, 3 high, 4 and 5 all levels, 6-9 debug. [5]
ショートリードを使う
https://github.com/isovic/racon/issues/75
リンク先のスクリプトを使うと、paired-end fastqをコンカテネートできます。単純に結合するとraconがエラーを吐きます。
illumina data to polish · Issue #75 · isovic/racon · GitHub
#raw fastqを指定
python script.py input_1.fastq input2.fastq > paired.fq
#mapping
minimap2 -t 20 -x sr raw_assembly.fa paired.fq > mapped.paf
追記
raconでpolishすると数Mbもトータルシーケンスサイズが減少することがあるが、これはaconでpolishできないlow qaulityの配列が除去されていることによる。回避するには -u, --include-unpolishedをつけてランする。
結果を比較しています。とても参考になります。
https://albertsenlab.org/what-is-a-good-genome-assembly/
Raconは繰り返し走らせるのが基本になっている(結果に変化がなくなるまで)。例えばアセンブラのUnicyclerがこの戦略を採用している(紹介)。
https://github.com/rrwick/Unicycler/issues/51
繰り返すには、サイクル1の出力を同じ流れで解析していけばよい。どのくらい精度がよくなるかは、論文の表1あたりを確認してください。Canuやfalconと比較されています(リンク)。精度はやはりカバレッジがディープなほうがよくなるようです。
簡単なループ用シェルスクリプト(raw_assemblyをONT long readで修正)
#settingのところのraw assembly.fastaやlong readのパスを追加して使って下さい。この状態だと10回ループします。必要に応じて-u、--bq -1もつけてください。
ONT raw fastq以外のリードを使う場合はminimap2のパラメータ-x <map-ont>を変えて下さい。
miniasmのraw assemblyをraconで11回処理してquastで結果を分析した。
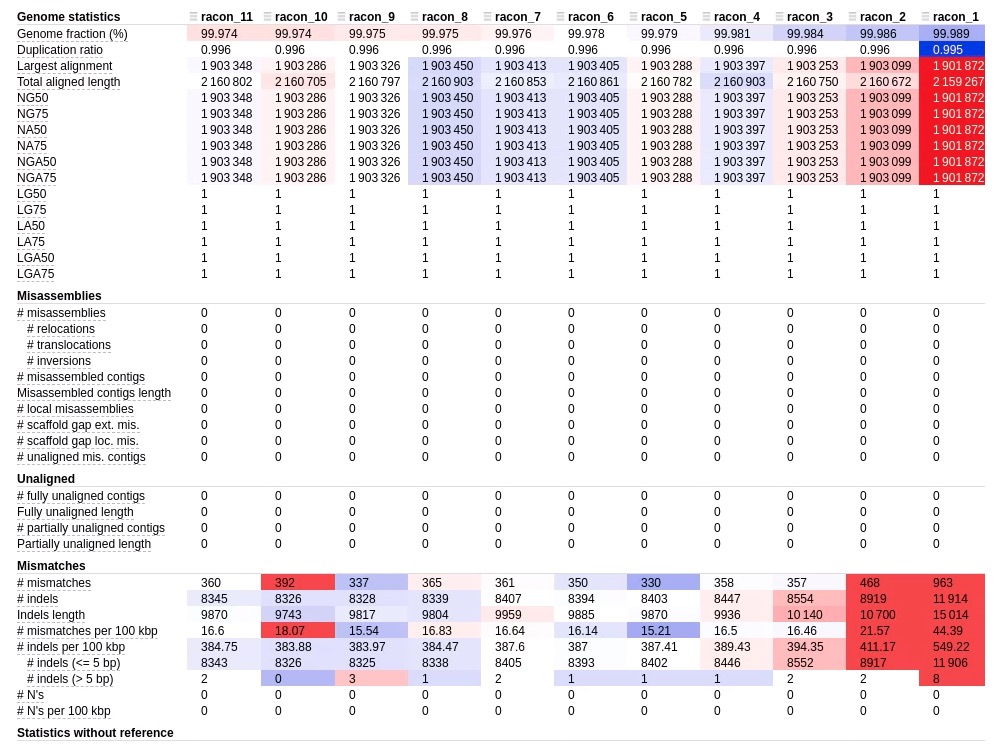
3回目以降はほとんど変化がない。また、ときおり悪化したりする。何度も実行する場合、結果を評価して意味のある処理か考えないといけない。結果を評価する時は、信頼できるリファレンスがあるならそれと比較する。信頼できるリファレンスが無いなら、BUSCOやcegmaなどを使ったり、配列間のdistanceを調べる方法を使う。
引用
Fast and accurate de novo genome assembly from long uncorrected reads.
Vaser R, Sović I, Nagarajan N, Šikić M
Genome Res. 2017 May;27(5):737-746.
Raconとnanopolishへの言及あり(査読中)
The long reads ahead: de novo genome assembly using the MinION
Carlos Victor de Lannoy, Dick de Ridder, Judith Risse
Published online 2017 Dec 12. doi: 10.12688/f1000research.12012.2
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5770995/
Question: How many iterations of Racon polishing should I expect???
https://github.com/rrwick/Unicycler/issues/51
関連